1.Путём поиска инвертированных повторов
Всё ещё работая с тРНК из прошлого практикума, на этот раз с помощью программы einverted была предсказана структура тРНК - этот способ отличается от предыдущего алгоритма find-pair, однако в идеальном варианте второй относительно предыдущего практикума способ должен дать схожие результаты.
Программа einverted принимает на вход fasta-файл последовательности тРНК(pdbid=1ml5) и анализирует его. Помимо исходного файла, есть возможность изменить параметры, с помощью которых проводится анализ, как то штраф за гэпы, или минимальное пороговое значение очков за совпадения. В принципе, программа способна работать и с заданными по-умолчанию параметрами, проблема лишь в том, что в случае 1ml5 она в таком случае не может найти вообще никаких компементарных участков.
Поэтому было решено изменить параметры, в частности, понизить пороговое значение (далее - mst) с 50 до, скажем, 20 и посмотреть,
какие результаты получатся в таком случае.
Мы стремимся получить около-достоверную картинку - 4 стебля = 4 двухцепочечных участка. Как выяснилось, именно mst вносит наибольший вклад в
выдачу программы. Попытавшись поэксперементировать с штрафами за гэп, было получено неадекватное восприятие со стороны einverted.
С тем же самым значением mst она выдавала достаточно похжие стебли, возможно, с большим, но сравнимым количеством
гэпов. Так что изменение штрафа не было внесено в конечный результат.
Исключительно эмпирическим путём было получено значение mst = 20, при котором программа отметила один из стеблей.
2.По алгоритму Зукера
Но прежде, чем переходить к сопоставлению, хотелось бы обратить внимание, что на руках у нас имелся ещё один метод анализа -
программа RNAfold. Это программа не из пакета EMBOSS, а из другого пакета, с которым мы ранее не сталкивались -
Viena Rna Package. Программа использует алгоритм Зукера, и на выходе мы так же имеем пары для стеблей.
Получилась весьма достоверная картинка:
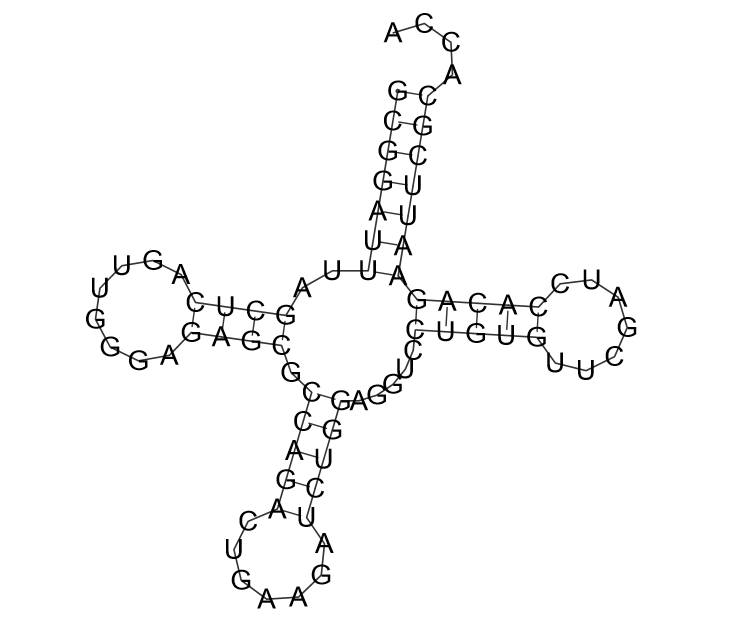
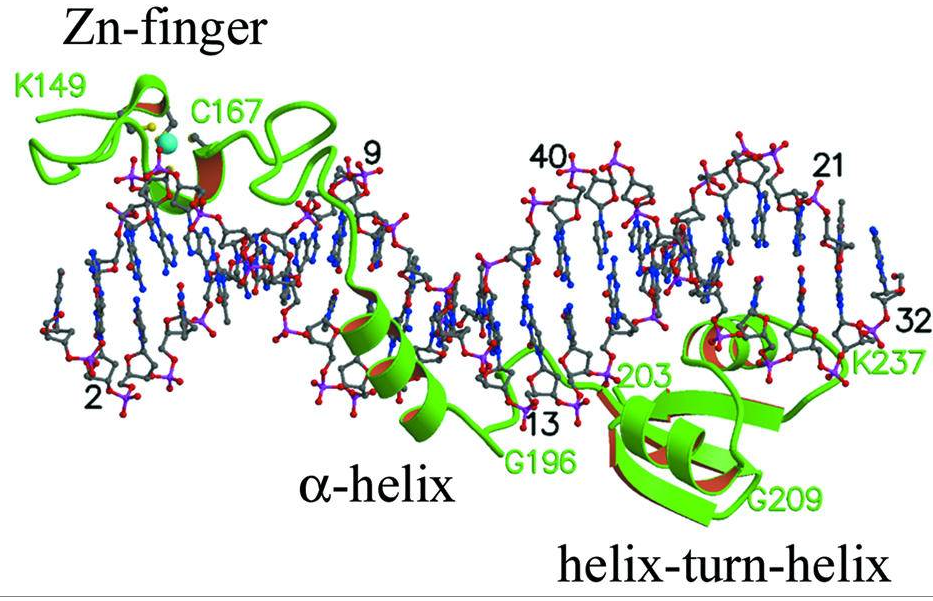
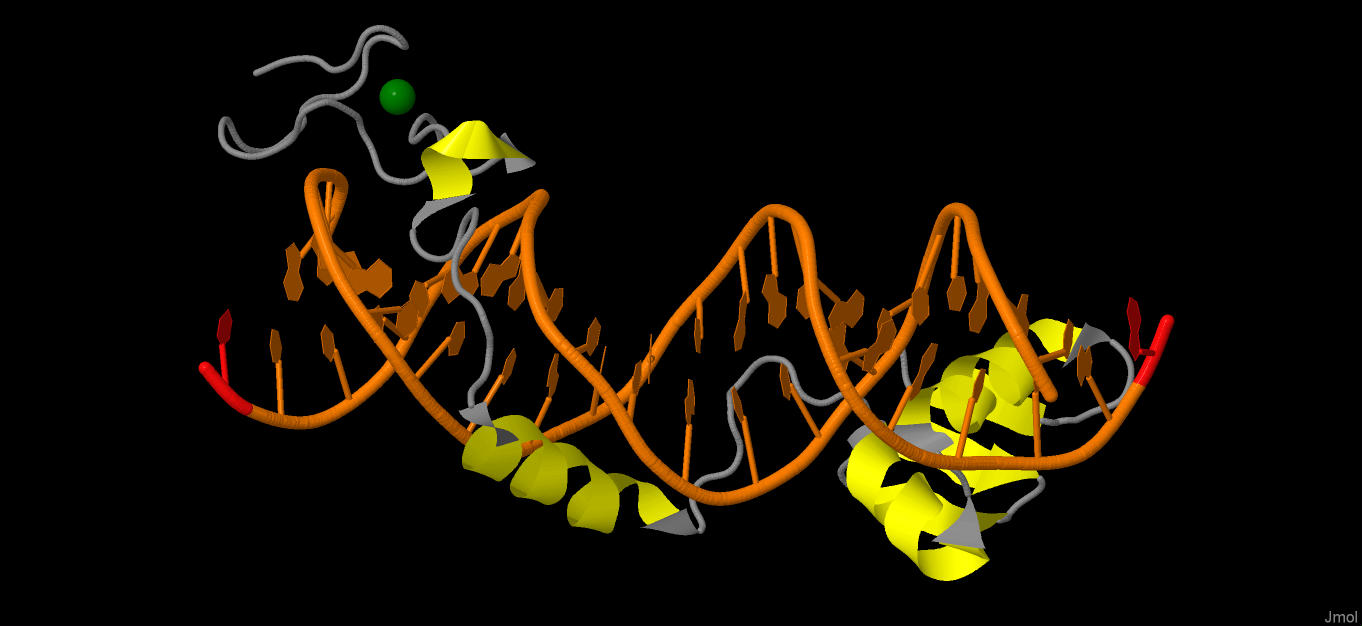
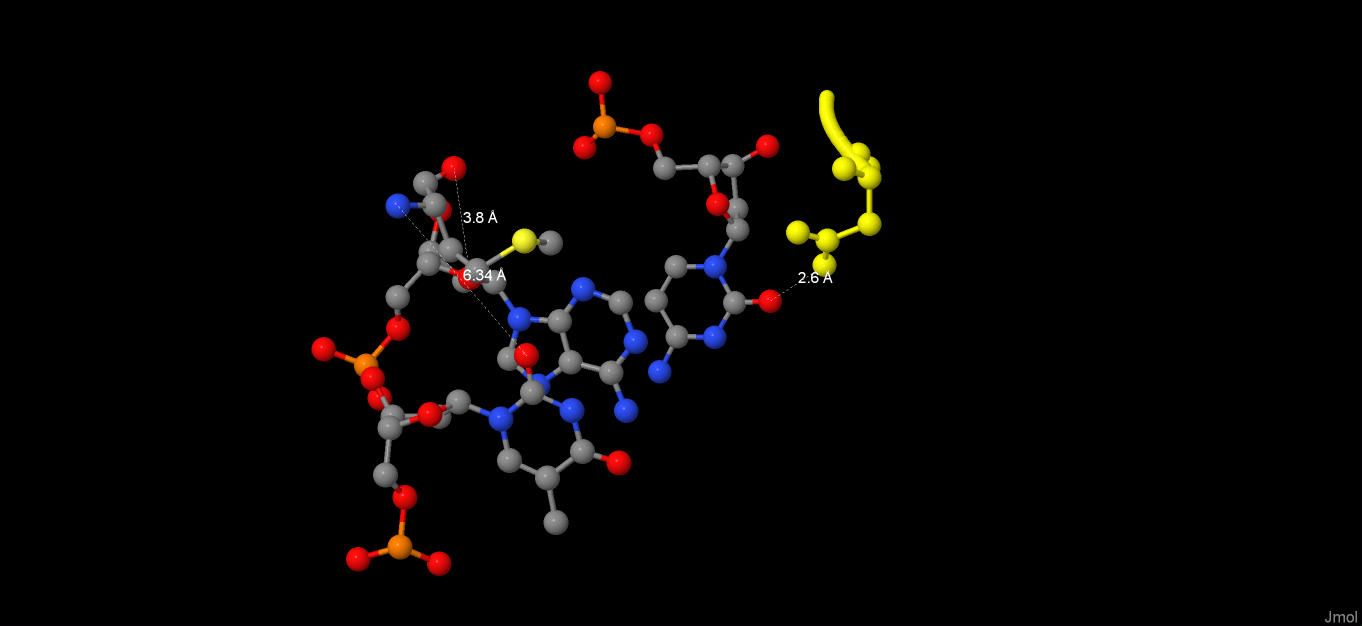 Рисунок 7. Вышеуказанные контакты(2)
Рисунок 7. Вышеуказанные контакты(2)