Нуклеотидный бласт
1. Таксономия и функция прочтенной нуклеотидной последовательности.
Для рассмотрения была выбрана последовательность из шестого практикума - Ae2_18SII_F_E05_WSBS-Seq-1-08-1, правда, уже отредактированная,
согласно требованиям прошлого задания. Ознакомиться с последовательностью можно по ссылке.
Был использован алгоритм megablast(поиск очень похожих последовательностей, чтобы точнее определить организм) Blast выдал следующий результат:
| RID | ZBETFTDGO14 |
| Molecule type | nucleic acid |
| Description | Nucleotide collection |
| Database name | nr |
| Query Length | 611 |
Таблица 1 Результат выдачи BLAST
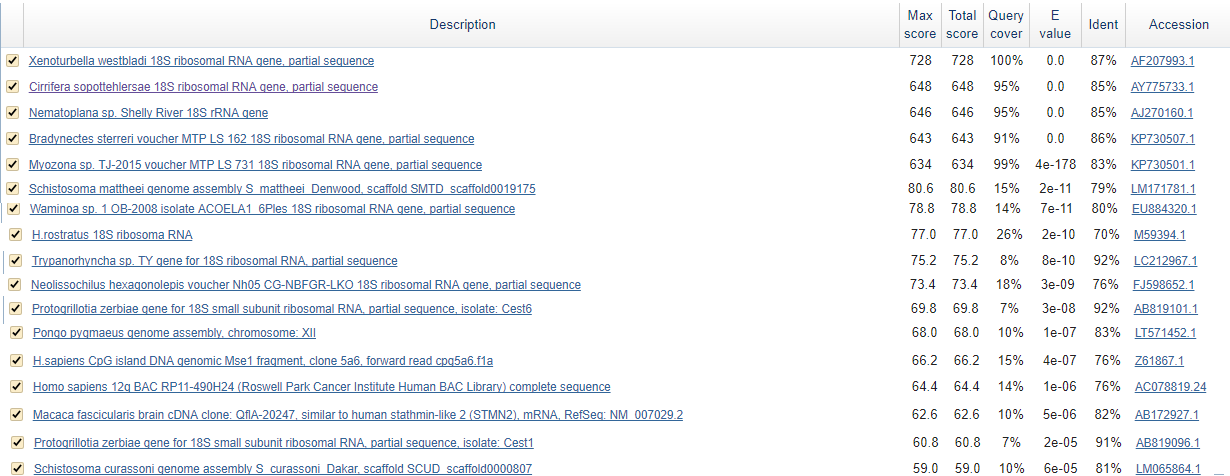
Судя по кандидатам, мы можем заключить, что это ген 18S рибосомальной РНК.
Данная НК c 99% идентичностью (+query cover - 100%) совпадает со
"Scoloplos acutissimus voucher W.44175.001 18S ribosomal RNA gene, partial sequence" - 607 положений из 611.
 Рисунок 1. Совпавшая последовательность.
Рисунок 1. Совпавшая последовательность.
Таким образом, совпадение довольно впечатляющее, однако последовательности не идентичны.
Значит достоверно с точностью нам удалось определить организмы до рода Scoloplos,
но лично мне источник исходной последовательности не известен, поэтому я не берусь утверждать, вызваны ли несовпадения случайными мутациями (что наиболее вероятно),
или это другой вид.
Для этого сравним изменчивость последовательностей от вида к виду, построив выравнивание в JalView.
В BLAST есть возможность построить таксономическое дерево, для начала выберем оттуда несколько последовательностей генов 18S-рибосомальной-РНК из рода Scolopus:
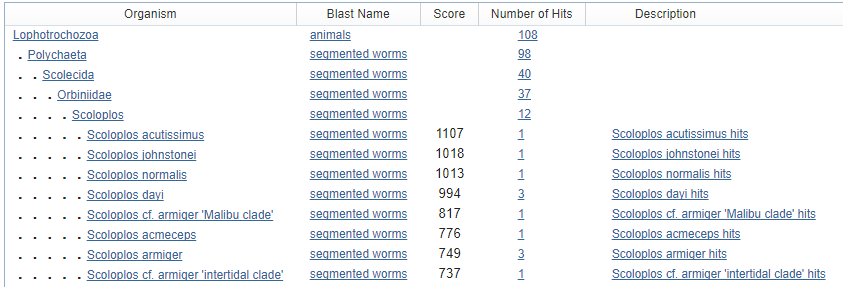 Рисунок 2. Исследуемые Scolopus из автоматического таксономического дерева BLAST..
В принципе, уже можно прдположить, что виды достаточно сильно отличаются, судя по данным. К слову, это один из немногих родов, котоый в таком количестве попал в выборку,
остальные встречаются поштучно.
Рисунок 2. Исследуемые Scolopus из автоматического таксономического дерева BLAST..
В принципе, уже можно прдположить, что виды достаточно сильно отличаются, судя по данным. К слову, это один из немногих родов, котоый в таком количестве попал в выборку,
остальные встречаются поштучно.
Ссылка на выравнивание
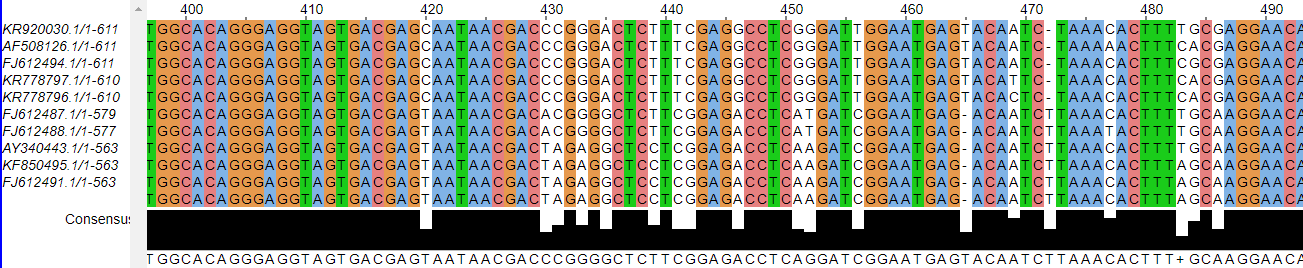 Рисунок 3. Выравнивание Scolopus..
У самых далёких родственников различие идёт на 20-30 аминокислотных замен, но у второй и третьей последовательностей, например, я насчитала всего 5 замен,
а это разные виды.
5 и 4 сравнимые числа, значит нельзя с уверенностью утверждать, что это один и тот же вид.
Рисунок 3. Выравнивание Scolopus..
У самых далёких родственников различие идёт на 20-30 аминокислотных замен, но у второй и третьей последовательностей, например, я насчитала всего 5 замен,
а это разные виды.
5 и 4 сравнимые числа, значит нельзя с уверенностью утверждать, что это один и тот же вид.
Scoloplos - род полихет, кольчатых червей
(Eukaryota; Opisthokonta; Metazoa; Eumetazoa; Bilateria; Protostomia; Lophotrochozoa; Annelida; Polychaeta; Scolecida; Orbiniidae; Scoloplos),
обитатели и глубин, и неритической зоны, то есть ареал обитания имеют достаточно разнообразный, принадлежат к экологической группе детрита.
Конкретно про этот род не так много информации.
2. Сравнение списков находок нуклеотидной последовательности 3-мя разными алгоритмами blast
Для анализа вновь была использована последовательность из
предыдущего практикума.
Сравнение трех алгоритмов представляет из себя запуск BLAST c одинаковыми параметрами, чтобы выявить качественные отличия.
Изначально мы имеем некоторую базовую информацию, что алгоритм megablast более избирателен, нежели другие.
Область поиска была ограничена так, чтобы результат сравнения был показателен - мы поднимались вверх по таксонам до тех пор, пока не увидели значительного
различия в значениях e-value. К сожалению, для этого пришлось сильно постараться, потому что, похоже, это достаточно консервативная. Были предриняты попытки ограничить поиск внутри семейства,
но при исключении более низлежазщих таксонов, даже в большом количестве, E-value всё равно оставали равны 0 у порядка первых 50-100 находок. Поэтому начали двигаться по таксономическому дереву вверх.
Параметры поиска:
- E-value = 0.001
- Огранияенные таксоны - до Bilateria
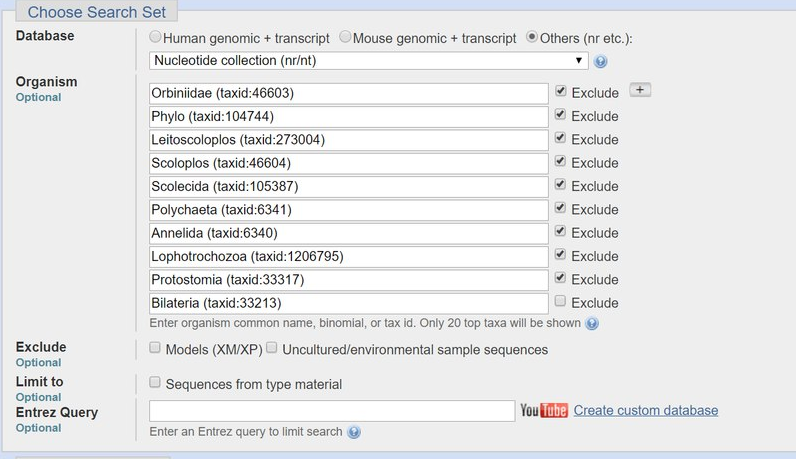 Рисунок 4. Ограничения. Добавлялось новое, пока E-value не изменило значение с 0. Здесь виден весь путь, который для этого пришлось пройти
Рисунок 4. Ограничения. Добавлялось новое, пока E-value не изменило значение с 0. Здесь виден весь путь, который для этого пришлось пройти
Результат для трёх способов поиска:
| megablast | dis-blast | blastn |
| Количество находок | 7053 | 7657 | 7846 |
| Длина слова | 28 | 11 | 11 |
Таблица 2 Сравнение алгоритмов BLAST
Megablast:
 Dis-megablast:
Dis-megablast:
 Blastn:
Blastn:
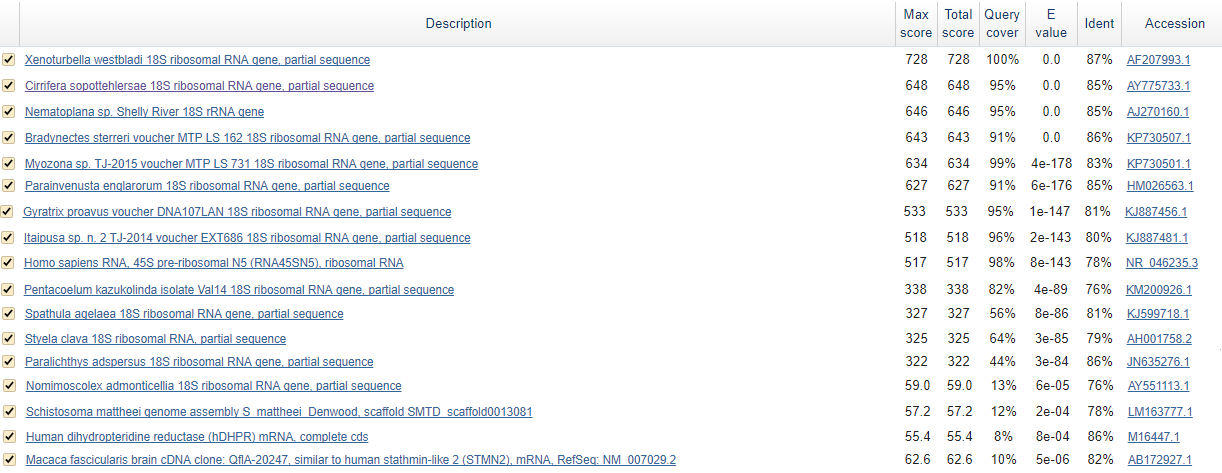 Рисунок 5 Скриншоты выдачи.Частичное представление находок
Рисунок 5 Скриншоты выдачи.Частичное представление находок
Величина выборок растёт от megablast к blastn.
Последний ищет последовательности с меньшим сходством, чем megablast, а discontiguous-megablas это промежуточный вариант, поэтому и выборка у него средней величины между двумя другими. Последовательности, которыми другие бласты восполняют количество относительно megablast - не самые похожие последовательности. От первого до последнего результата E-value имеет меньший разброс именно что у megablast, то есть ближе всего к указанному барьеру 0.001 ближе число именно в dis.../blastn
18S rRNA нам попалась достаточно консервативной, тем не менее разница всё равно видна.
3. Проверка наличия гомологов трех белков в геноме одного организма.
Для анализа был рассмотрен организм X5 (Amoeboaphelidium protococcarum), сборка генома X5 взята с диска P: в директории y15/term3/block2/pr8.
Гомологи искались у следующих белков (последовательности именно этих оказалось проще всего найти):
- TERT_SCHPO - теломераза,восстанавливающая длину хромосомы при репликации; имеется у большинства эукариот
- HIS31_HUMAN - гистон H3, белок, участвующий в образовании нуклеосомы
- TBB_NEUCR - тубулин, белок, участвующий в образовании микротрубочек
Как выяснилось после дальнейшего анализа, два последних белка действительно являются ортологами, когда как в первом, скорее, выявлено случайное сходство.
Результаты local BLAST, проведённом с помощью Putty, можно наблюдать ниже. Были вынесены перые находки. Обратите внимание на их значения идентичности и E-value. На основе этих
данных и было выявлено, насколько последовательности близки.
4. Поиск гена белка, закодированного в одном скэффолде "Amoeboaphelidium protococcarum"
Для поиска был выбран один контиг(случайным образом random rnd) длины порядка десятков тысяч пар нуклеотидов, а точнее - scaffold 258, длины 99529 bp.
Интроны у амебоафилидум короткие, так что мы имeем хороший шанс найти ген в одном контиге.
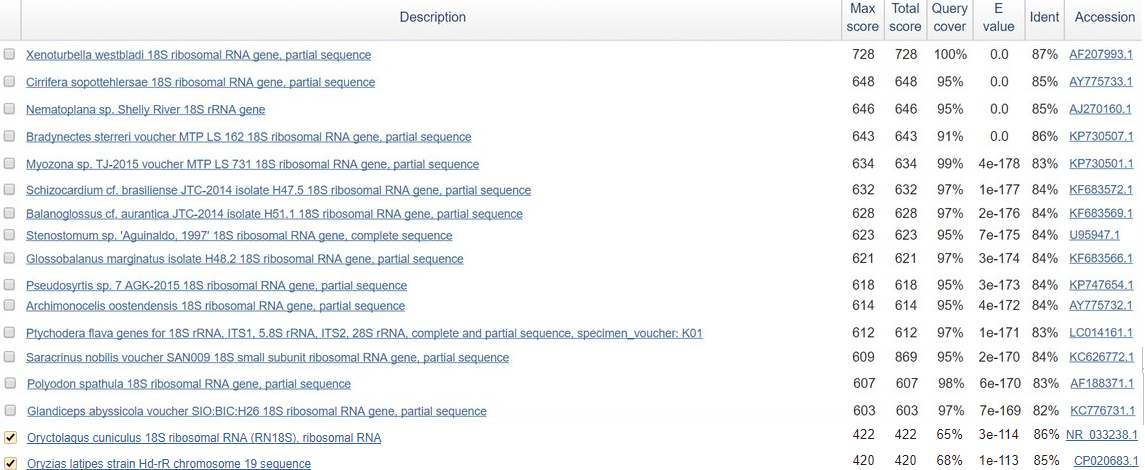
И действительно, выравнивание показало нам лучшую находку:
 Рисунок 6 NCBI-выдача.
Рисунок 6 NCBI-выдача.
Всю актуальную информацию касательно параметров поиска и изученного скэффолда можно свести в таблицу:
| Идентификатор скэффолда | scaffold258 |
| Длина скэффолда | 99529 |
| Разновидность blast | blastx |
| Область поиска | база nr, в таксоне Opisthokonta |
| Лучшее совпадение | PREDICTED: Daucus carota subsp. sativus coatomer subunit alpha-1-like |
| Ориентировачные границы гена | ~446-742 |
| Ориентация | Прямая |
Лучшее совпадение: 2% гэпов, 78 процентов идентичности, остальные 46 находок имеют гораздо меньшее Е-валью, разница - 2e-44
к 41-й степени у остальных. Этот белок описан не так хорошо, как мог бы быть, потому что гипотетический - предсказан компъютерными
вычислениями.
Вернуться назад
На главную страницу
©Solonovich Vera,2017








