Предсказание трансмембранных сегментов почти вручную.
Белок для исследования: AQPA_RANES(SWISS_Prot ID) и P50501(SWISS-Prot AC);Белок-прототип: AQPA_RANES(SWISS_Prot ID) и P50501(SWISS-Prot AC);
PDB: 1J4N.
Шкала гидрофобности аминокислот по Kyte&Doolittle:
Arg R -4.50 Lys K -3.90 Glu E -3.50 Gln Q -3.50 Asp D -3.50 Asn N -3.50 His H -3.20 Pro P -1.60 Tyr Y -1.30 Trp W -0.90 Ser S -0.80 Thr T -0.70 Gly G -0.40 Ala A 1.80 Met M 1.90 Cys C 2.50 Phe F 2.80 Leu L 3.80 Val V 4.20 Ile I 4.50
Представлен график из результата c сопровождением:
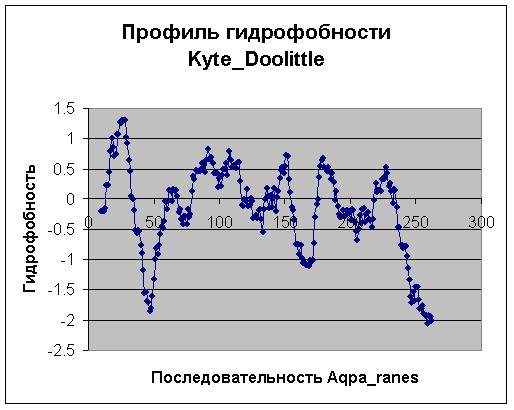 |
| |||||||||||||||||||||||

На представленной последовательности нашего белка отмечены красным цветом трансмембранные участки,
синим — внутренние, розовым — наружные. Данная разметка проводилась по правилу von Hejne,
при этом обращалось внимание на число положительно заряженных остатков (K,R) и на странное перекрытие,
которое отмечено ранее. Т.к. пики на профиле в месте перекрытия одинаковые, то ни один из них нет
основания не учитывать, значит разделим один фрагмент на два сегмента, которые могут пронизывать один
за другим мембрану, а для этого надо удлинить фрагмент — добавленные позиции отмечены зелёным цветом.
P.S. Приношу извинения за не очень хорошее качество изображения, поэтому
ссылка.
Сервер TMHMM предсказывает топологию.
# P50501_AQPA_RANES_ Length: 272 # P50501_AQPA_RANES_ Number of predicted TMHs: 6 # P50501_AQPA_RANES_ Exp number of AAs in TMHs: 126.3067 # P50501_AQPA_RANES_ Exp number, first 60 AAs: 24.10113 # P50501_AQPA_RANES_ Total prob of N-in: 0.61042 # P50501_AQPA_RANES_ POSSIBLE N-term signal sequence P50501_AQPA_RANES_ TMHMM2.0 inside 1 12 P50501_AQPA_RANES_ TMHMM2.0 TMhelix 13 35 P50501_AQPA_RANES_ TMHMM2.0 outside 36 73 P50501_AQPA_RANES_ TMHMM2.0 TMhelix 74 96 P50501_AQPA_RANES_ TMHMM2.0 inside 97 100 P50501_AQPA_RANES_ TMHMM2.0 TMhelix 101 123 P50501_AQPA_RANES_ TMHMM2.0 outside 124 137 P50501_AQPA_RANES_ TMHMM2.0 TMhelix 138 160 P50501_AQPA_RANES_ TMHMM2.0 inside 161 171 P50501_AQPA_RANES_ TMHMM2.0 TMhelix 172 194 P50501_AQPA_RANES_ TMHMM2.0 outside 195 213 P50501_AQPA_RANES_ TMHMM2.0 TMhelix 214 236 P50501_AQPA_RANES_ TMHMM2.0 inside 237 272
И сопроводительная картинка:
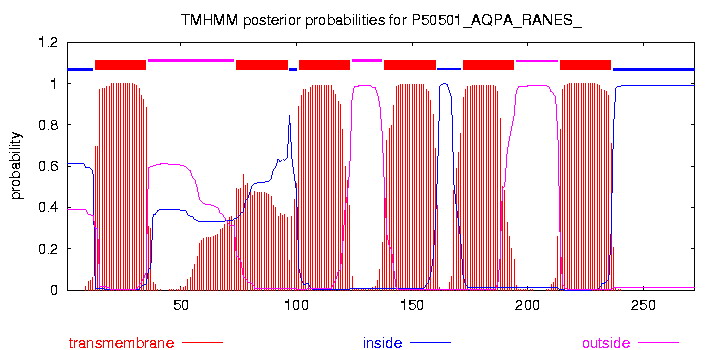
Комментарий:
Оказывается, в месте перекрывания у нас, действительно, есть 2 спирали, которые могут пронизывать мембрану, правда одной из них сервер доверяет больше, чем другой. Интересно, что между этими спиралями существует 4 аминокислотных остатка, среди них лизин. Наверное, можно предположить, что спираль, только выйдя из мембраны, тут же в неё входит; в соответствии с правилом von Hejne, скорее всего, изгиб обращён в цитоплазму. Редактирование в GeneDoc.
Выделение трансмембранных сегментов в 3D–структуре прототипа.
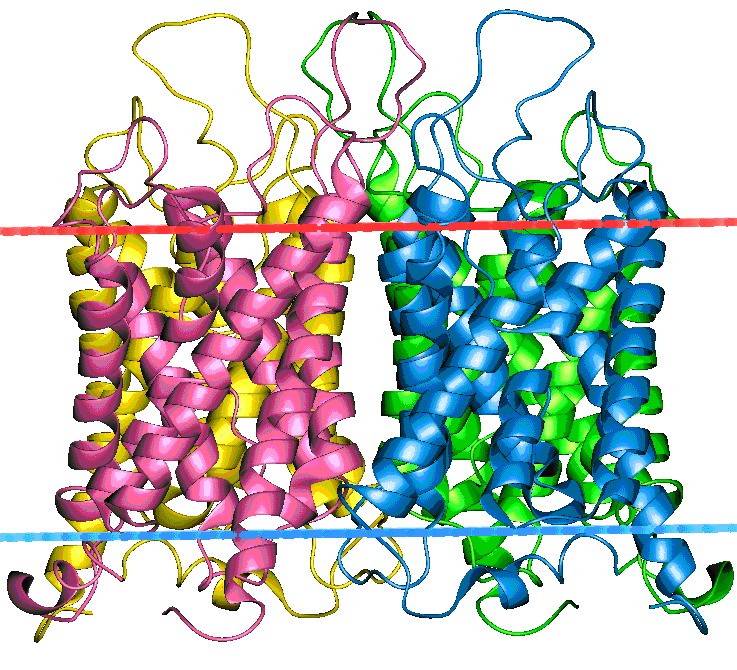 |
Excel — ТМ сегменты по данным ОРМ Новая строчка в GeneDoc. |
Сравнение предсказанных топологий с описанием трансмембранных сегментов в 3D-структуре прототипа.
| По профилю гидрофобности | TMHMM | ||
| Всего предсказано аминокислотных остатков (N) | 128 | 138 | |
| Из них | |||
| А. По данным ОРМ находятся внутри мембраны | 99 | 101 | |
| В. Из них - "торчащие" из мембраны остатки на концах спиралей | 10 | 16 | |
| С. Вообще не имеют никакого отношения к трансмембранным спиралям | 19 | 21 | |
| D. Число непредсказанных остатков, которые по данным ОРМ находятся в мембране. | 43 | 41 | |
| Точность предсказания, A/N | 0,77 | 0,73 | |
| Сверхпредсказание, C/N | 0,15 | 0,15 | |
| Недопредсказание, D/(L-N), где L-длина последовательности(=272) | 0,3 | 0,3 | |
Дополнительная информация об α–спиралях изучаемого белка, взятая из PDB-файла:
HELIX 1 1 PHE A 5 GLY A 34 1 30 HELIX 2 2 PHE A 35 TYR A 37 5 3 HELIX 3 3 ASP A 50 GLY A 74 1 25 HELIX 4 4 ASN A 78 SER A 88 1 11 HELIX 5 5 SER A 92 THR A 118 1 27 HELIX 6 6 GLY A 138 GLY A 140 5 3 HELIX 7 7 LEU A 141 THR A 159 1 19 HELIX 8 8 SER A 169 GLY A 190 1 22 HELIX 9 9 ASN A 194 THR A 205 1 12 HELIX 10 10 TRP A 212 PHE A 231 1 20 HELIX 11 11 ASP A 239 LYS A 245 1 7 HELIX 12 12 VAL A 246 THR A 248 5 3
Она была необходима для установления "торчащих" остатков на концах спиралей: одного лишь перепредсказания по ОРМ здесь не достаточно и, разумеется, нужно было проверить, принадлежит ли данный остаток какой-либо спирали.
Cудя по результатам, предсказания по профилю гидрофобности немного точнее, чем по TMHMM, хотя это почти одно и то же. Наибольшую уверенность вселяет предсказание по OPM, где анализируется 3D–структура.