Составление выборки аминокислотных последовательностей.
Таксоны:| Caprinae | Caudata |
 |
  |
Выборка состоит из 3–х частей:
- белки семейства глобинов, PF00042 (seqtaxon.txt),
из заданных таксонов.
Примечание. Запрос сначала выглядел следующим образом:
Query "(([uniprot-DBxref:PF00042] > parent ) & ([uniprot-Taxonomy:Caprinae*] | [uniprot- Taxonomy:Caudata*])) " found 43 entries ,
затем были отсеяны фрагменты длиной менее 90 а.о. и из двух подвидов одного вида был выбран тот, в котором больше белков данного семейства. Для этого использовалась сортировка по длине и сортировка по названию организма. - глобины человека - Q8WWM9, P09105, P02008, P69905, P68871, P02042, P02100, P69891, P69892,
Q9NPG2 (seqhomo.txt).
Примечание. В указанном файле два последних фрагмента Q14475_HUMAN и Q6QLV0_HUMAN длиной 39 и 42 соответственно были оставлены по 2 причинам: 1. по заданию только первую часть выборки надо фильтровать, 2. в обоих документах есть ссылки на один и тот же номер PF00042 из Pfam. Сам запрос выглядел так:
Query "(((((((((([uniprot-AccNumber:Q8WWM9*] | [uniprot-AccNumber:P09105*]) | [uniprot- AccNumber:P02008*]) | [uniprot-AccNumber:P69905*]) | [uniprot-AccNumber:P68871*]) | [uniprot- AccNumber:P02042*]) | [uniprot-AccNumber:P02100*]) | [uniprot-AccNumber:P69891*]) | [uniprot- AccNumber:P69892*]) | [uniprot-AccNumber:Q9NPG2*]) & [uniprot-Taxonomy:Homo*]) " found 10 entries - внешняя группа (outgroup) - P02144, P02202, Q9DGJ1 (seqout.txt).
Примечание. При поиске никакой таксон не указывался:
Query "(([uniprot-AccNumber:P02144*] | [uniprot-AccNumber:P02202*]) | [uniprot-AccNumber:Q9DGJ1*]) " found 3 entries
Построение филогенетического дерева.
Выборка всех последовательностей в формате FASTA была подана на вход программе emma, которая построила множественное выравнивание (все параметры были заданы по умолчанию). По полученному выравниванию программа eprotdist построила матрицу попарных расстояний между белковыми последовательностями. Далее реконструирование филогенетического дерева по алгоритму ближайших соседей (Neighbour-Joining) было произведено с помощью программы eneighbor.
Изображение дерева:
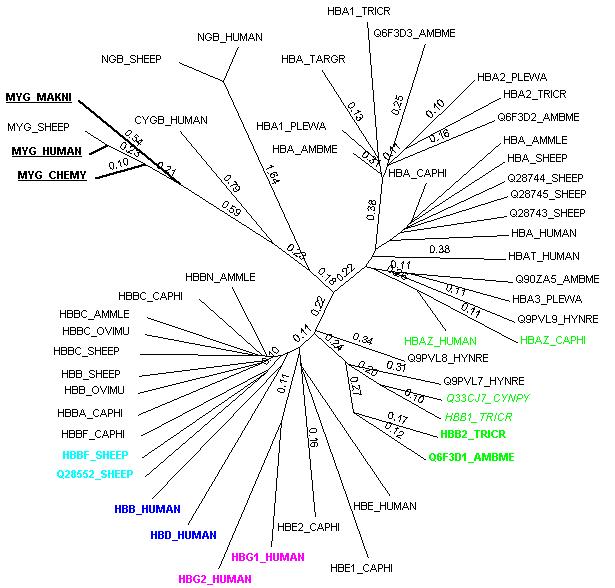
Ветви, соответствующие внешней группе, выделены жирными линиями, а соответствующие подписи вершин — жирным шрифтом и подчёркиванием.
Паралоги — это гомологичные аминокислотные последовательности из одного организма, появившиеся в результате дупликации гена. Примеры их были найдены в соответствии с определением и выделены синим, бирюзовым и сиреневым цветами.
Ортологи — гомологичные последовательности из разных организмов, имеющие, однако, общую предковую последовательность. Ортологами мы можем считать, например, пары, выделенные светло–зелёным цветом (три пары: обычным, курсивным и жирным шрифтом).
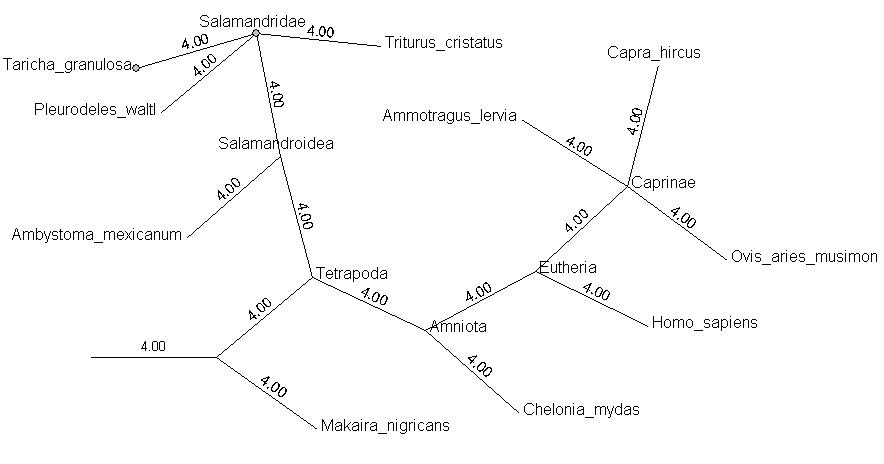
С помощью сервера NCBI было также построено таксономическое дерево по названию организмов, последовательности из которых мы использовали ранее: