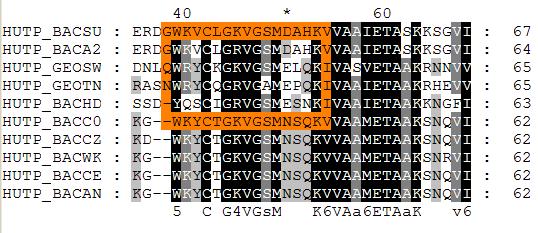
| Характеристика паттерна |
Паттерн |
В скольких последовательностях банка Swiss-Prot
найден мотив, удовлетворяющий паттерну? |
Все ли последовательности из Вашего выравнивания найдены? |
| Фрагмент последовательности |
G-W-K-V-C-L-G-K-V-G-S-M-D-A-H-K-V |
1 |
Найдена только моя последовательность |
| Сильный |
[GQN]-[WY]-[KRQ]-[VYS]-C-[LKIQT]-G-[RK]-V-G-[SA]-M-[DENq]-[ALPS]-[HQN]-K-[VI] |
20 |
Найдены все последовательности из моего выравнивания и близкородственные (HUTP_BAC**), которые я намеренно не включала в выравнивание. |
| Слабый |
[WY] - [KRQ] - [VYLST] - C - X - G - [RK] |
42 |
Нашлись все белки выравнивания, но и много других. |
Prosite находит 25 белков с функцией моего белка, тогда как PSI-Blast находит 12.
Из всех находок слабого выравнивания выделяются 11 длинных (900 остатков) бактериальных белков (SECA_***** Protein translocase subunit secA).