У данного белка две ферментативные активности:
|
 |
| KEGG ID | Английское название | Русское название |
|---|---|---|
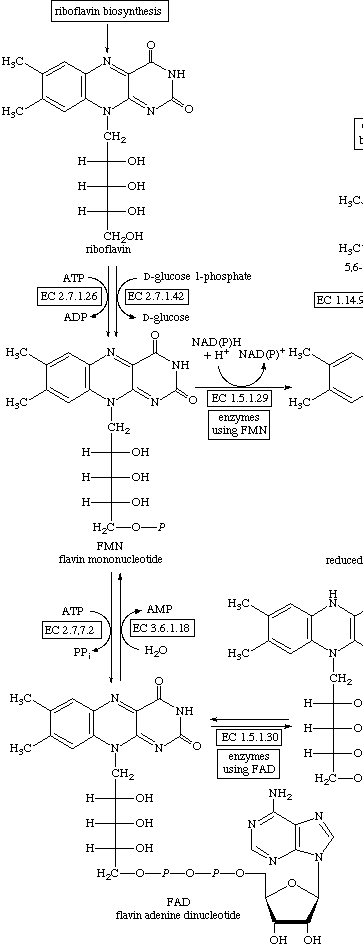
| eco00740 | Riboflavin metabolism | Метаболизм рибофлавина |
| eco01100 | Metabolic pathways | Метаболические пути |
| Русское название | Английское название | ID | Структурная формула |
|---|---|---|---|
| (S)-малат | (S)-malate | C00149 | 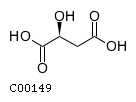 |
| Пируват | Pyruvate | C00022 |  |
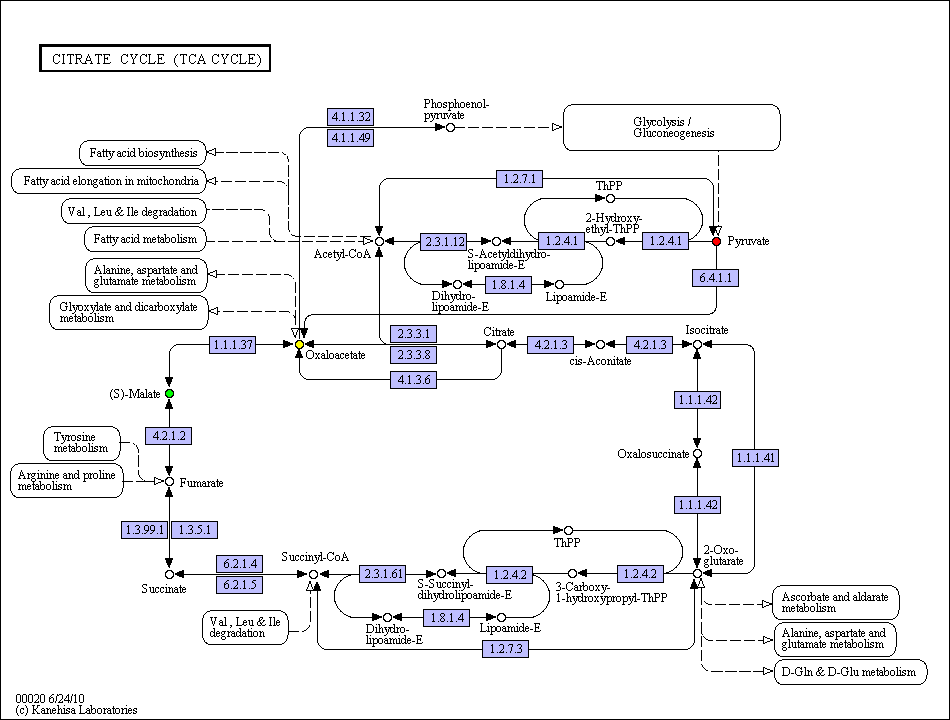
Выбранный путь: Citrate cycle (TCA cycle)
Цепочка: пируват -> (S)-малат.
Промежуточное соединение - щавелевоуксусная кислота.

| Организм | Возможна ли цепочка реакций (да/нет/неизвестно) |
Обоснование |
| Escherichia coli K-12 MG1655 | нет | Неизвестен фермент, осуществляющий первую стадию (пируват -> щавелевоуксусая кислота) EC 6.4.1.1 |
| Archaeoglobus fulgidus | да | |
| Arabidopsis thaliana | нет | Неизвестен фермент, осуществляющий первую стадию (пируват -> щавелевоуксусая кислота) EC 6.4.1.1 |
| Homo sapiens | да |
Карта процесса для H. sapiens
- ЕC 4.1.1.23 (оротидин-5'-фосфат декарбоксилаза)
- Запрос SRS: (([uniprot-ID:*_HUMAN*] | [uniprot-ID:*_ARCFU*]) & [uniprot-ECNumber:4.1.1.23*])
- Сняли опцию маски чтобы искать белки с заданным EC, а не с примерно похожим.
- Нашлись 5 белков: B5LY70_HUMAN, B5LY64_HUMAN, PYR5_HUMAN, PYRF_ARCFU и B5LY72_HUMAN.
- Все белки имеют общий домен - PF00215 (Orotidine 5'-phosphate decarboxylase domain)
Кроме того, PYR5_HUMAN имеет дополнительный домен PF00156 (Phosphoribosyltransferase), что наверное соответствует второй ферментативной активности данного белка. А также B5LY72_HUMAN имеет удвоенный основной домен PF00215, во что не очень верится: белок самый маленький из всех 5 (200 а.о. а у всех остальних человеческих - минимум 300). Может, наоборот, там один раздробленный домен, который Pfam принимает за два? P.S. я посмотрела, так и есть. - Для дальнейшей работы выбрали PYRF_ARCFU и PYR5_HUMAN т.к. только для них было указано имя локуса гена (afu:AF0929 и hsa:7372)
- Для PYRF_ARCFU лучший ортолог из эукариот tan:TA16510; организм - Theileria annulata; identity - 0,258; overlap - 198 при длине самого белка - 205.
- Для PYR5_HUMAN лучший ортолог из архей pab:PAB2430 ; организм - Pyrococcus abyssi; identity - 0.351; overlap - 171 при длине самого белка - 480.
- Для человека ортолог нашёлся явно лучше, чем для археи. Но так как в человеческом белке два домена, мы не можем гарантировать что выровнялся нужный. Найденные ортологи показывают, что два белка с одинаковой ферментативной активностью могут иметь очень различающиеся последовательности.