Реконструкция филогении по нуклеотидным последовательнсотям
Правильное дерево:
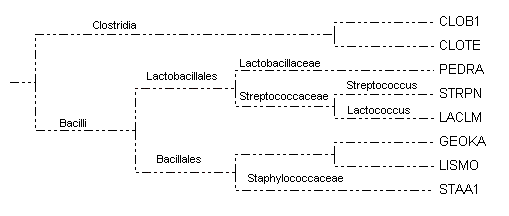
Я реконструировала дерево программой fdnapars. Программа выдала два дерева:
+---STAA1 +---GEOKA
+--6 |
+--5 +---LISMO +-----3 +--STAA1
| | | | +--6
| +--GEOKA | | | +--LISMO
+-----3 | +--5
| | +---PEDPA | | +---PEDPA
| +--4 | +--4
| | +---STRPN | | +---STRPN
| +--2 | +--2
| +---LACLM | +---LACLM
| |
1--CLOTE 1--CLOTE
| |
+-CLOB1 +-CLOB1
Как видно, деревья не совпадают с правильным. Причём оба не совпадают положением листа GEOKA.
Таким образом, ни одним сопособом реконструкции не удалось установить правильное взаимное расположение
GEOKA, LISMO и STAA1, однако fdnapars единственной удалось хотя бы выделить GEOKA, LISMO, STAA1 в отдельную
правильную ветвь - {GEOKA, LISMO, STAA1} против {CLOB1, CLOTE, STRPN, LACLM, PEDRA}.
Из-за этого, результат работы fdnapars мне показался наиболее удачным.
Построение и анализ дерева, содержащего паралоги
Находки Blast:
Subject id % identity e-value bit score
sp|A7FYI1|CLPX_CLOB1 71.75 5e-157 549
sp|Q891J8|CLPX_CLOTE 71.93 7e-159 555
sp|A7X396|CLPX_STAA1 71.50 1e-161 564
sp|Q8Y7K9|CLPX_LISMO 82.42 0.0 632
sp|Q03F27|CLPX_PEDPA 67.87 4e-152 533
sp|Q8GJP6|CLPX_LACLM 68.64 2e-149 524
sp|P63791|CLPX_STRPN 66.92 6e-143 502
sp|Q8Y7J8|HSLU_LISMO 45.63 2e-20 95.5
sp|A7X1N1|HSLU_STAA1 42.98 1e-19 92.8
sp|Q5L0N1|HSLU_GEOKA 42.72 1e-19 92.4
Реконструкция дерева программой fprotpars
+-----HSLU_LISMO
+-----------9
! ! +--HSLU_GEOKA
! +--8
! +--HSLU_STAA1
+-----7
! ! +--CLPX_CLOTE
! ! +--6
! ! +--5 +--CLPX_CLOB1
! ! ! !
+--3 +--------4 +-----CLPX_LISMO
! ! !
! ! +--------CLPX_STAA1
! !
1 ! +--CLPX_STRPN
! +--------------------2
! +--CLPX_LACLM
!
+--------------------------CLPX_PEDPA
Ортологи: CLPX_STRPN и CLPX_LACLM; CLPX_CLOTE и CLPX_CLOB1; HSLU_GEOKA и HSLU_STAA1.
Паралоги: HSLU_STAA1 и CLPX_STAA1; HSLU_LISMO и CLPX_LISMO.
© Медведева
