| Главная | Семестры | Проекты | Обo мне | Ссылки | Заметки | Назад к оглавлению |
Multiple alignment
Некоторая нужная информация для иследований:
- Филумы - самые крупные единицы дробления у прокариот. Организмы из разных филумов разнообразнее по типам метаболизма, а соответственно и по биологическим последовательностям, чем эукариоты, но можно очень грубо сказать, что деление на филумы похоже на деление эукариот на грибы, растения, животные и несколько групп одноклеточных простейших.
Составление репрезентативной выборки гомологов белка PDAA_BACSU с помощью BLAST
Для выборки определяем филум для Bacillus subtilis - организма, который содержит PDAA_BACSU (мой белок). Им является Firmicutes. Осуществляем поиск BLAST, в качестве исходной последовательности используя PDAA_BACSU. Для поиска по прокариотам: изменяем стоящие по умолчанию параметры: изменяем максимальное количество хитов с 100 на 1000. При поиске в поле organism вводим найденный филум, ставим галочку напротив "Exclude" (запрещаем BLAST выдавать сходные белки из того же филума), добавляем еще поле organism и им исключаем эукариот, порог e-value поставим 1, в качестве базы данных используем RefSeq (достаточно крупная и с полными геномами). Пробуем. У последнего результата e-value оказался равным 3e-14 - список обрывается на приличном e-value и хороших выравниваниях следовательно изменяем максимальное количество хитов на 5000. Пробуем. Самый последний результат имеет e-value 0.004, изменяем количество хитов на 10000. Опять пробуем. Последний с e-value=1 - хорошо, мы получили тот результат, который хотели максимальным. Пробуем получить Дерево с индификаторами последовательностей, для этого нажимаем в секции "Описание" все последовательности и получаем о них выдачу GenBank. Он выдает ошибку, видимо, слишком много последовательностей. Да. Их 3280. Многовато. Сокращаем поиск до e-value равного 6e-17, начиная от этой величины идут более хорошие выравнивания и их существенно меньше (700). Получаем древо жизни (рис. 1). Для поиска по эукариотам: в поле organism вводим Eukaryota и не ставим галочку напротив Exclude, выбираем порог e-value равный 0.01 и максимальное количество хитов 1000. Получаем 329 результатов. Смотрим на древо жизни (рис. 2). Таблицу 1 с финальными параметрами BLAST и филогенетические деревья можно увидеть ниже (рис. 1 и 2). Если хотите можете так же посмотреть другие филогенетические деревья, полученные с помощью GenPept для прокариот и эукариот из которых впоследствии выбирались организмы. Для этого нажмите на соответсвующую империю.
Вы можете посмотреть таблицу 1, описывающую финальные параметры BLAST, использованные для поиска:
| Поиск | Алгоритм BLAST | Название базы данных | Ограничения по таксонам | Порог e-value | Количество найденных гомологов и максимальное поличество хитов |
| По прокариотам | BlastP | RefSeq | Exclude Firmicutes + Exclude Eukaryota | 6e-17 | 700 и 1000 |
| По эукариотам | BlastP | RefSeq | Eukaryota | 0.01 | 329 и 1000 |
Пройдя по этой ссылке вы можете получить ссылки на выдачи BLAST по 1)про- и 2)эукариотам.
Филогенетические деревья белка PDAA_BACSU для Prokaryota и Eukaryota
 |

|
| Рис. 1. Филогенетическое дерево белка PDAA_BACSU для Prokaryota |
 |

|
| Рис. 2. Филогенетическое дерево белка PDAA_BACSU для Eukaryota |
|
Построение множественного выравнивания (см. далее) основывается на последовательностях голомогичных белку PDAA_BACSU (табл. 2).
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Таблица 2. Встречаемость белков, гомологичных PDAA_BACSU, в различных таксонах про- и эукариот. Фиолетовым обозначен сам белок PDAA_BACSU. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Судя по записям в поле "COMMENT" при переходе по ссылке "DBSOURCE" у разных царств эукариот нет нет указаний о том, что этот белок принадлежит митохондрии или хлоропласту, он ядерный а значит это позволяет нам предположить, что это исходно эукариотический белок.
Множественное выравнивание гомологов белка PDAA_BACSU и мои наблюдения
Для множественного выравнивания используются белки из организмов, названия организмов приведенные в таблице 2. Среди них есть белок PDAA_BACSU, с которым мы выравниванием его белки-гомологи: среди прокариотических взато 2 из архебактерий и 26 из бактерий (всего выходит 28) и среди эукариотических белков 8 (при клике можно посмотреть конкретные последовательности в fasta-формате). Выравнивание осуществляется с помощью программы Muscle EBI (Европейского Биоинформатического Института), его можно увидеть на рисунке 3.
У выравнивания есть строки анотации. Они находятся под ним; в них можно узнать различную информацию о выравнивание:
- BLOCKS: показывает блоки выравнивания. Блоки выбирались не только с абсолютно идентичными аминокислотами, но и те, что имели не большое число отклонений от преобладающей кислоты.
- LIGANDS: показывает место срязывания белка с лигандами (ионы кадмия).
- SECONDARY: показывает информацию о трехмерной структуре белка - красным обозначены альфа-спирали, зеленым - бета-листы.
- Conservation: позволяет судить о консервативности каждого остатка по высоте - чем выше, тем более консервативен.
- Quality: показывает качество выравнивания.
- Consensus: показывает консервативность каждой последовательности и указывает преобладающую на этом участке аминокислоту.
 |
|
 |
|
| Рис. 3. Множественное выравнивание белка PDAA_BACSU с гомолагами из разных групп огранизмов. Первые 2 последовательности принадлежат Archaea, следующие 26 - Bacteria, а последние 8 - Eukaryotes. На первом изображении можно увидеть полное выравнивание с выставленным процентом консервативности по окраски равным 30%, а на втором изображении - наиболее хороший участок выравнивания с двумя аминокислотами из двух, которые учавствуют в связывание ионов кадмия, а процент консервативности здесь выставлен меньшим и он равен 17%. | |
 |
 |
 |
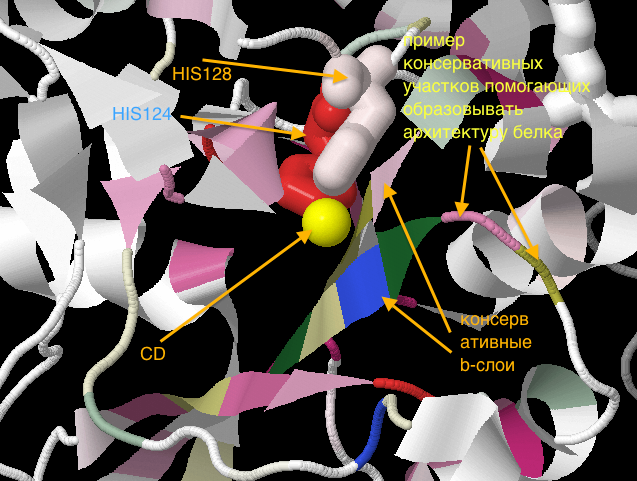 |
| Рис. 4-7. Изображен белок с раскрашеными консервативными участками выравнивания, на рисунках обозначены аминокислоты расположенные рядом с лигандом (ион кадмия). На первом и втором рисунке хорошо видно общую структуру цепи (на заднем плане изображен серым кусочек второй цепи - она идентична 1 (они у меня так и называются: 1 и 2, см. здесь)). А на третьем и четвертом - приближенное изображение. | |
