| Главная | Семестры | Проекты | Обo мне | Ссылки | Заметки | Назад к оглавлению |
А- и В- формы ДНК. Структура РНК.
На этой странице анализируются и А, B, Z формы ДНК и РНК.
С помощью инструментов пакета 3DNA программы Putty были получены 3 формы ДНК. Действия:
- подключаемся к серверу kodomo.cmm.msu.ru используя протокол ssh
- переходим в нужную директорию
- указываем путь к пакету 3DNA:
export PATH=${PATH}:/home/preps/golovin/progs/X3DNA/bin export
X3DNA=/home/preps/golovin/progs/X3DNA - запускаем описание програмы fiber: fiber -h
- создаем A-ДНК, B-ДНК с последовательностью gatc, повторенной 5 раз
- создаем Z-ДНК
- сохраняем полученные структуры в соответствующих файлах gatc-*.pdb, где * - форма ДНК.
A-DNA
Выделяем различные атомы и химические группировки в A-ДНК, используя программу JMol (рисунки 1-4).
 |  |
| Рис. 1. Сахарофосфатный остов ДНК (красный). |
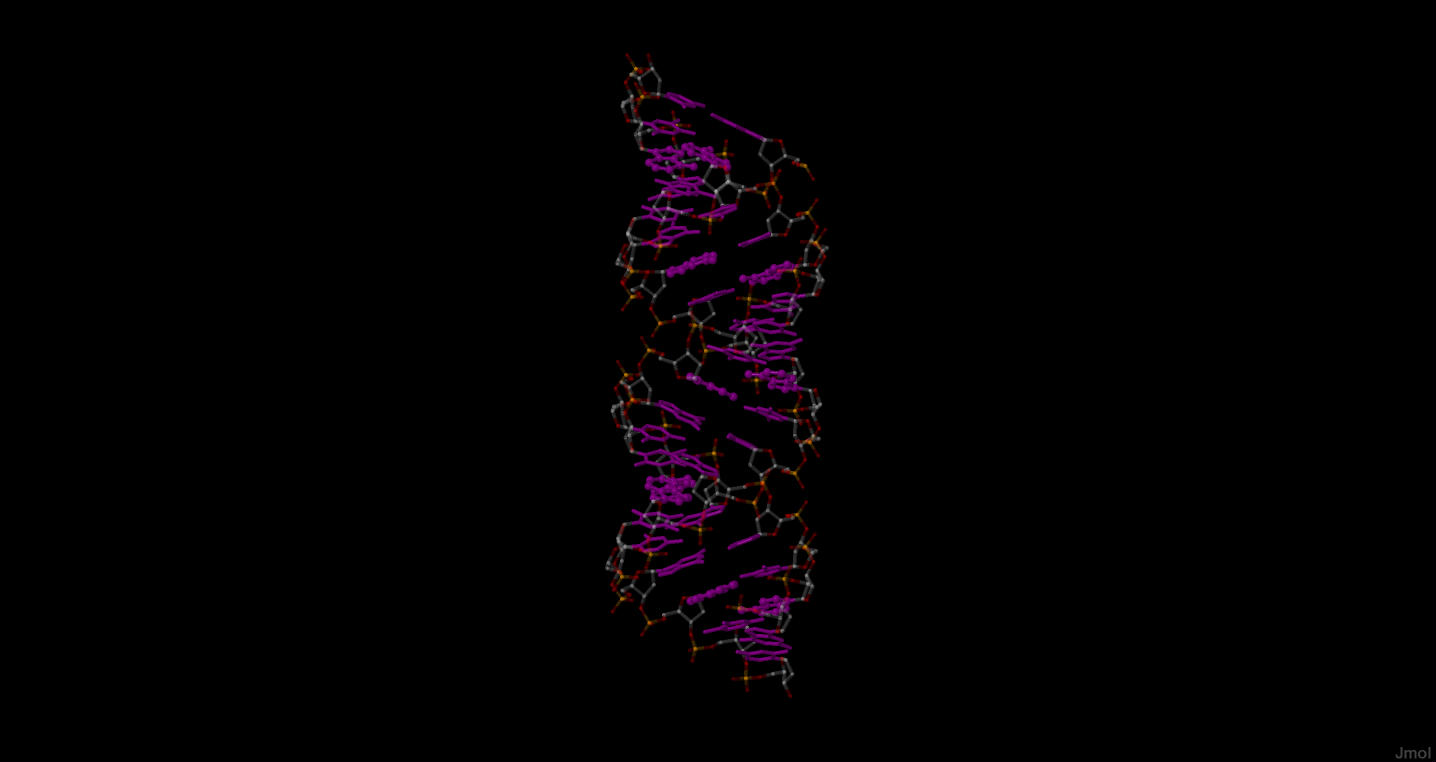 |  |
| Рис. 2. Все нуклеотиды (фиолетовые). |
 |  |
| Рис. 3. Все аденины (зеленые). |
 | 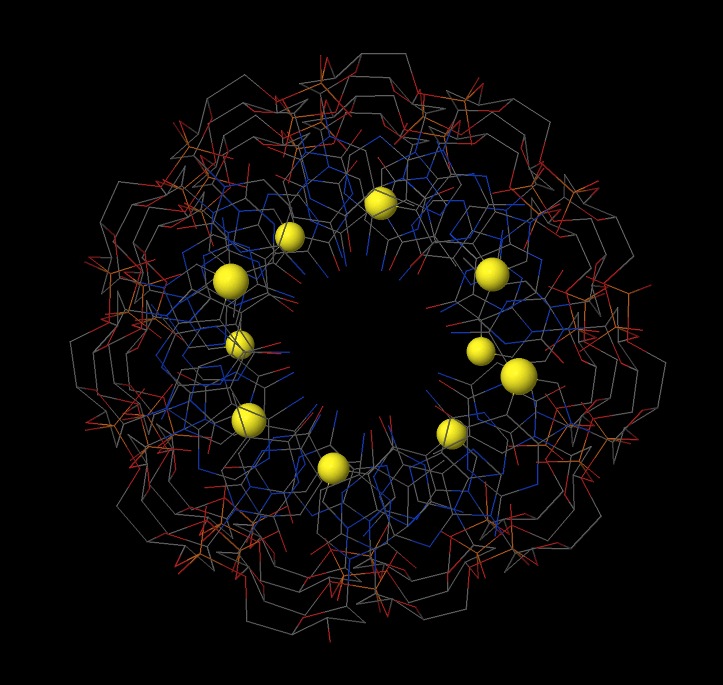 |
| Рис. 4. Атом N7 во всех гуанинах (желтый). |
Структуры ДНК и РНК
На сайте http://www.rcsb.org/pdb/ скачала PDB-файлы 1H4S.pdb и 1MHD.pdb, в которых находится структура РНК-белкового и ДНК-белкового комплексов соответственно.
1. 1H4S
Структура 1H4S изначально была в виде cartoons (рис. 5), оставляю на экране только РНК (рис. 6), изменяю структуру на проволочную (рис. 7).
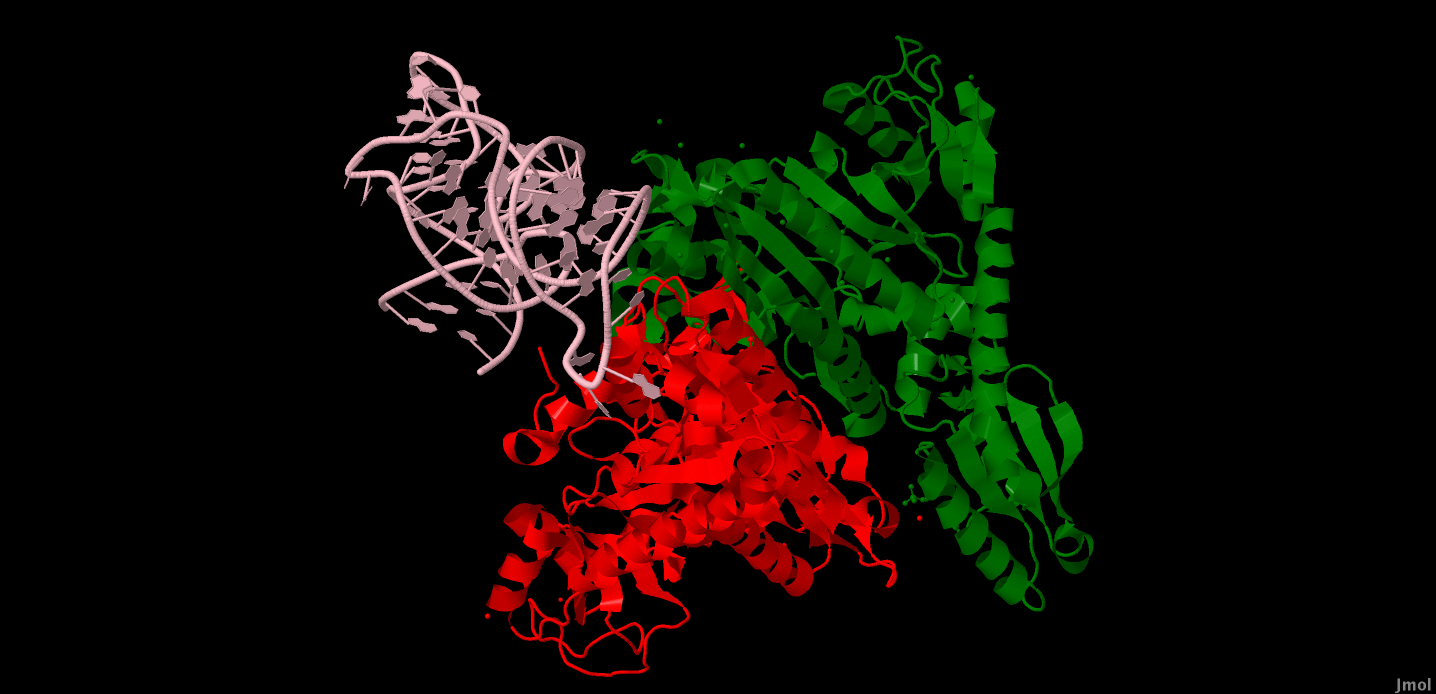 |
| Рис. 5. РНК и белок в виде cartoons. Розовым выделена РНК, зеленым А-цепь белка, а красным - B. |
 |
| Рис. 6. Только РНК. |
 |
| Рис. 7. Проволочная РНК. |
Разрывы отсутствуют в структуре 1H4S.
2. 1MHD
Проводим аналогичные опирации с ДНК (рис. 8). Разрывы также отсутствуют.
 |
| Рис. 8. Проволочная ДНК. |
Сравнение структур 3-х форм ДНК с помощью JMol.
Упр. 1
В файле gatc-b.pdb определяем, где находится большая и маленькая бороздки ДНК. Находим в структуре цитозин, например 12, и определяем какие атомы у него будут обращены в сторону большой бороздки (красный), а какие в сторону малой (синий):
 |  |
| Цитозин. Слева сриншот из JMol, а справа его схематичное изображение из ChemSketch. |
| Форма ДНК | В сторону малой бороздки: | В сторону большой бороздки: | Другие атомы основания: |
| A-форма | c12.c5, c12.c4, c12.n4 | c12.n1, c12.c2, c12.o2, c12.n3 | c12.c6 |
| B-форма | c12.n1, c12.c2, c12.o2 | c12.c6, c12.c5, c12.c4, c12.n4 | c12.n3 |
| Z-форма | c6.n1, c6.c2, c6.o2 | c6.c5, c6.c4, c6.n4 | c6.c6, c6.n3 |
Упр. 2
Сравнение основных спиральных параметров разных форм ДНК:
| A-форма | B-форма | Z-форма | |
| Тип спирали (правая или левая) | правая | правая | левая |
| Шаг спирали (ангстрем) | 28.03 | 33.75 | 43.12 |
| Число оснований на виток | 11 | 10 | 12 |
| Ширина большой бороздки (ангстрем) | 16.97 | 17.76 | 18.30 |
| Ширина малой борозки (ангстрем) | 7.98 | 11.69 | 8.87 |
Упр. 3
Сравнение торсионных углов в структурах А- и В-форм ДНК:
Измерение торсионных углов в цитозине 12 А-ДНК можно увидеть на рисунке 9.
| P - O5' | O5' - C5' | C5' - C4' | C4' - C3' | C3' - O3' | O3' - P | C1' - N | |
| A-ДНК | -51.7 | 174.8 | 41.7 | 79.1 | 147.7 | -75.1 | -157.2 |
| B-форма | -29.9 | 136.3 | 31.2 | 143.3 | -140.8 | -160.5 | -98.0 |
| Значения, приведенные в презентации: | |||||||
| A-ДНК | -62 | 173 | 52 | 88 или 3 | 178 | -50 | -160 |
| B-ДНК | -63 | 171 | 54 | 123 или 131 | 155 | -90 | -117 |
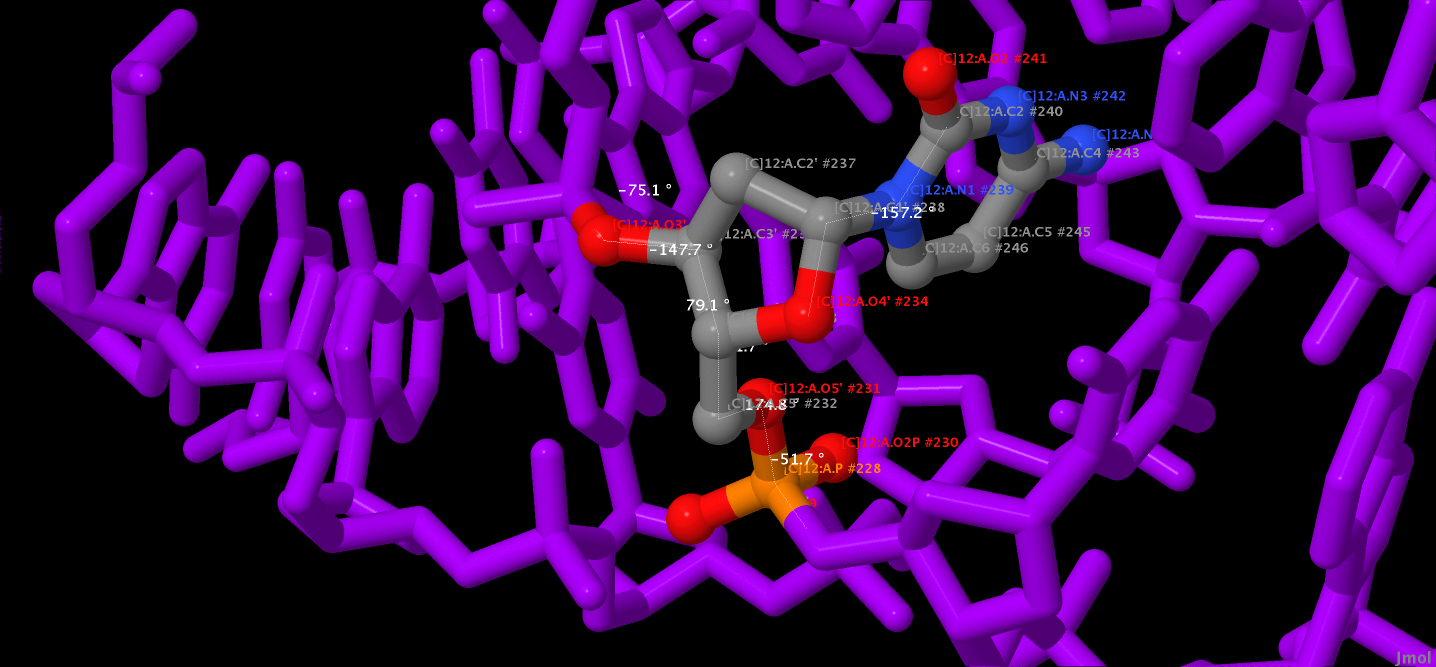 |
| Рис. 9. Торсионные углы в А-ДНК. Подписаны атомы цитозина под номером 12. |
Определение параметров структур нуклеиновых кислот с помощью программ пакета 3DNA
Упр. 1 Научиться определять торсионные углы нуклеотидов
Для 3 форм ДНК получим 3 файла с различными параметрами структуры с помощью команды:
find_pair -t gatc-*.pdb stdout | analyze, где * форма ДНК. Файлы в которых можно найти значения торсионных углов тут: A-ДНК, B-ДНК, Z-ДНК.
Далее для того, удобно было сравнивать торсионные углы создадим файл, который будет содержать их для всех трех ДНК. Наиболее сильно отличаются значения углов: alpha для всех ДНК (особенно для A c Z и B с Z), gamma для A,B с Z), delta для A и B, zeta для A и B, chi для A и B.
Так как пакет 3DNA работает только со старым форматом pdb, то для начала переведем имеющийся у нас pdb в старый (для файлов 1H4S.pdb и 1MHD.pdb):
remediator --old "XXXX.pdb" > "XXXX_old.pdb
Получим файл 1MHD_old.out и определим среднее значение каждого из торсионных углов (краевые нуклеотиды не рассматриваем); определим номер самого "деформированного" нуклеотида. Им оказывается C10 из первой цепи и A12 - из второй (рисунок 10 и 11).(с наиболее отклоняющимся значением какого-либо из торсионных углов, а лучше сразу и нескольких углов
 |
| Рис. 10. Цепь 1 ДНК. Отклонение у самого "деформированного" нуклеотида С10 выделено оранжевым цветом. Фиолетовым выделены самые отклоняющиеся торсионные углы. |
 |
| Рис. 11. Цепь 2 ДНК. Отклонение у самого "деформированного" нуклеотида A12 выделено оранжевым цветом. Фиолетовым выделены самые отклоняющиеся торсионные углы. |
Упр. 2 Научиться определять структуру водородных связей
Определим номера нуклеотидов, образующих стебли(stems) во вторичной структуре заданной тРНК. Для этого воспользуемся файлом 1H4S_old.out.
 |
| Рис. 12. Cтебели в тРНК 1H4S окрашены желтым. А нуклеотиды не входящие в стебели нуклеотиды - зеленым. |
Неканонические пары оснований в структуре тРНК показаны на рисунке 13.
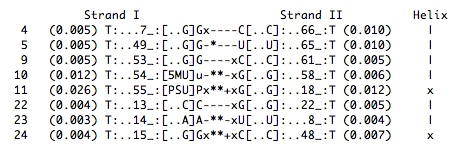 |
| Рис. 13. Неканонические пары оснований в тРНК. |
Дополнительные водородные связи в структуре тРНК показаны на рисунке 14.
 |
| Рис. 14. Дополнительные водородные связи в 1H4S. Интересно, что 5 из этих связей неканонические. |
Упр. 3 Научиться находить возможные стекинг-взаимодействия
Cтэкинг взаимодействия - это гидрофобные связи, которые возникают при таком расположении ароматических молекул, которое напоминает расположение монет в стопке и поддерживается ароматическими взаимодействиями.
Посмотрим еще раз в файл 1H4S_old.out, найдем в нем информацию о стекинг взаимодействиях, найдем с набольшей и наименьшей площадью взаимодействия (рис. 15).
 |
| Рис. 15. Стекинг взаимодействия. Зеленым подчеркнуты те, которые имеют наибольшую площадь перекрывания, а красным - наименьшую. |
Теперь получим изображения подчеркнутых стекинг взаимодействий с помощью команд:
ex_str -* stacking.pdb step*.pdb
stack2img -cdolt step*.pdb step*.ps, где * номер взаимодействия.
Ниже представлены изображения для взаимодействий с максимальной площадью перекрывания:
 |
| Стекинг взаимодействие 5 для GC/GU. |
| |
| Стекинг взаимодействие 9 для Gu/GC. |
| |
| Стекинг взаимодействие 10 для Gu/GC. |
И с минимальной:
| |
| Стекинг взаимодействие 18 для GG/CA. |
| |
| Стекинг взаимодействие 24 для GG/CC. |
На изображениях хорошо видно, что площадь перекрывания азотистых оснований в стекинг взаимодействиях 18 и 24 существенно ниже, чем в 5, 9 и 10.
Посмотрим взаимодействие 9 в JMol:
 |
| Стекинг взаимодействие 9 для Gu/GC в JMol. |
