| Главная | Семестры | Проекты | Обo мне | Ссылки | Заметки | Назад к оглавлению |
EMBOSS
1. Программа getorf пакета EMBOSS
Программа getorf извлекает из нуклеотидной последовательности открытые рамки считывания (ОРС).
Создадим в своей дирректории файл с записью D89965 банка EMBL: entret embl:D89965 -auto.
Запустим программу getorf с параметрами (командой tfm getorf ищем параметры), которые удолетворяют:
1) набор трансляций всех ОРС последовательности, которые определены при использовании стандартного кода (-table 0)
2) длина не менее 30 аминокислотных остатков (-minsize 90, 90 тк считает нуклеотиды)
3)начинаются со старт-кодона (или начала последовательности) и заканчиваются стоп-кодоном (или концом последовательности) (-find 1)
Команда: getorf d89965.entret -table 0 -minsize 90 -find 1
Получаем файл d89965.orf
Определим, какая из найденных открытых рамок соответствует (полностью или частично) приведённой в поле FT кодирующей последовательности (CDS). В EMBL: FT CDS 163-435. Ей соответствует третья ОРС:
>D89965_3 [163 - 432] Rattus norvegicus mRNA for RSS, complete cds. MALMHFQFTFKQFEQRKSIRSTARKARDDFVVVQTADLFHVAFHYGIAQRGLTITSDDHM AVTAYAYYSCHELTPWLRIQSTNPVQKYGA
Запись Swiss-Prot, на которую ссылается данная запись EMBL, полученная командой seqret sw:p0a7b8.
Выясним какой из полученных открытых рамок соответствует полученная последовательность hslv_ecoli.fasta. Для этого, чтобы не искать "глазами", запустим команду: blastp -query hslv_ecoli.fasta -subject d89965.orf -out blastp_result.out:
Query= HSLV_ECOLI P0A7B8 ATP-dependent protease subunit HslV (3.4.25.2)
(Heat shock protein HslV)
Length=176
Subject= D89965_5 [294 - 1] (REVERSE SENSE) Rattus norvegicus mRNA for RSS, complete cds.
Length=98
Score = 200 bits (509), Expect = 4e-71, Method: Compositional matrix adjust.
Identities = 98/98 (100%), Positives = 98/98 (100%), Gaps = 0/98 (0%)
Query 28 MKGNVKKVRRLYNDKVIAGFAGGTADAFTLFELFERKLEMHQGHLVKAAVELAKDWRTDR 87
MKGNVKKVRRLYNDKVIAGFAGGTADAFTLFELFERKLEMHQGHLVKAAVELAKDWRTDR
Sbjct 1 MKGNVKKVRRLYNDKVIAGFAGGTADAFTLFELFERKLEMHQGHLVKAAVELAKDWRTDR 60
Query 88 MLRKLEALLAVADETASLIITGNGDVVQPENDLIAIGS 125
MLRKLEALLAVADETASLIITGNGDVVQPENDLIAIGS
Sbjct 61 MLRKLEALLAVADETASLIITGNGDVVQPENDLIAIGS 98
Заметим, что запись D89965 банка EMBL по описанию содержит мРНК серой крысы, в то время как, запись в банке SwissProt на которую ссылаются авторы этой записи принадлежит E.coli. Интересно, как такое могло произойти? Скорее всего авторы записи в банке EMBL допустили ошибку, проводя анотацию полученной последовательности и их крыса (крысы) были заражены кишечной палочкой, что не удивительно, ведь кишечная палочка очень распространенная бактерия, не даром же ее используют как хороший модельный объект. Осталось выяснить почему в банке EMBL этот промах никак не исправлен. А не исправлен он потому, что в банке EMBL поправить запись может только ее автор, а в банке SwissProt записи проверяются и редактируются.
2. Файлы-списки
С помощью программ пакета EMBOSS:
- Скачаем в файл adh.fasta в fasta-формате все доступные в Swissprot последовательности алкогольдегидрогеназ: seqret sw:adh*_* adh.fasta.
- Получим файл с универсальными адресами (USA) этих последовательностей: infoseq adh.fasta -only -usa > adh.infoseq.
- Получим из этого файла-списка другой, меньший, с адресами только тех последовательностей, которые взяты из моих организмов: grep -f organisms.txt adh.infoseq > adh_only_my.infoseq.
- На основе adh_only_my.infoseq получим fasta-файл с последовательностями дегидрогеназ моих организмов: seqret @adh_only_my.infoseq adh_only_my.fasta
3. EnsEMBL
Портал EnsEMBL (читается "ансамбль", от французского слова "ensemble" отличается написанием, намекающим на банк EMBL) предназначен для визуализации известной информации о геномах человека и животных.
Поищем информацию о гене TM50B_HUMAN человека. Получим последовательность всего гена,кодирующего данный белок.
Поищем ген в человеческом геноме на портале EnsEMBL сервисом "BLAST/BLAT". Что получили?

Этот блок носит название "Alignment Locations vs. Karyotype", в нем находится информация о расположении гена, кодирующего выбранный траспортный белок (TM50B_HUMAN), на хромосоме. Судя по изображению, ген находится на большом плече 19 хромосомы. А вот что означают стрелочки на других хромосомах до конца не ясно, но возможно они указывают на достаточно хорошо выровненные участки, т.е. имеющие близкую последовательность с геном. "Полосатость" хромосом, скорее всего, указывает схематичное изображение экзон-интронной структуры, но тогда не понятно, почему участок кодирующий белок светло-серый.. Скорее всего это именно "схематичное" изображение.
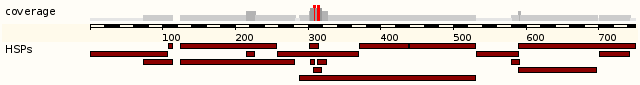
В этом разделе, назвающимся "Alignment Locations vs. Query" приведена информация о самом выравнивании, и видно, что на участке 300 наблюдается высокий уровень консервативности. HSP - это high-scoring segment pair, поэтому получается, что попарное выравнивание приведено для различных, наиболее "удачных", близких по последовательности участков. Это подтвердается следущим изображением.

На этом изображении, которое находится в последнем разделе "Alignment Summary" приведена в табличном виде информация о выравнивании гена с различными хромосомами. В этой таблице можно наверху выбирать интересующие строки, и тем самым редактировать под свои нужды таблицу. Какую "различную" информацию можно узнать из этой таблицы?
- номер запроса
- ссылку G (Genome Sequence) на последовательность выровненого участка хромосомы, где можно выбирать самые различные параметры: вланкирующие области (Flanking sequence) на 5' и 3' концах, координатную схему (хромосомную (даже четыре раза?), клон, контиг, суперконтиг), ориентацию (по выравниванию вперед, по координатной схеме), разметку (по различным типам экзонов) и другие.
- ссылку С "Contig view", чтобы посмотреть хромосому, ее последовательность, информацию о расположенных генах (по GENCODE)
- можно пройти также по ссылке A(Alignment) на само выравнивание (например, для 19 хромосомы, на которой, как определила программа, находится ген)
- непосредственно номер хромосомы про которую мы смотрим информацию
- старт и конец выравнивания, его длину
- наличие ориджина репликации
- e-value
- процент идентичности
- а так же можно посмотреть p-value - вероятность ошибки при отклонении нулевой гипотезы (статистическая величина)
Посмотрим более подробно, что мы можешь увидеть по ссылке С "Contig view".


Чего только нельзя! Эта ссылка позволяет нам смотреть на хромосому с различным приближением (буквально). Мы можем:
- перемещаться по ней
- смотреть разметку по генам (даже псевдогены отмечены), экзонам, интронам
- сохранять изображения с различными данными в нужном формате
- приближать и удалять изображения, нажимая правой кнопкой на изображение
- нажимать на гены и смотреть информацию о них
- смотреть насположени промоторов(изображение 3)
- можно даже поделиться страницей
- и нажать наверху экрана ссылку help, чтобы посмотреть еще какие-нибудь интересные "фишки" сайта.
