| Главная | Семестры | Проекты | Обo мне | Ссылки | Заметки | Назад к оглавлению |
Филогенетические деревья
Выберем 7 бактерий из отдела Firmicutes:
- Название - Мнемоника
- Clostridium tetani - CLOTE
- Clostridium botulinum - CLOB1
- Finegoldia magna - FINM2
- Bacillus anthracis - BACAN
- Bacillus subtilis - BACSU
- Listeria monocytogenes - LISMO
- Staphylococcus aureus - STAA1
- Staphylococcus epidermidis - STAES
Изображение таксономического дерева:
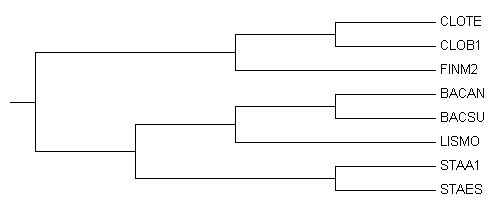 |
| Рис. 1. |
Определим, к каким таксонам относятся отобранные бактерии с помощью сервиса NCBI: http://www.ncbi.nlm.nih.gov/taxonomy/:
 |
| Рис. 2. |
Выводы:
Как видно из рисунка 2, отдел Firmicutes делится на 2 порядка (в составе класса Bacilli) Bacillales и Clostridiales (в составе класса Clostridia):
- В порядке Bacillales есть:
- род Staphylococcus (Семейство: Staphylococcaceae):
- вид Staphylococcus epidermidis
- вид Staphylococcus aureus
- вид Listeria monocytogenes (Семейство: Listeriacea, род: Listeria)
- род Bacillus (Семейство: Bacillaceae):
- вид Bacillus subtilis
- вид Bacillus anthracis
- В порядке Clostridiales есть:
- род Clostridium (Семейство: Clostridiaceae):
- вид Clostridium tetani
- вид Clostridium botulinum
- вид Finegoldia magna
Будем реконструировать филогенетическое дерево по белкам, выполняющим функцию Фактор инициации трансляции 2; их мнемоника: IF2
Для этого на сайте Uniprot вводим запрос: (mnemonic:if2_clote OR mnemonic:if2_clob1 OR mnemonic:if2_finm2 OR mnemonic:if2_bacan OR mnemonic:if2_bacsu OR mnemonic:if2_lismo OR mnemonic:if2_staa1 OR mnemonic:if2_staes) AND reviewed:yes
Получаем последовательности интересующих белков: файл
Выравниваем в JalView с помощь Web Service -> Muscle
Получаем:
И получаем выравнивание в .fasta формате - файл
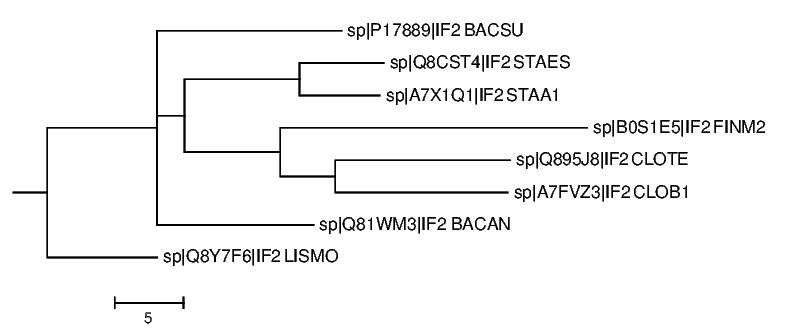
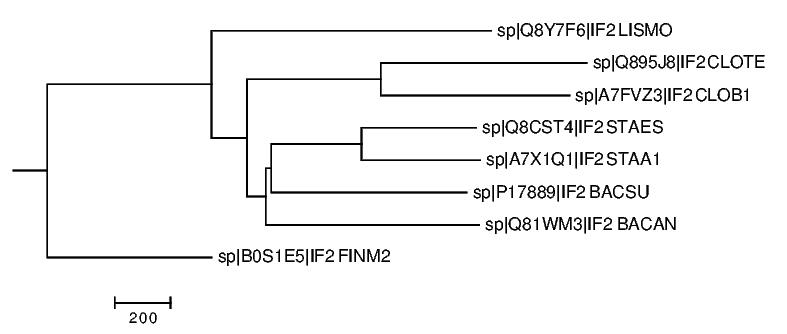
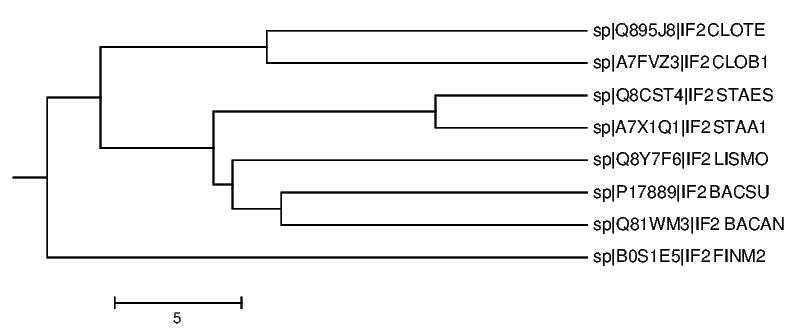
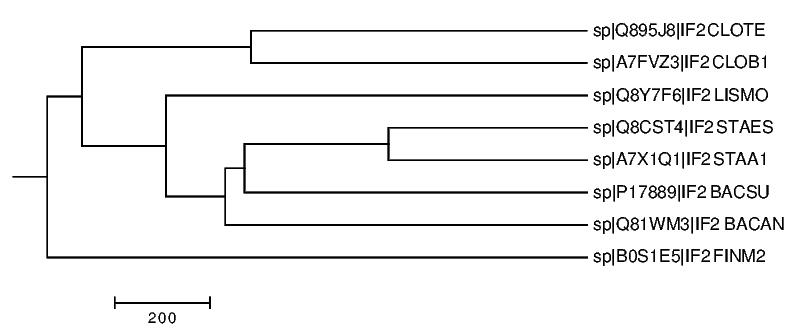
Посчитаем в JalView деревья четырьмя возможными способами,построим их с помощью программы Mega и сравним с "правильным" деревом (рис. 1):
- Neighbour joing using % identity
 vs
vs
- Neighbour joing using BLOSUM62
 vs
vs
- Average distance using % identity
 vs
vs
- Average distance using BLOSUM62
 vs
vs
Полученное выравнивание в .fasta формате импортируем в программу Mega, реконструируем дерево методом "Maximum Parsimony" и укореним в наиболее правильную ветвь (меню Subtree -> Root):

Полученное дерево полностью совпадает с "правильным" деревом на рис. 1.