| Главная | Семестры | Проекты | Обo мне | Ссылки | Заметки | Назад к оглавлению |
Реконструкция деревьев по нуклеотидным последовательностям. Деревья. содержащие паралоги.
1. Построение дерева по нуклеотидным последовательностям
Требовалось построить филогенетическое дерево, используя последовательности 16S рРНК для некоторых бактерий.
| Название | Мнемоника | AC 16S рРНК | координаты | цепь |
| Clostridium tetani | CLOTE | AE015927 | 176113..177621 | прямая |
| Clostridium botulinum | CLOB1 | CP000726.1 | 48468..49969 | прямая |
| Finegoldia magna | FINM2 | AP008971 | 197837..199361 | прямая |
| Bacillus anthracis | BACAN | AE016879 | 29129..30635 | прямая |
| Bacillus subtilis | BACSU | AL009126 | 30279..31832 | прямая |
| Listeria monocytogenes | LISMO | CP007492.1 | 242732..244284 | прямая |
| Staphylococcus aureus | STAA1 | CP003808 | 540419..542060 | прямая |
| Staphylococcus epidermidis | STAES | AE015929 | 1598006..1599559 | обратная |
Все последовательности лежат тут. Выравнивались эти последовательности в программе Jalview алгоритмом Muscle. Методом Neigbour joining пакета MEGA было реконструировано дерево.
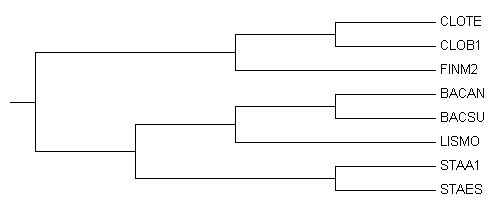
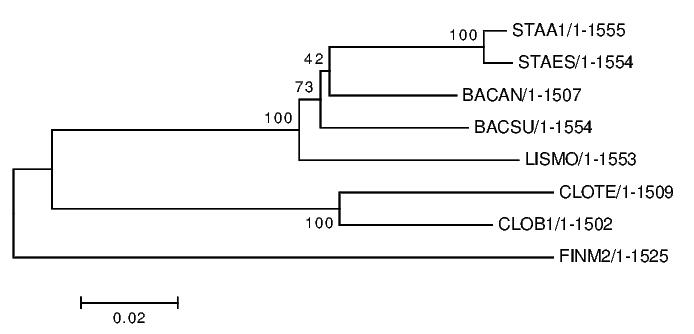
| Правильное дерево | Полученное дерево |
 |
 |
Для того, чтобы сравнить деревья сделали таблицу:
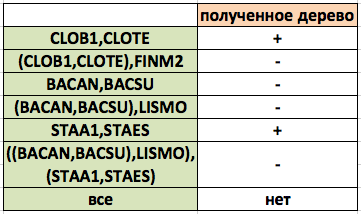
Как видно из изображения нового дерева и таблицы, основная проблема при построении дерева была в ветви ((STAA1,STAES),BACAN), неверность данного построения и подтверждается бутстреп анализом (подкрепление данной ветви мало).
2. Построение и анализ дерева, содержащего паралоги
В задание требовалось найдите в своих бактериях достоверные гомологи белка CLPX_BACSU. Построить дерево этих гомологов. Считая дерево реконструированным верно, указать несколько пар ортологов и несколько пар паралогов. Привести примеры отражённых на дереве эволюционных событий двух типов: 1) дупликация гена; 2) разделение путей эволюции белков в результате видообразования.
Чтобы найти гомологов в заданных организмах, воспользовались файлом proteo.fasta, где лежат записи банка UNIPROT, относящиеся к выбранным бактериям. Провели поиск гомологов программой blastp(с порогом на E-value 0,001) и отобрали по мнемонике видов только те находки, которые относятся к отобранным ранее бактериям.
Получили файл homologs.fasta с 37 гомологами белка CLPX_BACSU. Затем получили файл homolog-ali.fasta, выровнив последовательности командой:
muscle -in homologs.fasta -out homolog-ali.fasta.
Произведем реконструкцию дерева в программе MEGA методом Neighbour Joining с использованием метода Bootstrap для большей наглядности. Полученное дерево:
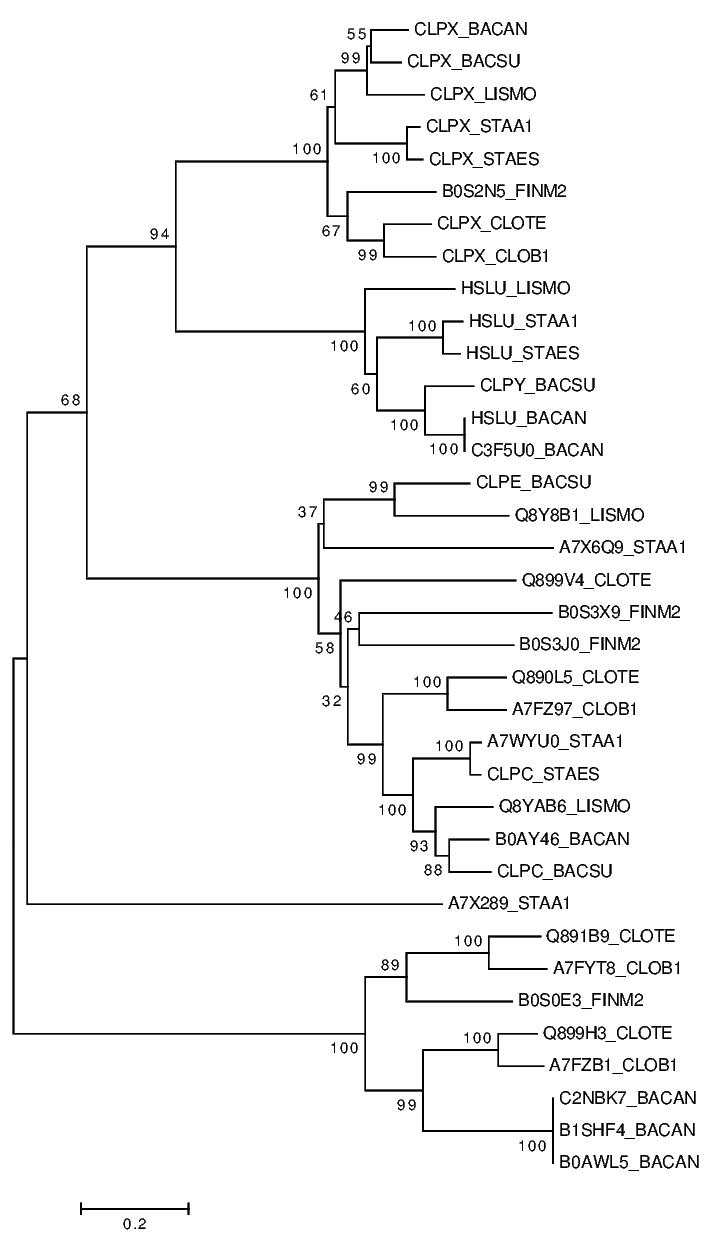 |
Будем считать:
Ортологи а) из разных организмов; б) разделение их общего предка на линии, ведущие к ним, произошло в результате видообразования.
Паралоги гомологичные белки из одного организма.
Примеры:
Ортологи: HSLU у STAA1, STAES, LISMO и BACAN
Паралоги: C2NBK7, B1SHF4 и B0AWL5 у BACAN
Отраженные на дереве эволюционные события:
дупликация гена: HSLU и C3F5U0 у BACAN
разделение путей эволюции белков в результате видообразования: Q890L5_CLOTE и A7FZ97_CLOB1
