| Главная | Семестры | Проекты | Обo мне | Ссылки | Заметки | Назад к оглавлению |
Восстановление кристалла из PDB файла
Описание контактов молекул в структуре 3TW2
В работе использовалась структура белка 1 гистидиновой триады (3TW2). Были получены кристаллографические характеристики из записи PDB (поле CRYST1):
| Параметр | Значение |
|---|---|
| a | 77.4213 A |
| b | 46.4489 A |
| c | 64.0341 A |
| alpha | 90.0000 |
| beta | 94.4227 |
| gamma | 90.0000 |
| кристаллографическая группа | C 1 2 1 |
| число молекул в ячейке | 2 (1 глобула из двух цепей) |
Кристаллографическая группа говорит о том, что наш белок 3TW2 находится в элементарной ячейке, у которой углы alpa и gamma равны 90, а beta - нет, с примитивной решёткой Бравэ. Далее было получено изображение части кристалла — содержимого ячейки из PDB файла и ряда соседних ячеек (рис. 1). Комманда используемая:
symexp sym, 3tw2, chain b, 5
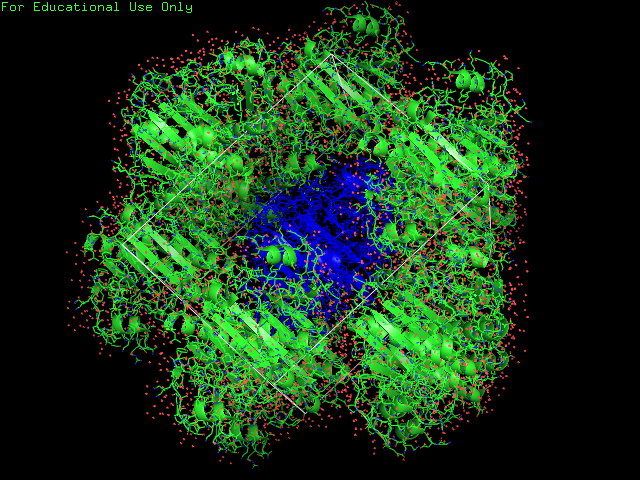 |
| Рис. 1. Молекула белка 1tw2 в элементарной ячейке с соседними белковыми молекулами. Соседствует 10 молекул. |
На рисунке 1 можно увидеть 10 соседствущих молекул белка, но не все из них взаимодействуют с нашим белком в ячейке, если считать минимальным расстоянием взаимодействия 3.5 А (рис. 2). Для того, чтобы найти контакты с белками из соседних ячеек уберем молекулы растворителя (чтобы не мешали). И рассмотрим возможные контакты на расстоянии 3.5 А. Число разных зон контакта между белками из соседних ячеек около 25. Используемые комманды:
remove solvent
show sticks, byres (prot_name and (neib-prot_name* around 3.5))
show sticks, byres (neib-prot_name* and (prot_name around 3.5))
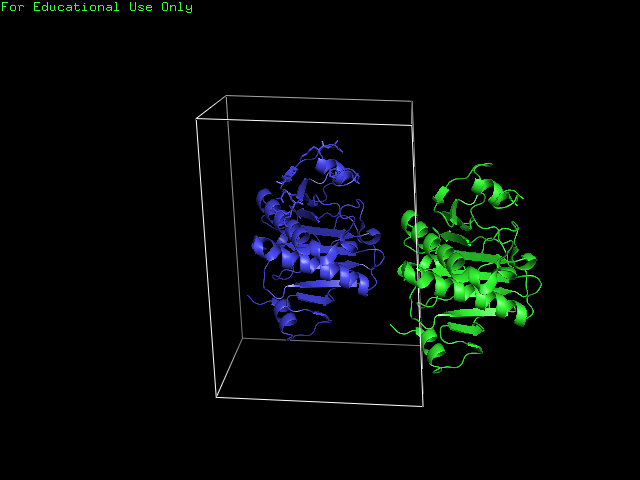 |
| Рис. 2. Пример молекулы белка, которая не взаимодействует с белком, хотя и попадает в элементарную ячейку. |
Переименуем для удобства контактирующие структуры:
set_name old_name, new_name
Построим поверхности контактов и водородные контакты структур на расстоянии менее 3.5 А (рис. 3):
sel surface*, prot_name within 3.5 of neib-prot_name*
distance hbonds, prot_name, neib-prot_name*, 3.5, mode=2
set surface_color, yellow, prot_name
set surface_color, cyan, neib-prot_name*
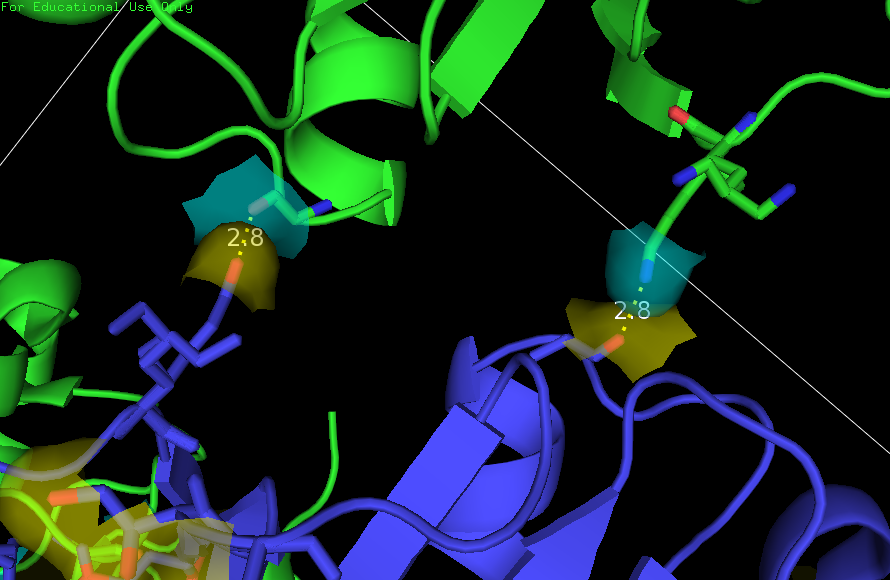 |
 |
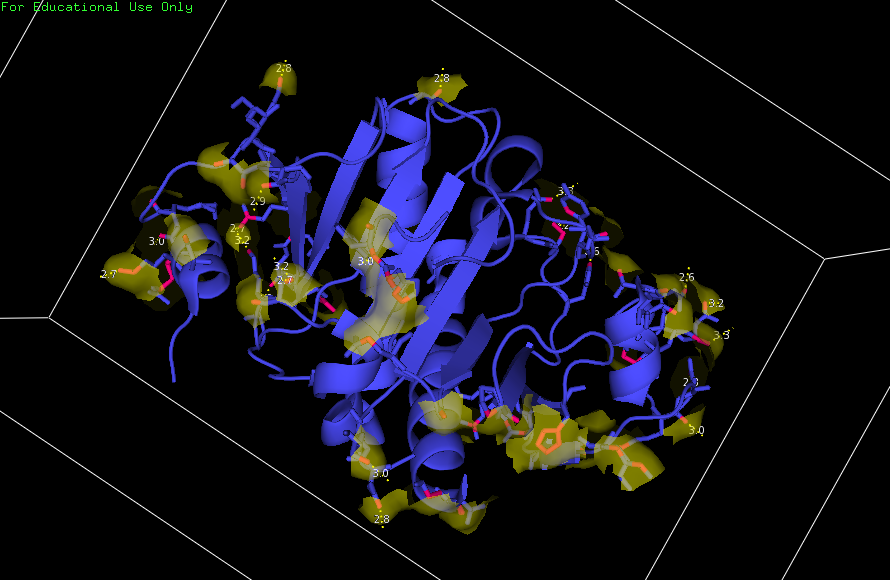 |
| Рис. 3. Поверхности контактов и водородные связи на расстоянии 3.5 А. |
Глобула белка взаимодействует с большим количеством соседствующих молекул (6), таким образом получается большая площадь поверхности контантакта (на расстоянии 3.5 А). Эти контакты, образованные в том числе водородными связями, настолько велики, что определяет возможность формрования кристалла. Данная модель хорошо описывает взаимодействия субъединиц белка в димере, таким образом сформирована глобула. Но другие контакты не описывают природные взаимодействия белков при дальнейшей олигомеризации в растворе, так как они выглядят неестественно.
Описание контактов в cтруктуре EHDD (Engrailed homeodomain DNA complex)
На сервере EDS нет карты электронной плотности, поэтому будем использовать построенную модель (рис. 4). Эта модель представляет собой комплекс из двух ДНК-связывающих белков "на краю" ДНК.
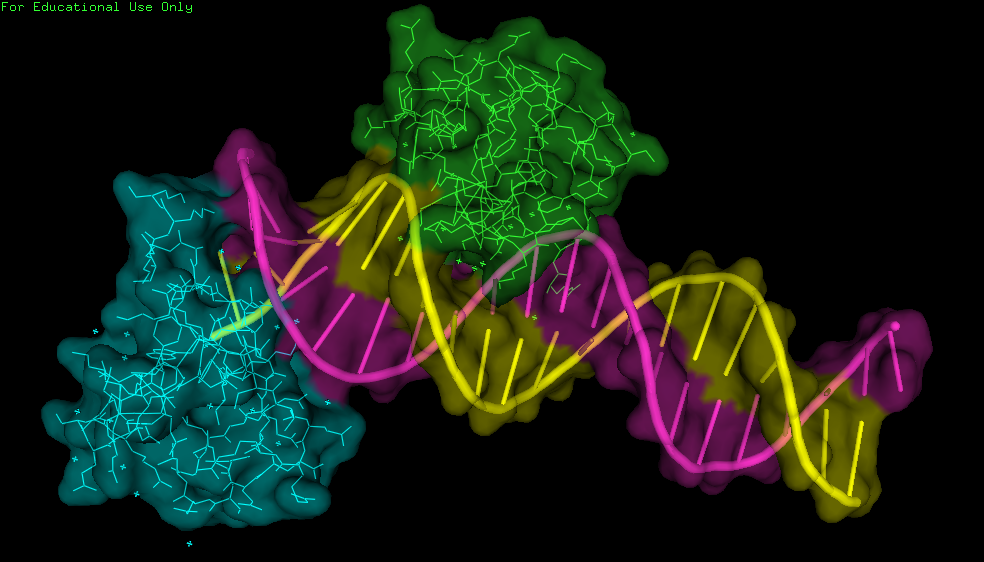 |
| Рис. 4. Структура 3HDD, построенная с помощью PyMol. |
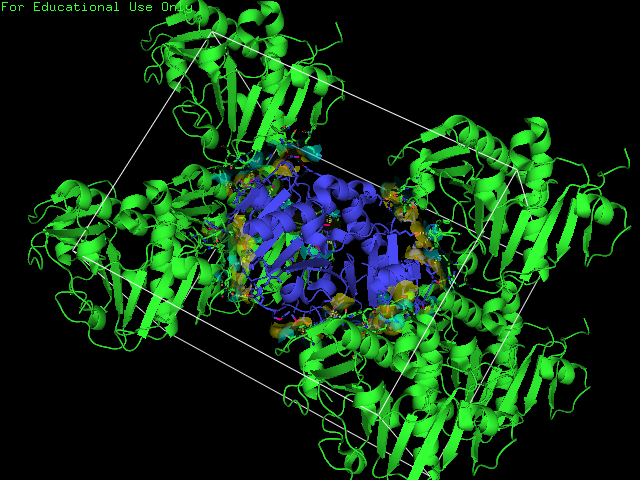
В данной структуре странны направленные в пустоту остатки голубого белка (цепь B). Попробуем разобраться. Используя тот же подход, что и раньше восстановим соседние с ним ячейки (рис. 5) и опишем контакты.
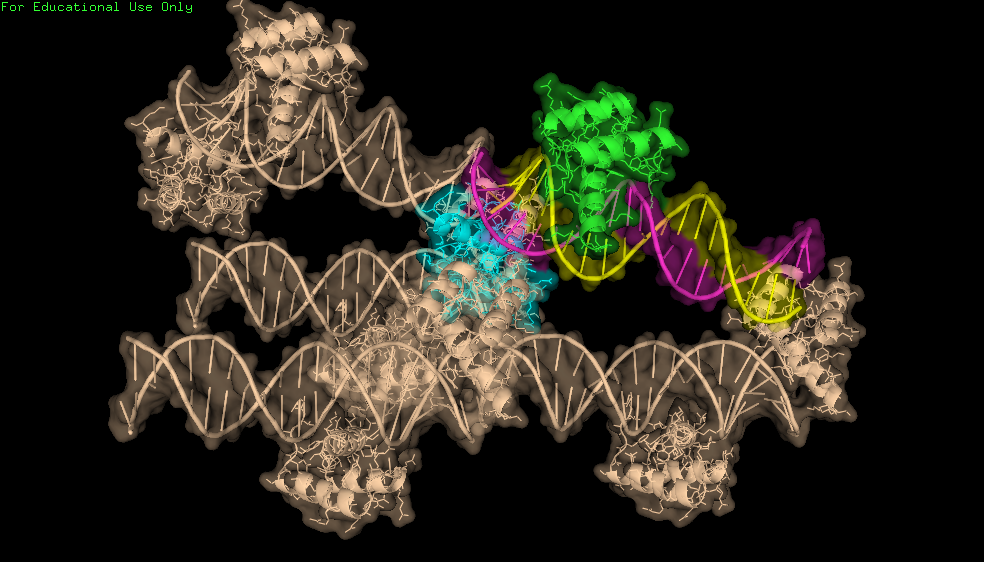 |
| Рис. 5. Структура 3HDD с восстановленными к цепи B структурами на расстоянии 5 ангстремов. |
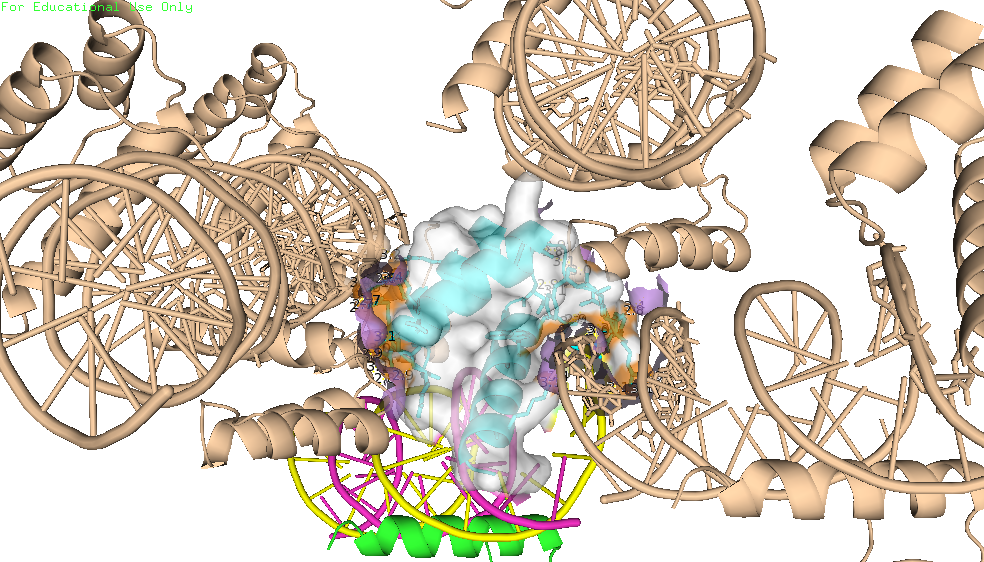
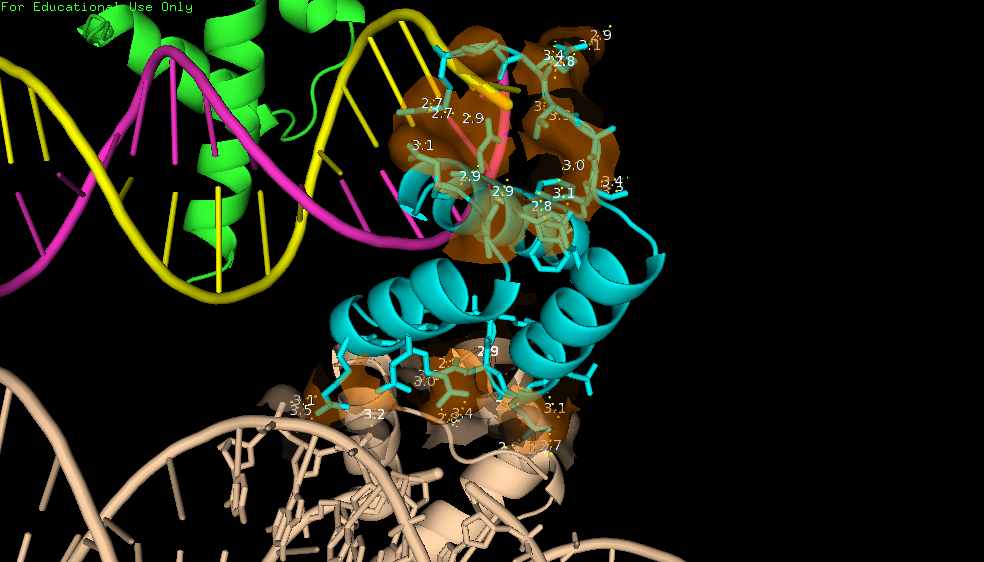
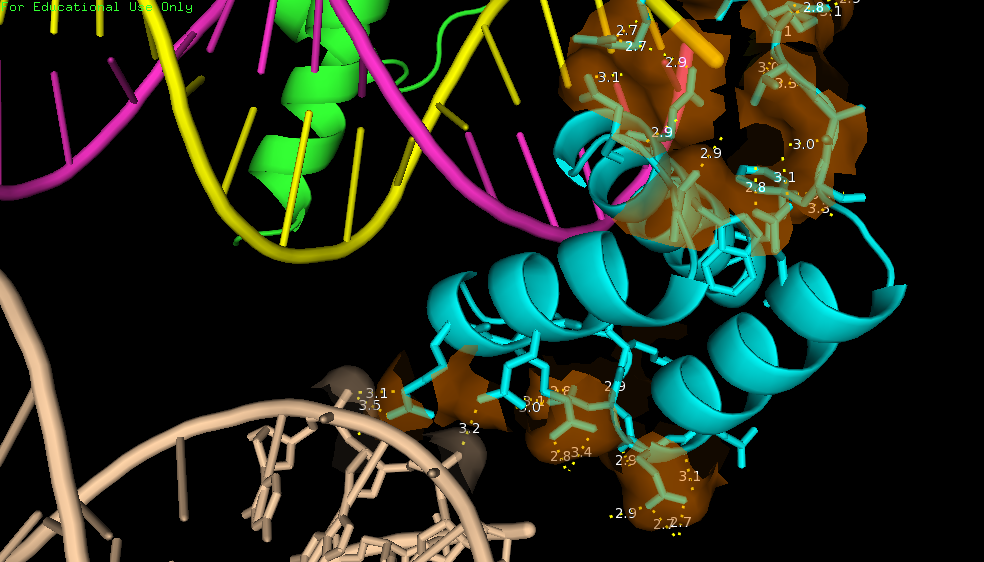
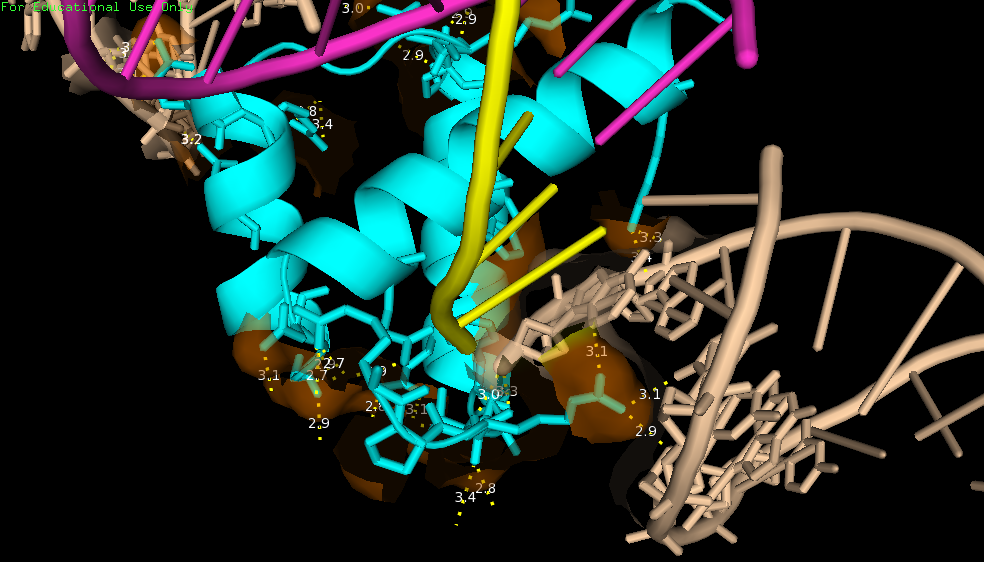
Теперь посмотрим на контакты (рис. 6). Как видно на рисунке цепь В (голубая) контактирует с 3 молекулами ДНК и 2 цепями В. Области контакта для цепи В из исходной модели покрашены оранжевым, а поверхности контактирующих молекул - фиолеотым.
 |
 |
 |
 |
 |
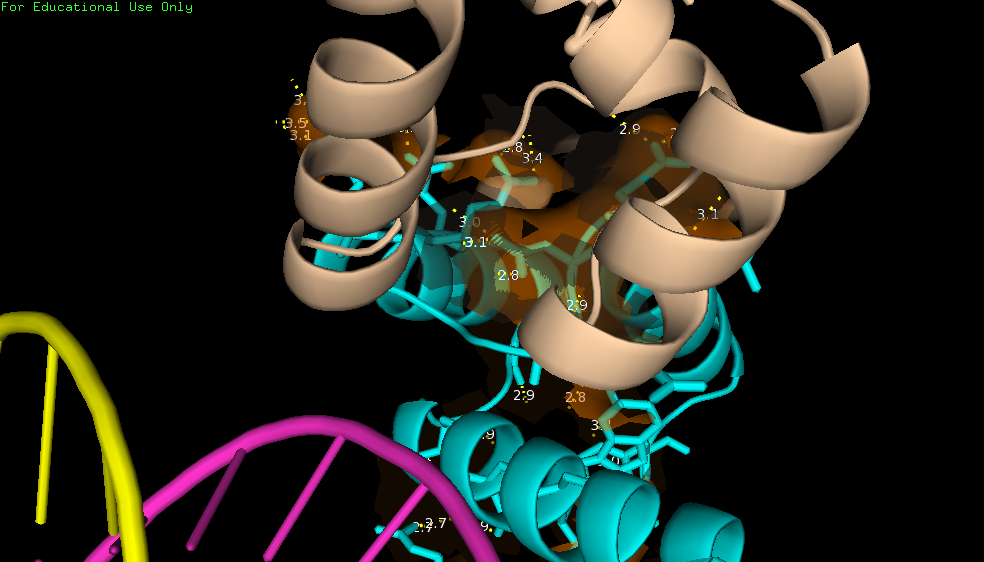 |
| Рис. 6. Места контактов цепи B c 3 молекулами ДНК и двумя другим цепями B. |
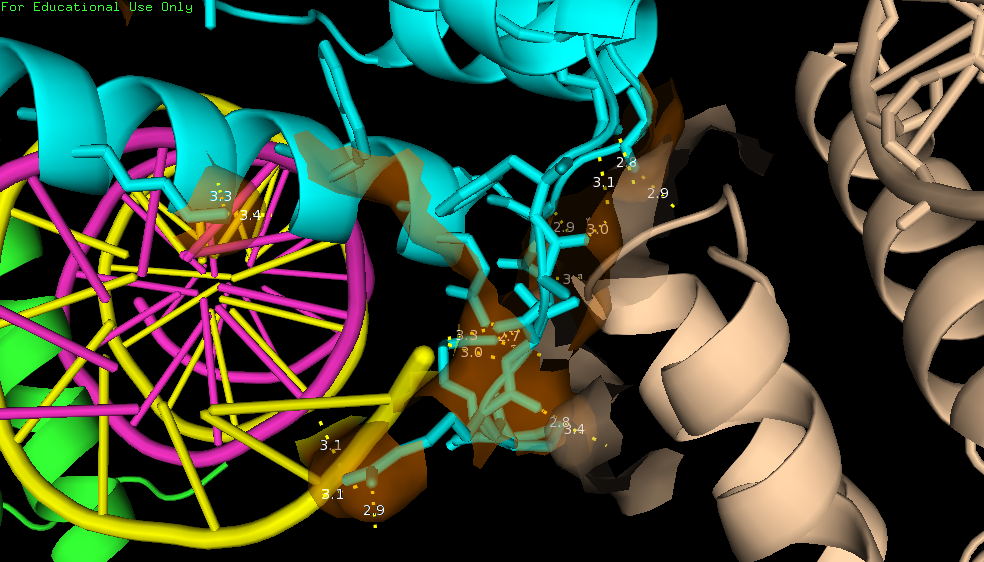
Таким образом, мы видим массу водородных связей между белковой цепью В и ДНК (рис. 6.3 и 6.4), а также с другими цепями В (рис. 6.5 и 6.6). Кроме остатков, способных образовать водородные связи в контактирующей области находится и явно гидрофобный остаток - триптофан (рис. 7). Он, как видно на рисунке, контактирует с сахарофосфатным остовом.
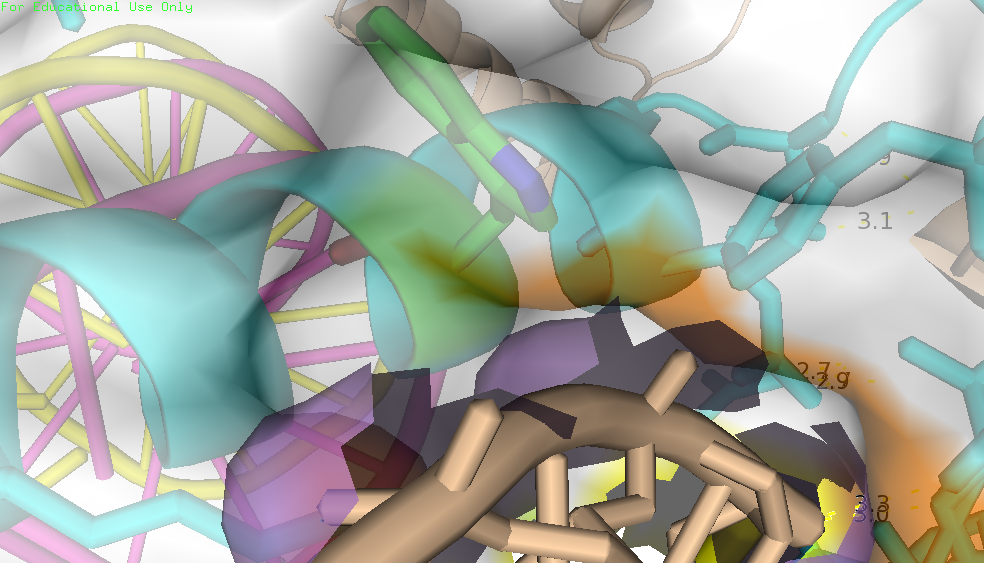 |
| Рис. 7. Область, где триптофан из цепи В контактирует с сахарофосфатным остовом ДНК из соседней ячейки. |
Из всего выше показанного и сказанного можно сделать вывод, что в данной структуре белок прочно взаимодействует, как с ДНК из своей ячейки, так и с ДНК из других ячеек, кроме того он имеет контакты и с цепями В из других ячеек. Последнее говорит о том, что возможно белок работает в виде ди- или даже тримера. Так же можно обратить внимание, что в областях контактов преобладают положительно заряженные аминокислоты, такие как аргинин и лизин, хотя есть и другие гидрофильные аминокислоты. Преобладание положительно заряженных аминокислот логично, ведь ДНК заряжена отрицательно, и ДНК-связывающему белку выгодно иметь в местах связывания с ней положительные аминокислоты для улучшения связывания. Этот белок имеет и типичную для ДНК-связывающих белков спираль, которая укладывается в большую бороздку ДНК.
Используемые команды:
show surface util.cbc set light_count, 1 select dna, chain c or chain d select cha, chain a select chb, chain b symexp sym, 3hdd, chain B, 5 color wheat, sym* remove solvent show sticks, byres (chain b and (sym* around 3.5)) show sticks, byres (sym* and (chain b around 3.5)) sel surface*, chb within 3.5 of sym* sel surface2, sym* within 3.5 of chb distance hbonds, chb, sym*, 3.5, mode=2 set surface_color, arsenic, sym* set surface_color, orange, chb set transparency, 0.5 hide cartoon, sym*
