На главную
Классификация функций. Ферменты.
IUPAC номенклатура ферментов
Код фермента:
EC 4.3.2.2
Расшифровка каждого пункта:
EC 4 - Lyases
EC 4.3 - Carbon-Oxygen Lyases
EC 4.3.2 - Lyases acting on Amides, Amidines, etc.
EC 4.3.2.2 - Adenylosuccinate lyase
Характеристики аденил-сукцинат лиазы (EC 4.3.2.2), предоставляемые
специализированным ресурсом Brenda
Ферментативная реакция, катализируемая ферментом:
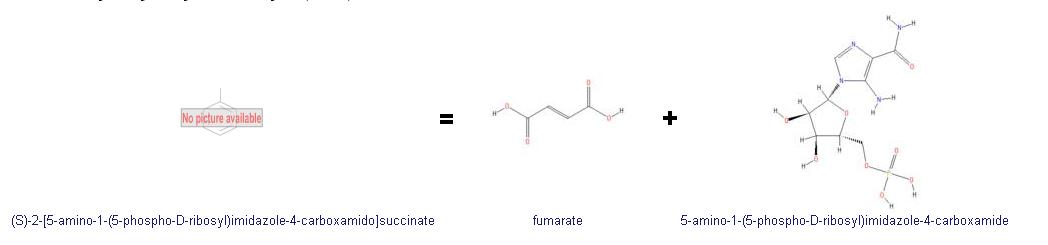
Общее число ферментов с кодом 4.3.2.2:
10
Ингибиторы:
Аденилэтилфосфанат, аденилсукцинад, АДФ, АМФ, ЭДТА, Фумарат, KCl,....
ЭДТА
 |
АДФ
 |
Активаторы:
Нет данных
Оптимум pH:
Нет данных
Сравнение ферментов с
одинаковым кодом из эволюционно далеких организмов, идентификатор PFam PF00206
| ID фермента в Swiss-Prot |
Начало домена |
> Конец домена |
| PUR8_ECOLI |
11 |
305 |
| PUR8_METJA |
12 |
342 |
| PUR8_HUMAN |
13 |
307 |
С помощью программы seqret пакета EMBOSS были получены аминокислотные
последовательности доменов.
Использованные команды:
seqret sw:P30566 -sask
seqret sw:Q25739 -sask
seqret sw:P25739 -sask
Команда -sask позволяет задать границы доменов в ручную.
Выравнивание полученных последовательностей было построено с помощью
программы needle командами
needle pur8_ecol6.fasta pur8_human.fasta -auto
needle pur8_ecol6.fasta pur8_metja.fasta -auto
needle pur8_human.fasta pur8_metja.fasta -auto
Полученны выравнивания:
########################################
# Program: needle
# Rundate: Fri May 26 2006 23:24:09
# Align_format: srspair
# Report_file: pur8_ecol6.needle
########################################
#=======================================
#
# Aligned_sequences: 2
# 1: PUR8_ECOL6
# 2: PUR8_HUMAN
# Matrix: EBLOSUM62
# Gap_penalty: 10.0
# Extend_penalty: 0.5
#
# Length: 332
# Identity: 72/332 (21.7%)
# Similarity: 127/332 (38.3%)
# Gaps: 74/332 (22.3%)
# Score: 142.5
#
#
#=======================================
PUR8_ECOL6 1 ---PVDGRYGDKVSALRGIFSEYGLLKFRVQVEVRWL------QKLAAHA 41
|:..||....... :||: ..|||...:: || |.|....
PUR8_HUMAN 1 YRSPLASRYASPEMCF--VFSD--RYKFRTWRQL-WLWLAEAEQTLGLPI 45
PUR8_ECOL6 42 AIKEVPAFAADAIGYLDAIVASFSEEDAARIKTIERTTNHDVKA-VEYF- 89
..:::....:: |:.|....:.|:..|:: |||.| |..|
PUR8_HUMAN 46 TDEQIQEMKSN----LENIDFKMAAEEEKRLR-------HDVMAHVHTFG 84
PUR8_ECOL6 90 -LKEKVAEIPELHAVSEFIHFACTSEDINNLSHALMLKTARDEVILPYWR 138
...|.|.| ||...||..:.:.:..::|:.|.| ::||...
PUR8_HUMAN 85 HCCPKAAGI---------IHLGATSCYVGDNTDLIILRNALD-LLLPKLA 124
PUR8_ECOL6 139 QLIDGIKDLAVQYRDIPLLSRTHGQPATPSTIGKE----MANVAYRMERQ 184
::|..:.|.|.:...:|.|..||.|||..:|:||. :.::...::..
PUR8_HUMAN 125 RVISRLADFAKERASLPTLGFTHFQPAQLTTVGKRCCLWIQDLCMDLQNL 174
PUR8_ECOL6 185 YRQLNQVEILGKINGAVGNYNAHIAAYPEVDWHQ-------------FSE 221
.|..:.:...| :.|..|...:.:..: |.|.|: |..
PUR8_HUMAN 175 KRVRDDLRFRG-VKGTTGTQASFLQLF-EGDDHKVEQLDKMVTEKAGFKR 222
PUR8_ECOL6 222 EFVTSLGIQWNPYTTQIEPHDYIAELFDCVARFNTILIDFDRDVWGYIAL 271
.|: |....||.::: .|:...:|.....:.....|:.....|
PUR8_HUMAN 223 AFI----ITGQTYTRKVD-----IEVLSVLASLGASVHKICTDIRLLANL 263
PUR8_ECOL6 272 NHFKQKTIAGEIGSSTMPHKVNPI-------- 295
...::.....:||||.||:|.||:
PUR8_HUMAN 264 KEMEEPFEKQQIGSSAMPYKRNPMRSERCCSL 295
#---------------------------------------
#---------------------------------------
########################################
# Program: needle
# Rundate: Fri May 26 2006 23:25:25
# Align_format: srspair
# Report_file: pur8_metja.needle
########################################
#=======================================
#
# Aligned_sequences: 2
# 1: PUR8_METJA
# 2: PUR8_ECOL6
# Matrix: EBLOSUM62
# Gap_penalty: 10.0
# Extend_penalty: 0.5
#
# Length: 360
# Identity: 97/360 (26.9%)
# Similarity: 146/360 (40.6%)
# Gaps: 94/360 (26.1%)
# Score: 257.5
#
#
#=======================================
PUR8_METJA 1 KMAVHPIDYRYG--TPEMRKVWEEENKLEKMLKVEAA-LAK--AQAELGL 45
|:|.||| ...:|.::.|...|:..::||.. |.| |.|.:..
PUR8_ECOL6 1 -----PVDGRYGDKVSALRGIFSEYGLLKFRVQVEVRWLQKLAAHAAIKE 45
PUR8_METJA 46 IPKEAAEEINK----KASTKYVKLERVKEIEKQTKHDVVA----MIRALA 87
:|..||:.|.. .||.......|:|.||:.|.|||.| :...:|
PUR8_ECOL6 46 VPAFAADAIGYLDAIVASFSEEDAARIKTIERTTNHDVKAVEYFLKEKVA 95
PUR8_METJA 88 EVCEGNA-GEYIHFGATSNDIVDTANSLLIKES-IEIIEDKLKQLRDILL 135
|:.|.:| .|:|||..||.||.:.:::|::|.: .|:|....:||.|.:.
PUR8_ECOL6 96 EIPELHAVSEFIHFACTSEDINNLSHALMLKTARDEVILPYWRQLIDGIK 145
PUR8_METJA 136 DKAEEHKYTVCVGRTHGQHAIPTTYGMRFALWAAEIDRHLERLKEAKKRI 185
|.|.:::....:.|||||.|.|:|.|...|..|..::|...:|.:.:
PUR8_ECOL6 146 DLAVQYRDIPLLSRTHGQPATPSTIGKEMANVAYRMERQYRQLNQVE--- 192
PUR8_METJA 186 CVSMITGAVGT----MAAMGEKGLEVHKRVAEI---LGLEPVLISNQVIQ 228
.:..|.||||. :||..| ::.|:...|. ||::....:.|:..
PUR8_ECOL6 193 ILGKINGAVGNYNAHIAAYPE--VDWHQFSEEFVTSLGIQWNPYTTQIEP 240
PUR8_METJA 229 RDRHAEFVFLLALIAQTLNKIGVTV-------RSMQRTEIGELEEEFDPT 271
.|..||....:|.....|......| ...|:|..||:
PUR8_ECOL6 241 HDYIAELFDCVARFNTILIDFDRDVWGYIALNHFKQKTIAGEI------- 283
PUR8_METJA 272 KQTGSSTMPHKRNPITFEQICGLSRVIKSLCIAEMDNIPLWEERDLTNSS 321
||||||||.|||
PUR8_ECOL6 284 ---GSSTMPHKVNPI----------------------------------- 295
PUR8_METJA 322 AERCIFAEVC 331
PUR8_ECOL6 296 ---------- 295
########################################
# Program: needle
# Rundate: Fri May 26 2006 23:48:36
# Align_format: srspair
# Report_file: pur8_metja.needle
########################################
#=======================================
#
# Aligned_sequences: 2
# 1: PUR8_METJA
# 2: PUR8_HUMAN
# Matrix: EBLOSUM62
# Gap_penalty: 10.0
# Extend_penalty: 0.5
#
# Length: 340
# Identity: 90/340 (26.5%)
# Similarity: 151/340 (44.4%)
# Gaps: 54/340 (15.9%)
# Score: 323.0
#
#
#=======================================
PUR8_METJA 1 KMAVHPIDYRYGTPEMRKVWEEENKLEKMLKVEAALAKAQAELGL-IPKE 49
...|:..||.:|||..|:.:..|.....::...||:|:..||| |..|
PUR8_HUMAN 1 --YRSPLASRYASPEMCFVFSDRYKFRTWRQLWLWLAEAEQTLGLPITDE 48
PUR8_METJA 50 AAEEINKKASTKYVKLERVKEIEKQTKHDVVAMIRALAEVCEGNAGEYIH 99
..:|: |::.:.:..:...|.||:.:|||:|.:......|...|| .||
PUR8_HUMAN 49 QIQEM--KSNLENIDFKMAAEEEKRLRHDVMAHVHTFGHCCPKAAG-IIH 95
PUR8_METJA 100 FGATSNDIVDTANSLLIKESIEIIEDKLKQLRDILLDKAEEHKYTVCVGR 149
.||||..:.|..:.::::.:::::..||.::...|.|.|:|......:|.
PUR8_HUMAN 96 LGATSCYVGDNTDLIILRNALDLLLPKLARVISRLADFAKERASLPTLGF 145
PUR8_METJA 150 THGQHAIPTTYGMRFALWAAEIDRHLERLKEAKKRICVSMITGAVGTMAA 199
||.|.|..||.|.|..||..::...|:.||..:..:....:.|..||.|:
PUR8_HUMAN 146 THFQPAQLTTVGKRCCLWIQDLCMDLQNLKRVRDDLRFRGVKGTTGTQAS 195
PUR8_METJA 200 M-------GEKGLEVHKRVAEILGLE-PVLISNQVIQRDRHAEFVFLLAL 241
. ..|..::.|.|.|..|.: ..:|:.|...|....|.:.:||.
PUR8_HUMAN 196 FLQLFEGDDHKVEQLDKMVTEKAGFKRAFIITGQTYTRKVDIEVLSVLAS 245
PUR8_METJA 242 IAQTLNKIGVTVRSMQRTEIGELEEEFDPTKQTGSSTMPHKRNPITFEQI 291
:..:::||...:|.: ..:.|:||.|: .:|.|||.||:||||:..|:.
PUR8_HUMAN 246 LGASVHKICTDIRLL--ANLKEMEEPFE-KQQIGSSAMPYKRNPMRSERC 292
PUR8_METJA 292 CGLSRVIKSLCIAEMDNIPLWEERDLTNSSAERCIFAEVC 331
|.|
PUR8_HUMAN 293 CSL------------------------------------- 295
#---------------------------------------
#---------------------------------------
Идентичность белков колеблеться от 20 да 29 процентов.
Это говорит о том,
что вероятность совпадения функций этих ферментов достаточно велика.
©
Шпильман Алексей, 2004




