На главную
Исследование филогенетического дерева семейства глобинов из Strepsirhini и Anguilliformes
Составление выборки белков семейства
С помощью SRS в базе данных UniProt были найдены аминокислотные
последовательности, нужные для составления выбороки белков
семейства глобинов (иденитфикатор Pfam PF00042), состоящей из трёх частей:
- белки семейства глобинов из таксонов Strepsirhini и Anguilliformes
- глобины человека — Q8WWM9, P09105, P02008, P69905, P68871, P02042,
P02100, P69891, P69892, Q9NPG2;
- внешняя группа (outgroup) — P02144, P02202, Q9DGJ1.
Аминокислотные последовательности находятся в файле
seq.txt
Перечнень организмов выборки находится в файле
taxon_names.txt
Построение филогенетического дерева
Программой emma было построено множественное вызавнивание аминокислотных
последовательностей, соответствующих выборке. С помощью программы eprotdist
была получена матрица попарных расстояний, которая было использована
программой eneighbor, чтобы реконструировать
филогенетическое дерево по
алгоритму ближайших соседей (Neighbour-Joining). Редактировалось дерево в
программе GeneMaster.
Текстовый файл находится здесь.
Изображение дерева:
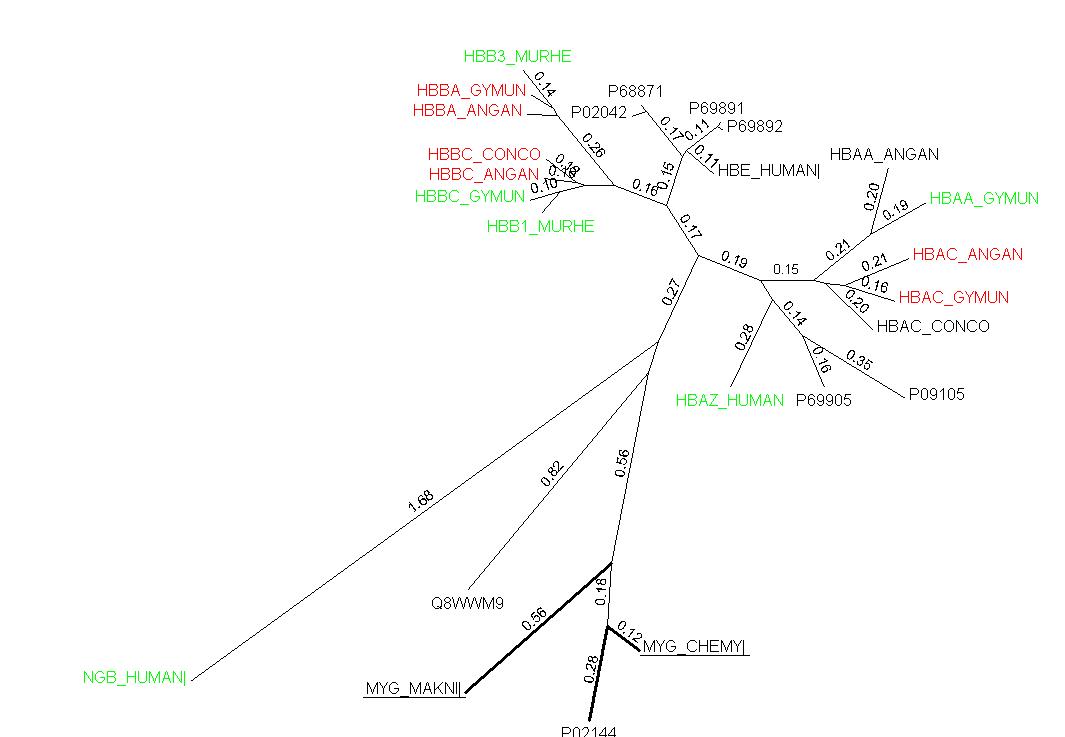
Ветви внешней группы более толстые, названия выделены жирным шрифтом с
подчеркиванием.
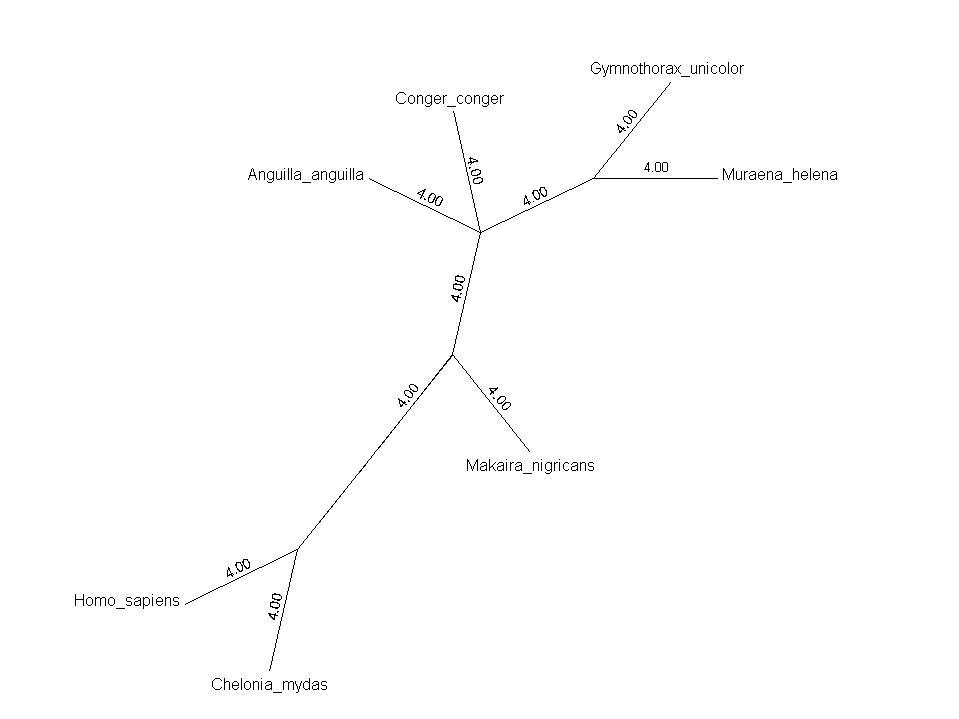
Построение таксономического дерева
На сайте NCBI по списку организмов было построено
таксономическое дерево:
Анализ дерева
Ортологи - это белки, возникшие из одного общего предшественника в процессе видообразования. Как правило, они имеют одну и ту же функцию. В данном случае ортологами можно считать, например, следующие пары:
- HBAC_ANGAN - HBAC_GYMUN
- HBBA_GYMUN - HBBA_ANGAN
- HBBC_CONCO - HBBC_ANGAN
На рисунке в п.2 эти пары отмечены красным.
Паралоги - это белки, возникшие из одного общего предшественника в результате дупликацииодного гена в одном организме. Обычно паралоги имеют разные функции. Для данной выборки паралогами являются эти пары:
- HBB3_MURHE - HBB1_MURHE
- HBBC_GYMUN - HBAA_GYMUN
- HGB_HUMAN - HBAZ_HUMAN
Эти белки отмечены зелёным.
©
Шпильман Алексей, 2005

