Навигация по сайту: |
Предсказание вторичной структуры тРНК. Комплексы ДНК-белокЗадание 1. Предсказание вторичной структуры тРНК (из структуры PDB 1H4S)С помощью программы EMBOSS einverted, запущенной с измененными опциями (gar penalty = 0, Score threshold = 0), нашли инвертированные повторы в последовательности тРНК: 1H4ST: Score 104: 25/25 (100%) matches, 21 gaps
1 cggggagtagc--gcag-c----ccggtagcg---cacctcgtt-c 35
||||||| | | | | | || |||| | ||||| | |
73 gcccctc-t-gacc-t-agacttgg---tcgcgggg-ggagc-agg 37
С помощью RNAfold получили предсказание вторичной структуры по алгоритму Зукера: 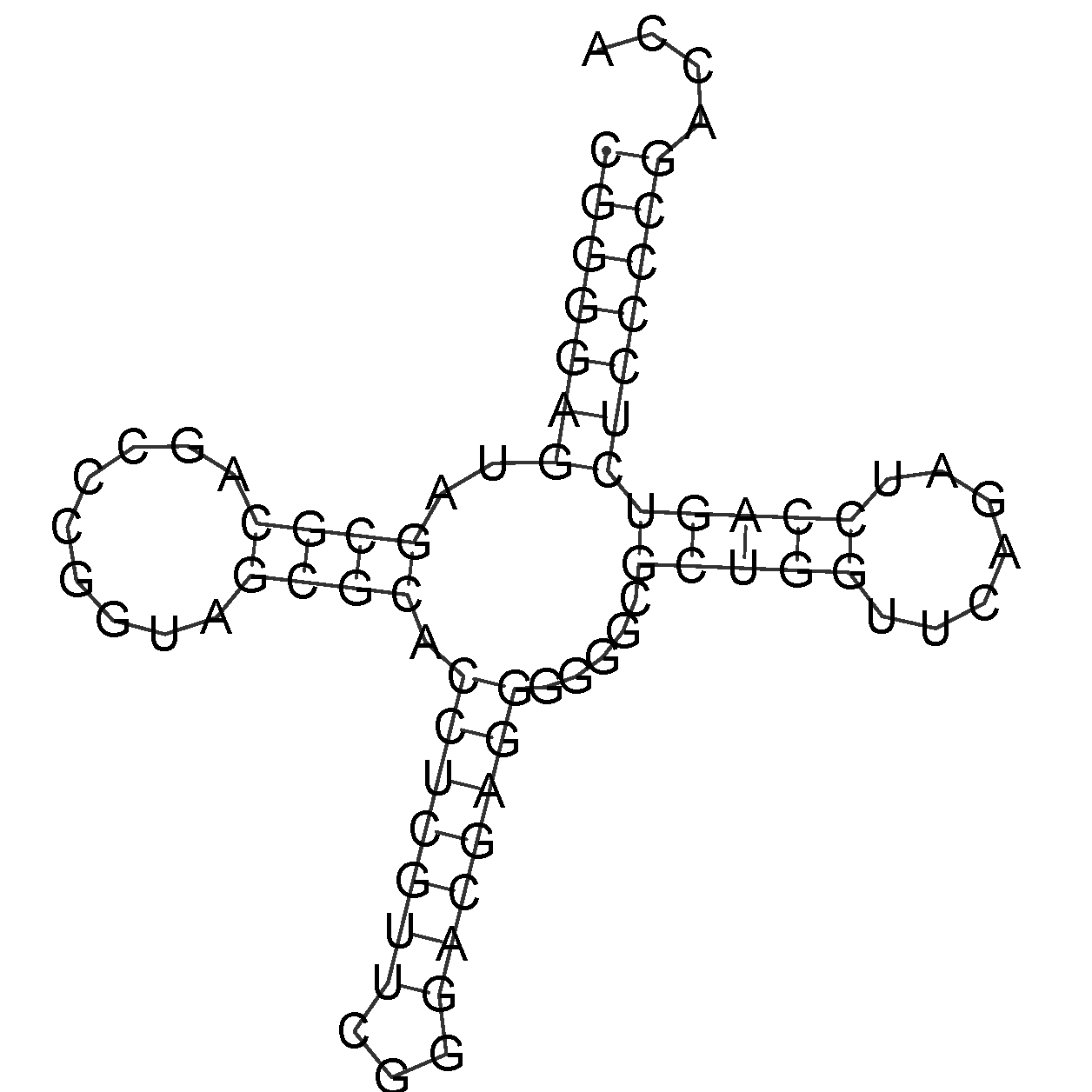
В таблице ниже приведено насколько точно программы einverted и RNAfold воспроизводят результаты find_pair.
Таким образом, RNAfold лучше подходит для предсказания вторичной структуры РНК, чем наивный подход с помощью einverted. Задание 2. Поиск ДНК-белковых контактов в структуре 1MHDПоиск контактовСкрипт с определениями наборов атомов. Скрипт с последовательным изображением set1, set2 и set3. Поскольку было указание брать любую цепь белка, описываем контакты ДНК и белка с цепью A.
Использовавшийся для удобства скрипт. Суммарно получилось 13 полярных контактов и 17 неполярных. NucplotС помощью nucplot была получена схема контактов ДНК-белок: 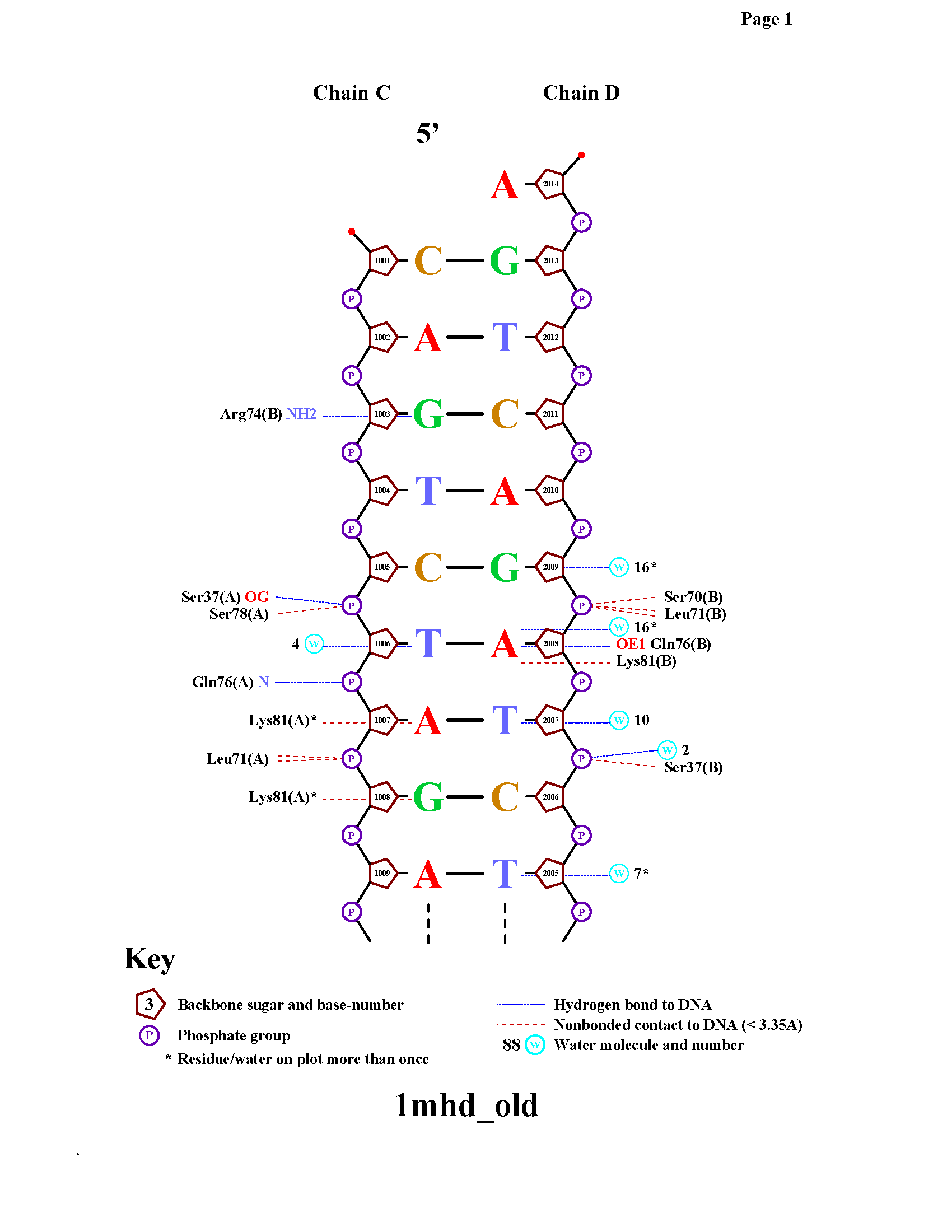
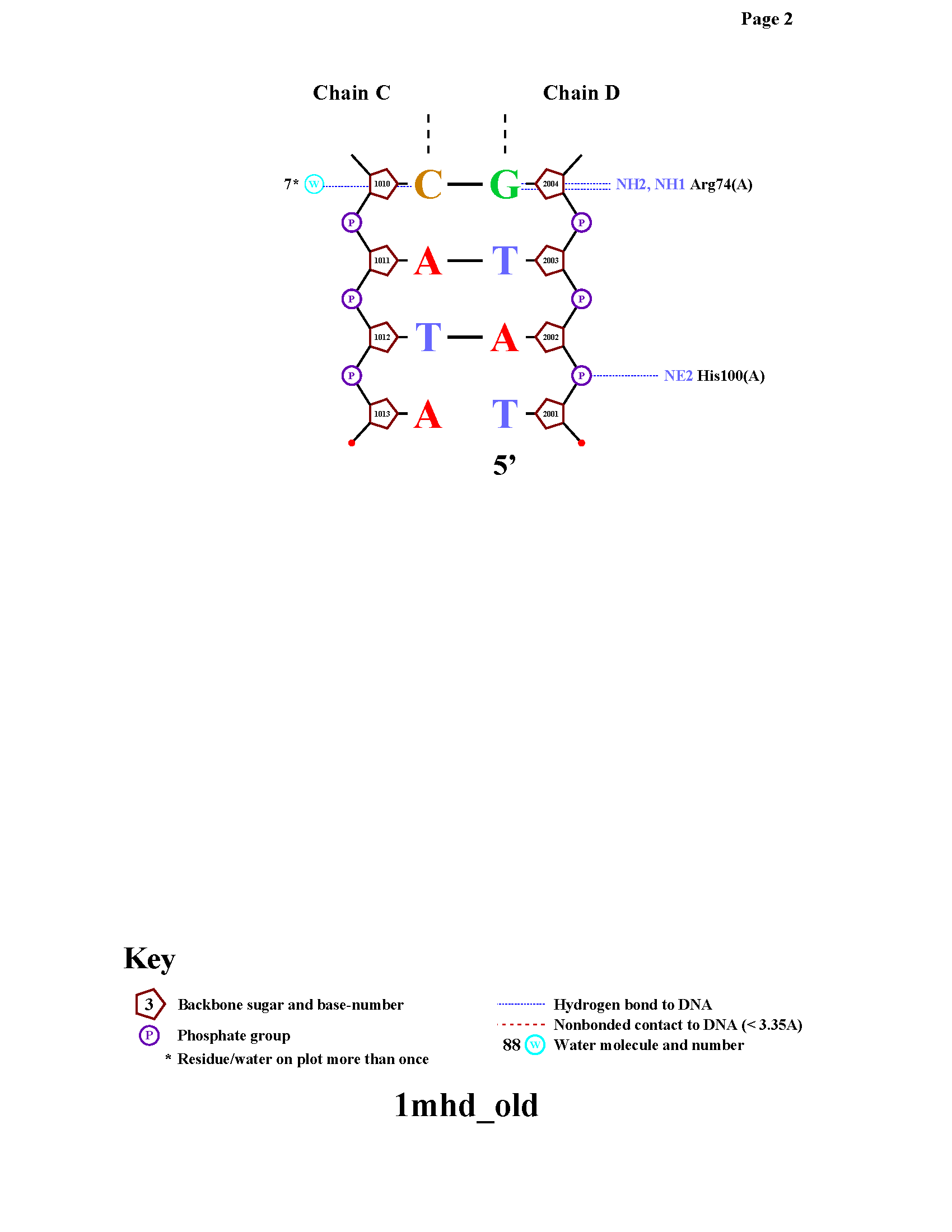
Как видно из схем, остатки K81, L71, R74 цепи А и L81 цепи B образуют наибольшее (2) число контактов с ДНК Очевидно, что для распознавания последовательности ДНК белку необходимо контактировать не с остовом, а с азотистыми основаниями. Также известно, что как правило водородные связи сильнее гидрофобных контактов. Будем ориентироваться на эти два критерия. Остаток R74 из цепи А удовлетворяет этим требованиям, и следовательно важен для распознавания последовательности ДНК. 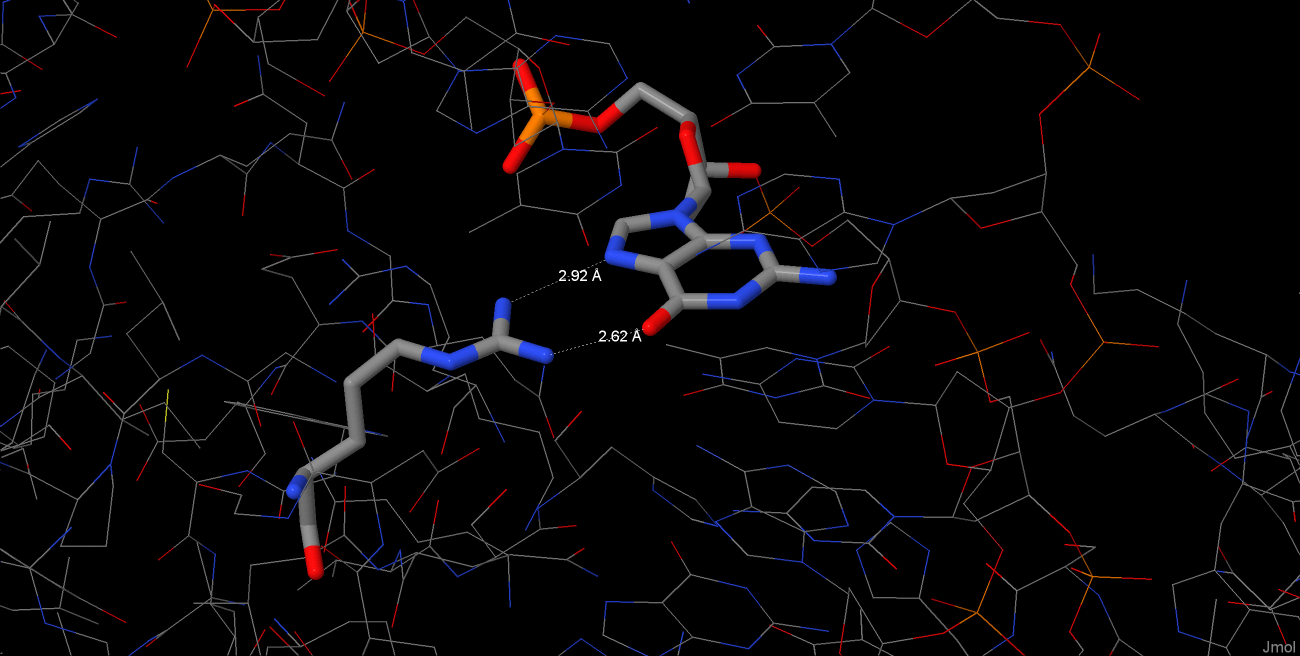
Здесь видно, что аргинин образует две хорошие (менее трех ангстрем между тяжелыми атомами) водородные связи. Такое взаимодействие довольно селективно по отношению к гуанину, так как для взаимодействия с обеими атомами азота аргинина(NH1 и NH2) требуется и атом N7(есть только у пуринов) и атом O6, который есть только у гуанина. У R74 из цепи B наблюдается похожая картина, но водородные связи не такие хорошие(одну nucplot вообще не отображает). В целом, объяснение тут аналогичное. Поскольку цепи А и В у нас один и тот же белок, то можно смело говорить о важности этого(R74) остатка. 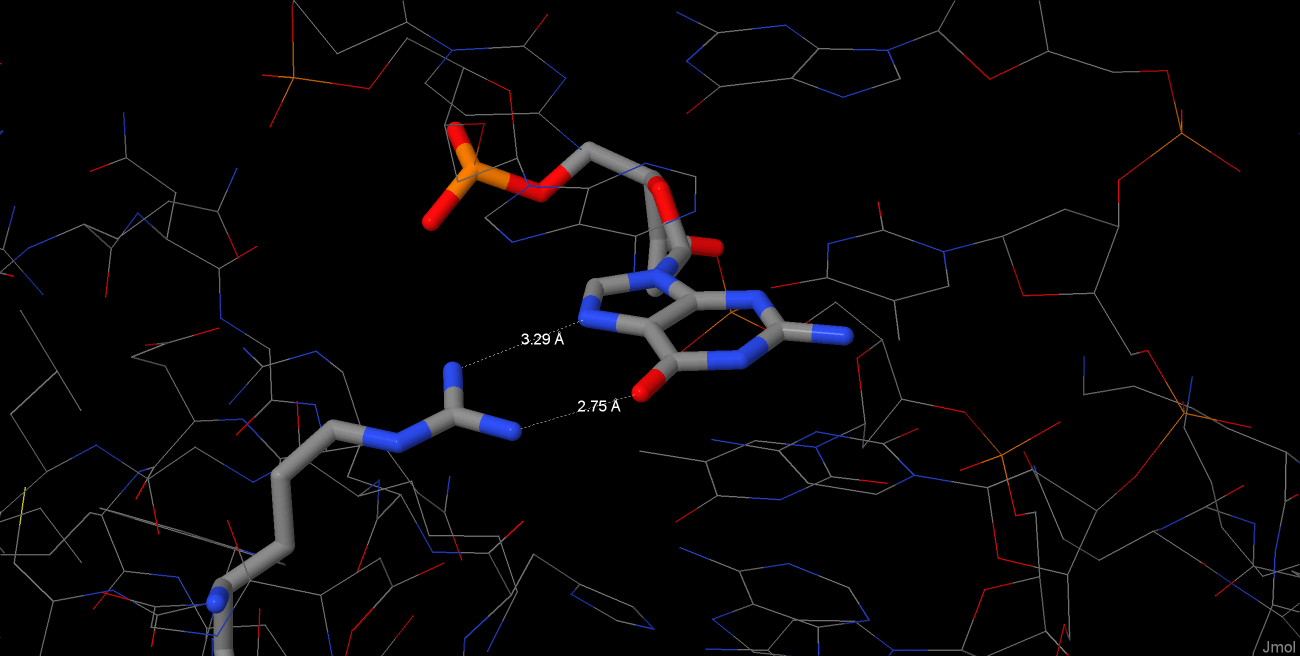
|