Навигация по сайту: |
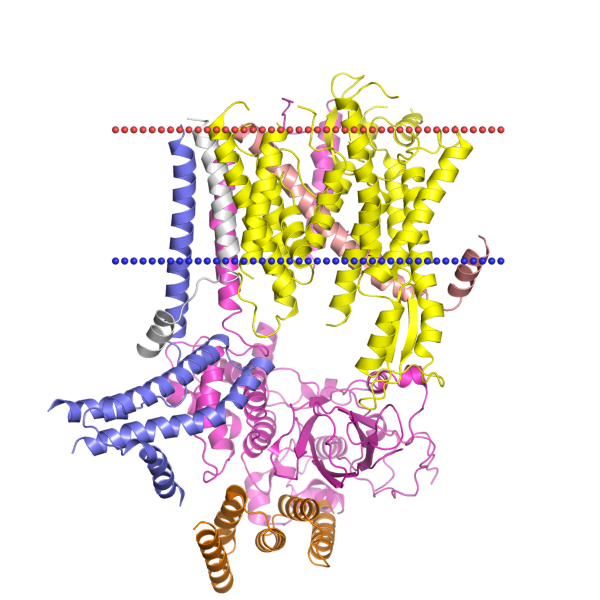
Трансмембранные белки1. База данных OPMБелок с трансмембранными α-спиралямиБудем описывать транслоказу белков Sec. Этот комплекс связывается с рибосомой (такие структуры тоже есть в OPM) и протаскивает новосинтезированный белок из цитоплазмы в ЭПР (у эукариот) или через цитоплазматическую мембрану (у прокариот).
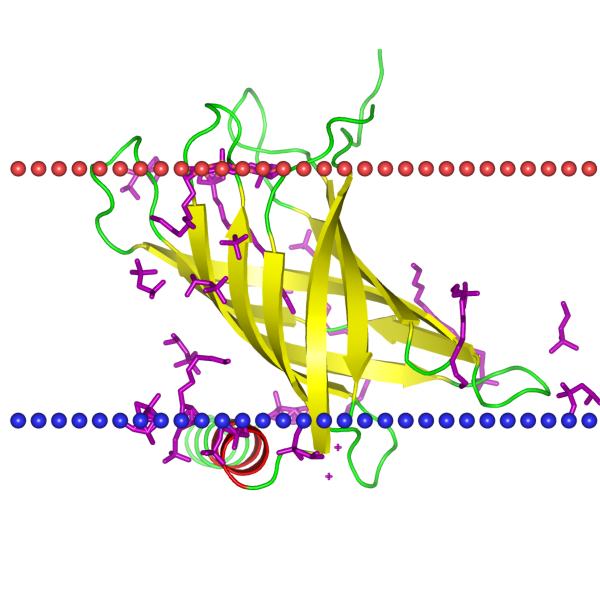
Белок с трансмембранными β-листамиРассмотрим PagP — ацилазу липида А , важного компонента липополисахаридов грамотрицательных бактерий. В мембране у нее β-бочонок из восьми листов.
2. Анализ предсказания трансмембранных спиралейВозьмем для анализа субъединицу А из комплекса Sec — белок Sec63. Сравним то, как работают два пресказателя трансмембранных спиралей: TMHMM и Phobius. TMHMMПодробный текстовый вывод (Extensive, no graphics): # 6ND1:A|PDBID|CHAIN|SEQUENCE Length: 677 # 6ND1:A|PDBID|CHAIN|SEQUENCE Number of predicted TMHs: 3 # 6ND1:A|PDBID|CHAIN|SEQUENCE Exp number of AAs in TMHs: 65.9381300000000001 # 6ND1:A|PDBID|CHAIN|SEQUENCE Exp number, first 60 AAs: 22.44151 # 6ND1:A|PDBID|CHAIN|SEQUENCE Total prob of N-in: 0.71814 # 6ND1:A|PDBID|CHAIN|SEQUENCE POSSIBLE N-term signal sequence 6ND1:A|PDBID|CHAIN|SEQUENCE TMHMM2.0 inside 1 12 6ND1:A|PDBID|CHAIN|SEQUENCE TMHMM2.0 TMhelix 13 35 6ND1:A|PDBID|CHAIN|SEQUENCE TMHMM2.0 outside 36 87 6ND1:A|PDBID|CHAIN|SEQUENCE TMHMM2.0 TMhelix 88 107 6ND1:A|PDBID|CHAIN|SEQUENCE TMHMM2.0 inside 108 221 6ND1:A|PDBID|CHAIN|SEQUENCE TMHMM2.0 TMhelix 222 244 6ND1:A|PDBID|CHAIN|SEQUENCE TMHMM2.0 outside 245 677 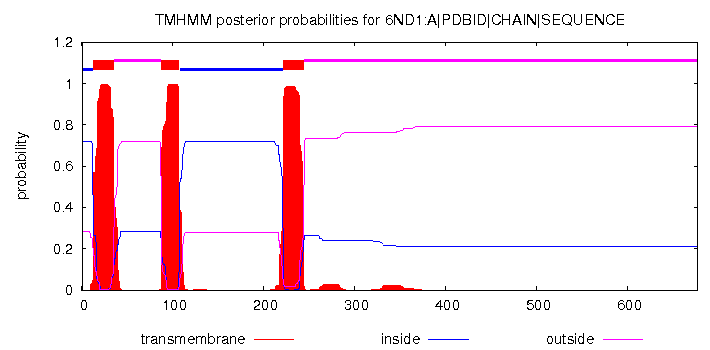
Расшифровка картинки: по оси ОХ отложена последовательность белка, по оси ОУ — вероятность предсказания. Для каждой возможной локализации аминокислоты соотвествующим цветом построен график вероятности, с которой она находится либо в трансмембранной спирали, либо в части белка с наружней стороны мембраны, либо с внутренней стороны мембраны. Выше единицы, в верхней части графика толстыми линиями отложено решение программы (для каждой аминокислоты просто наиболее вероятное предсказание). Цвета: красный — трансмембранная спираль, сиреневый — наружняя сторона мембраны, синий — внутренняя. PhobiusПодробный текстовый вывод (Long without Graphics): ID 6ND1:A|PDBID|CHAIN|SEQUENCE FT TOPO_DOM 1 13 NON CYTOPLASMIC. FT TRANSMEM 14 32 FT TOPO_DOM 33 92 CYTOPLASMIC. FT TRANSMEM 93 110 FT TOPO_DOM 111 218 NON CYTOPLASMIC. FT TRANSMEM 219 239 FT TOPO_DOM 240 677 CYTOPLASMIC. // 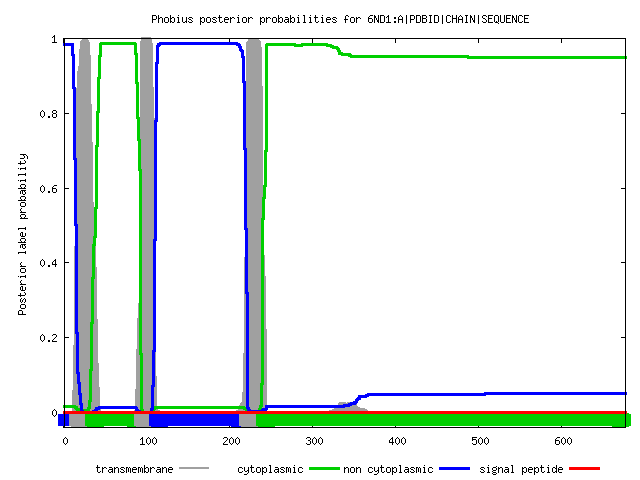
Расшифровка картинки: по оси ОХ отложена последовательность белка, по оси ОУ — вероятность предсказания. Для каждой возможной локализации аминокислоты соотвествующим цветом построен график вероятности, с которой она находится либо в трансмембранной спирали, либо в части белка с наружней стороны мембраны, либо с внутренней стороны мембраны. Ниже нуля, под графиком в виде прямоугольников отложено решение программы (для каждой аминокислоты просто наиболее вероятное предсказание). Цвета: серый — трансмембранная спираль, синий — наружняя сторона мембраны, зеленый — внутренняя, красный — сигнальная последовательность. Сравнение вывода программ и реальностиДанные по предсказаным спиралям собраны в таблице 3:
Из таблицы 3 видно, что TMHMM и Phobius неплохо справляются со своей задачей, иногда даже точно определяя координаты трансмембранных спиралей, при этому никогда друг с другом не соглашаясь (получились везде чуть-чуть разные спирали). Любопытно то, что был предсказан еще один трансмембранный регион, который в OPM не указан. Он скорее всего там действительно есть (в структуре присутствуют "огрызки" этой спирали, по которым это можно понять), но из-за качества структуры OPM его просто не указал. Такое вот торжество биоинформатического подхода над экспериментальным. 3. База данных TCDBSec63Из комплекса Sec снова возьмем только цепь А (белок Sec63). Местный вариант BLAST-a сразу нашел белок (Е-value = 0). Код TC: 3.A.5.8.1. Расшифровка кода:
PagPТак как PagP не транспортер, вряд ли он будет в БД транспортеров. Текстовый поиск не дал результатов, местный вариант BLAST-a дал только одну находку с Е-value 0.6 и маленьким покрытием. Её тоже можно проигнорировать. |