Секвенирование по Сэнгеру
Что было сделано:
- с помощью программы UGENE были прочтены хроматограммы прямой и обратной последовательности под кодом 12
- с помощью программы Jalview было произведено выравнивание данных последовательностей, и получена консенсусная последовательность нуклеотидов
Даны были последовательности - прямая и обратная - неизвестной мне ДНК, секвенированной по Сенгеру, под кодом 12 в формате ab1. Я открыла их в UGENE и посмотрела на их хроматограммы.
Ссылки на исходные последовательности представлены здесь:
Вначале выровняем прямую и обратную последовательности. Можно было это сделать в GeneStudio или UGENE, но получилось только в Jalview (выравнивание Clustal with Defaults).
Определим нечитаемые концы: прямая последовательность:5' 1-61 в хроматограмме (1-91 в выравнивании), 676-719 в хроматограмме (707-750 в выравнивании); обратная последовательность:5' 1-59 в хроматограмме (660-719 в выравнивании), 684-718 в хроматограмме (1-35 в выравнивании).
Примечание: файл с первоначальным выравниванием можно найти здесь.
Удалим нечитаемые концы в выравнивании.
Снова выравниваем. Файл с "перевыравниванием" можно посмотреть тут.
Если "на глаз" определить уровень шума, то он будет примерно 10-15%. Встречаются хроматограммы и почетче:). В середине пики читаются прекрасно, по краям качество оставляет желать лучшего. То же можно сказать о силе сигнала. В центральной части сигнал средний, идет ровно; в самом начале хроматограммы сигнал слишком сильный (перегрузка короткими фрагментами), в конце плавно затухающий. В этой хроматограмме встречаются "пузыри", возможно, это несвязавшиеся ddNTP.
Отредактируем прямую последовательность, просмотрев целиком ее хроматограмму:
1)В позициях 68 и 69 нетипично далеко друг от друга расположены нуклеотиды. Редактируем: на 68 позиции хроматограммы отчетливо виден нуклеотид А, принятый по ошибке за N. Cледующий за ним пик, подходящий по расположению - G, а красный пик N больше походит на шум, чем на нуклеотид. Получаем А и G на 68 и 69 позициях соответственно.
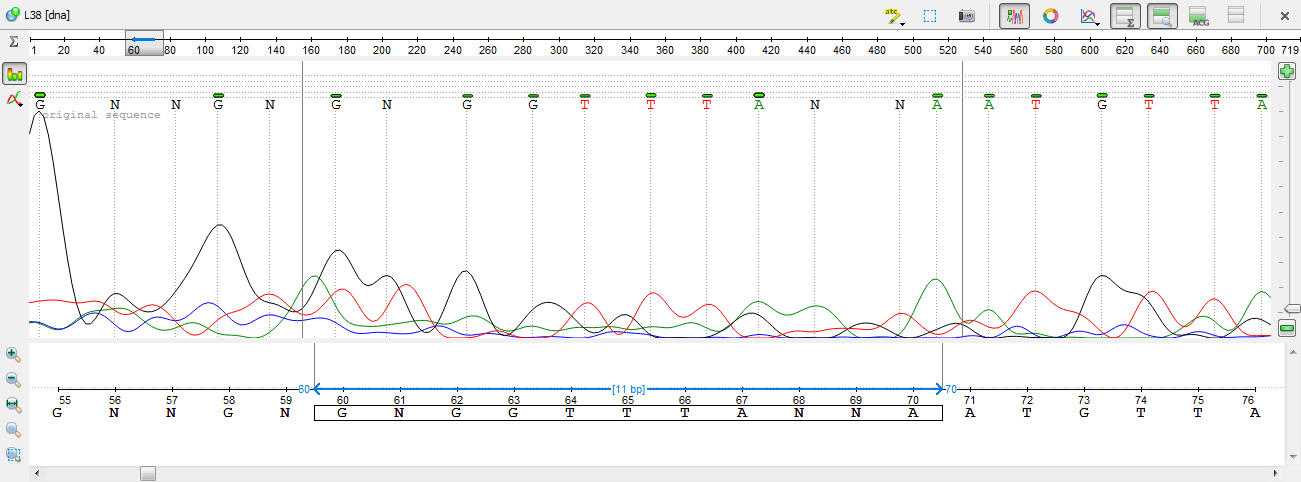
2)Зеленый пик на 81 позиции больше походит на шум, если судить по окружению. Поменяем на G.
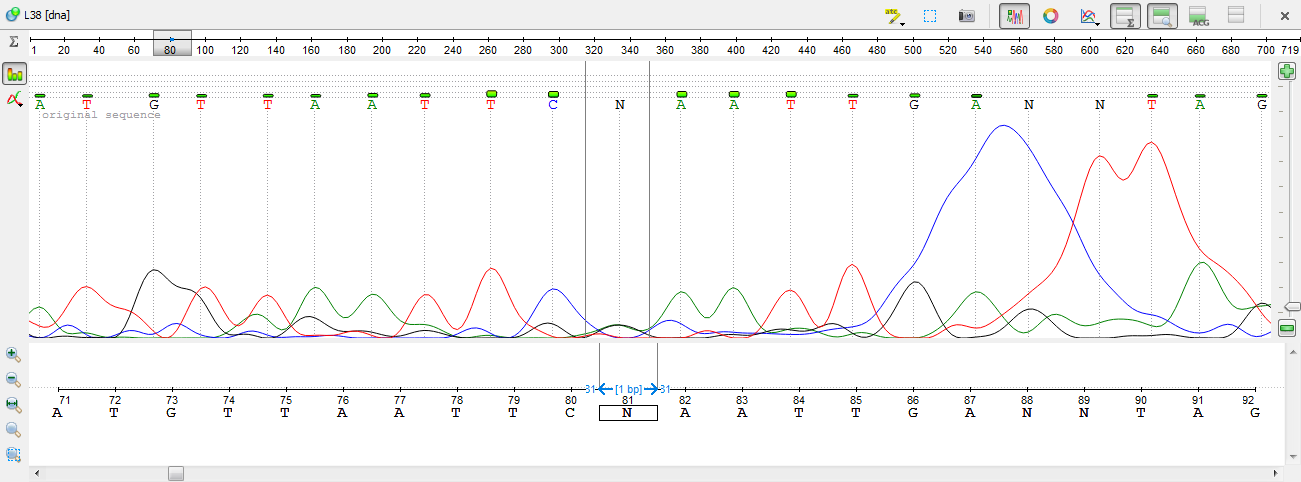
3)На позициях 88-89 зеленый пологий сигнал - шум, большой, широкий синий пик - несвязавшийся ддНТФ (но это не точно), Больше всего похож на сигнал черный пик на 88 позиции и красный пик на 89 позиции. В итоге поменяли нуклеотиды N на G и Т на 88 и 89 позициях соответственно.
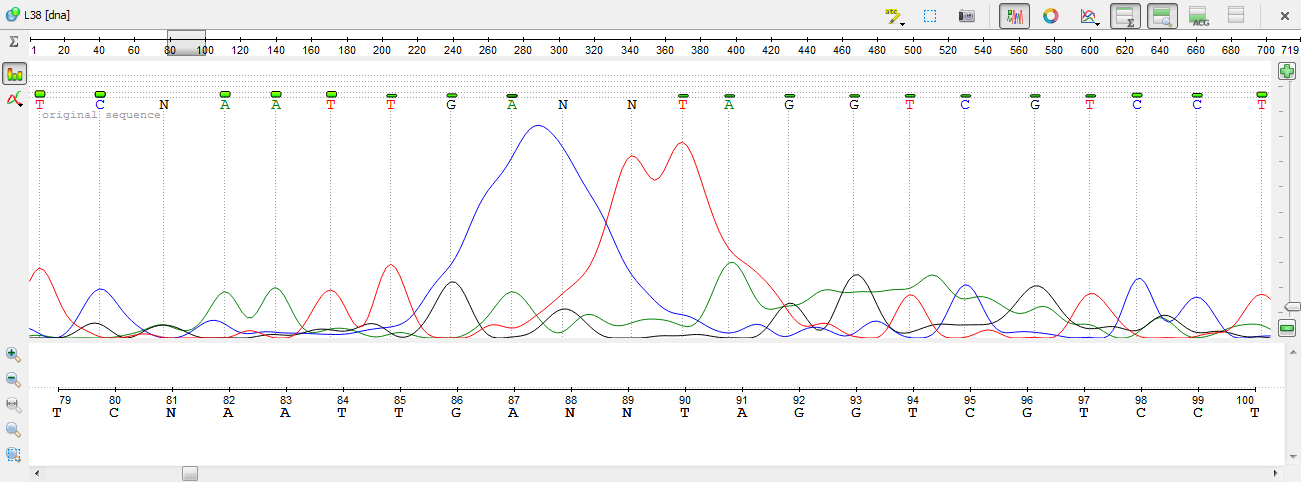
4)На 105 позиции синий и зеленый пики - точно шум. Осталось решить, какой пик истинный. Если судить по выравниванию, то тут должен быть нуклеотид G, т.е. черный пик истинный.
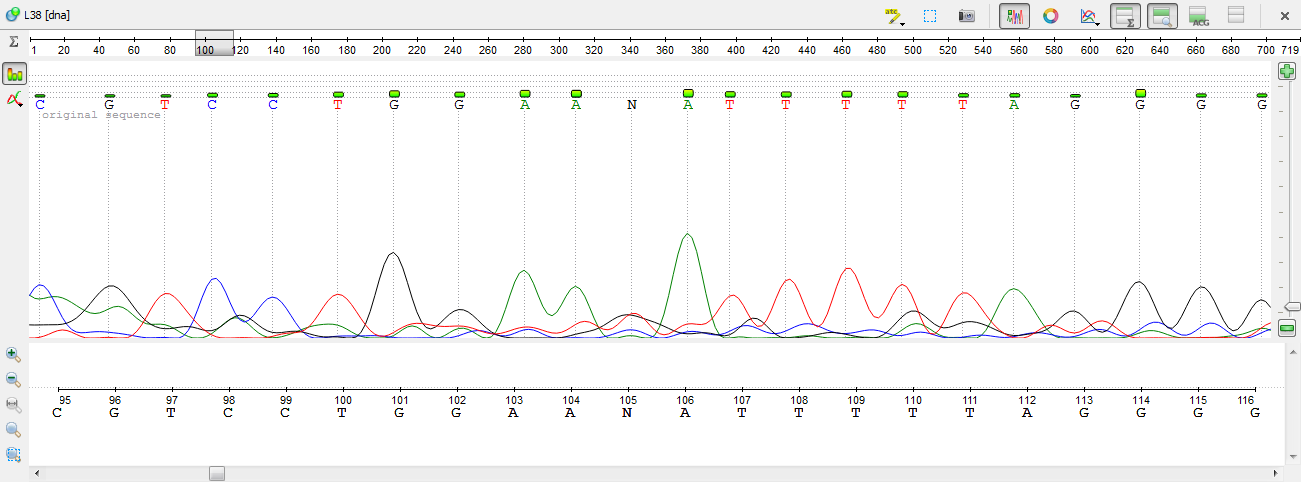
5)На позиции 158 виден слабый пик - сигнал гуанина. Поменяем N на G.
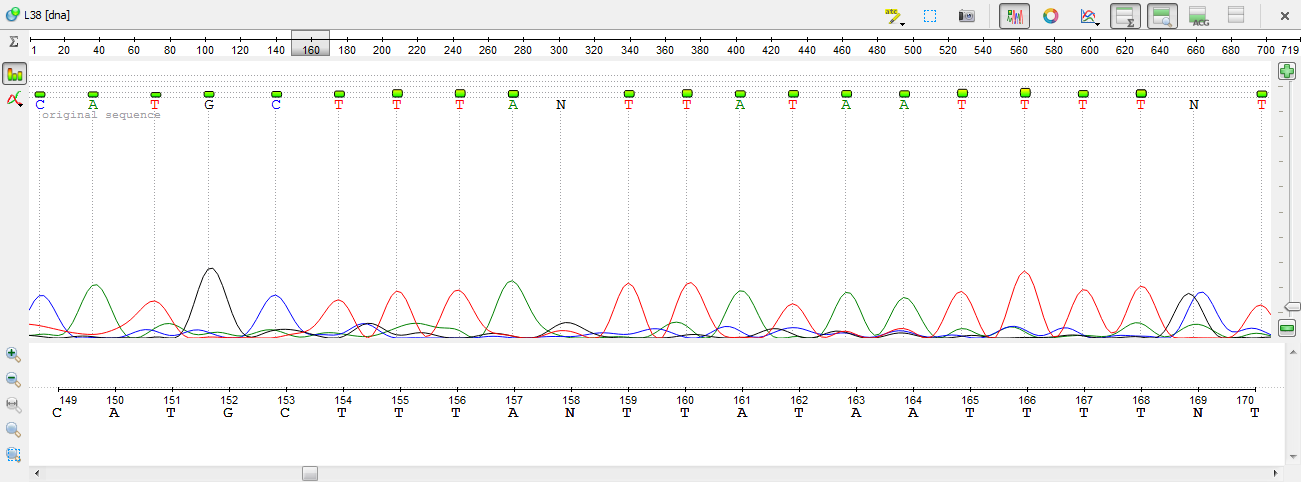
6)На позиции 169 имеются сигналы гуанина и цитозина одинаковой интенсивности. Обратившись за помощью к выравниванию, мы ответа не получим. Поменяем N на S соответственно таблице.
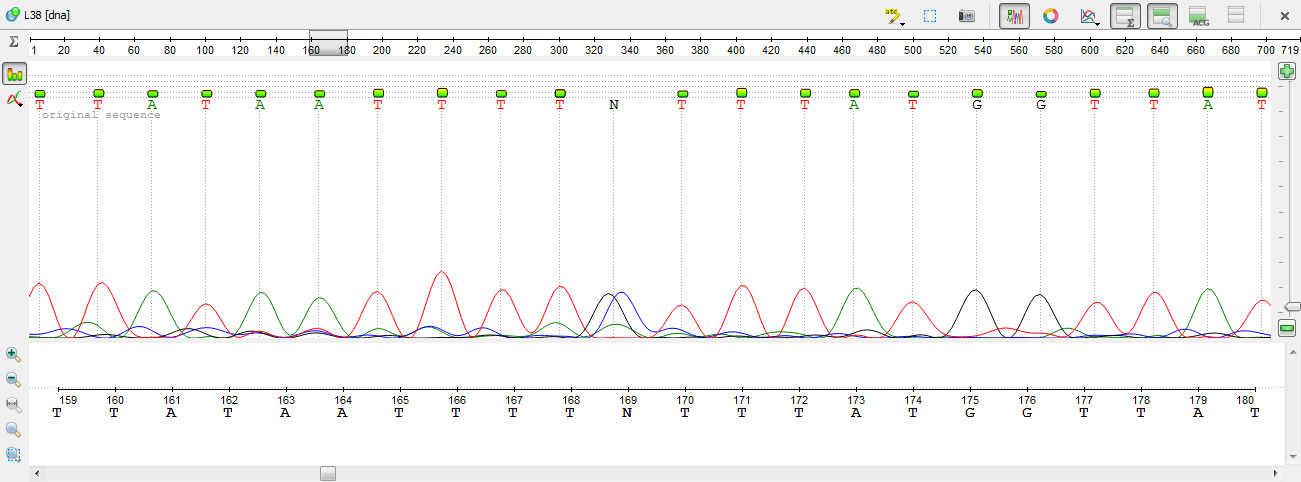
7)На позиции 387 зеленый сигнал больше похож на шум, поэтому ставим во внимание только сигнал гуанина. На всякий случай обратимся к выравниванию. Оно нам говорит, что наш выбор скорее всего, верный.
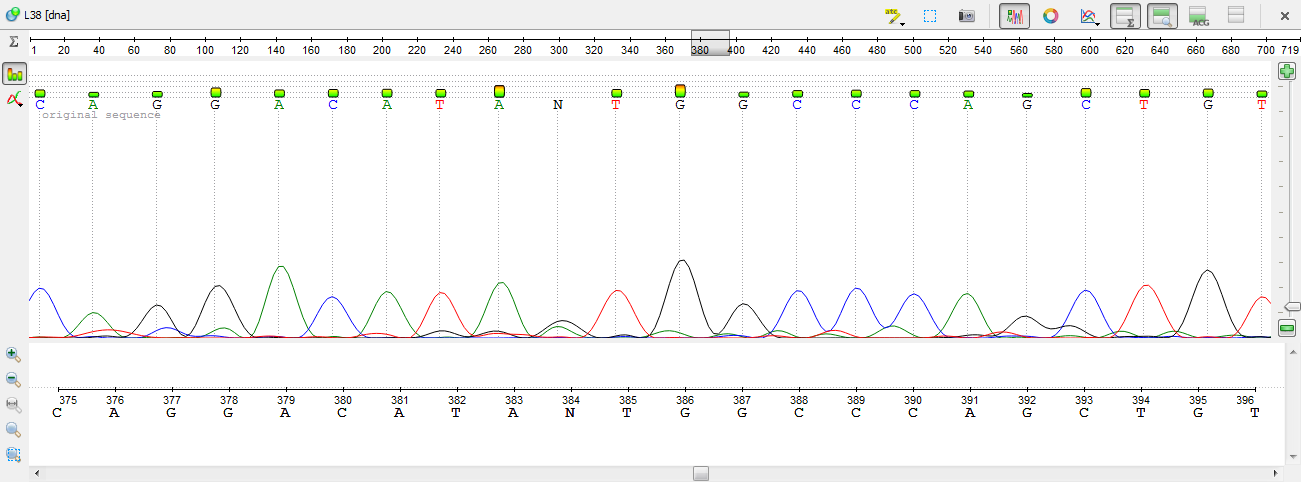
8)На позиции 464 поменяем N на А. Тут, вроде бы, все очевидно.
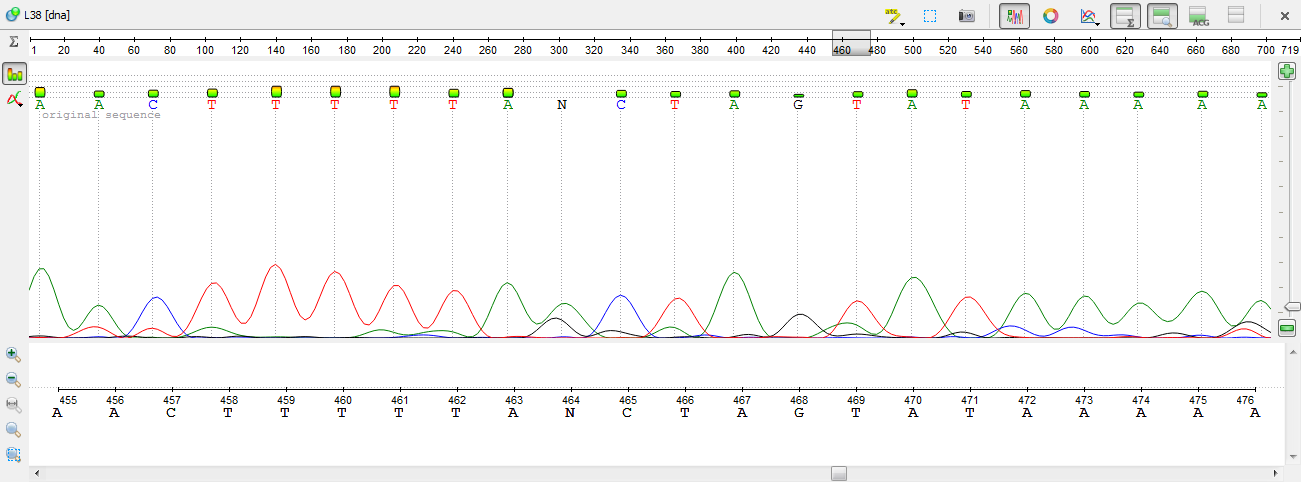
9)На позиции 560 находим, что шумовой уровень зеленого сигнала в окружении выше, чем у красного. Поэтому ставим во внимание красный пик. Меняем N на T.
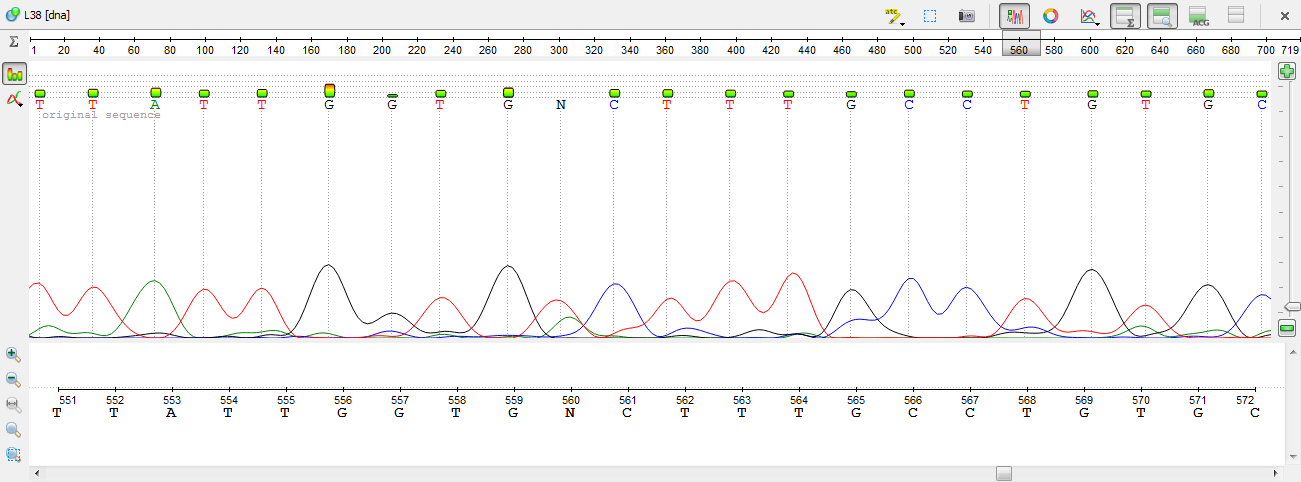
осталось совсем немного
10)Красный "пузырь" на 636 позиции не внушает доверия. Смело меняем N на А.
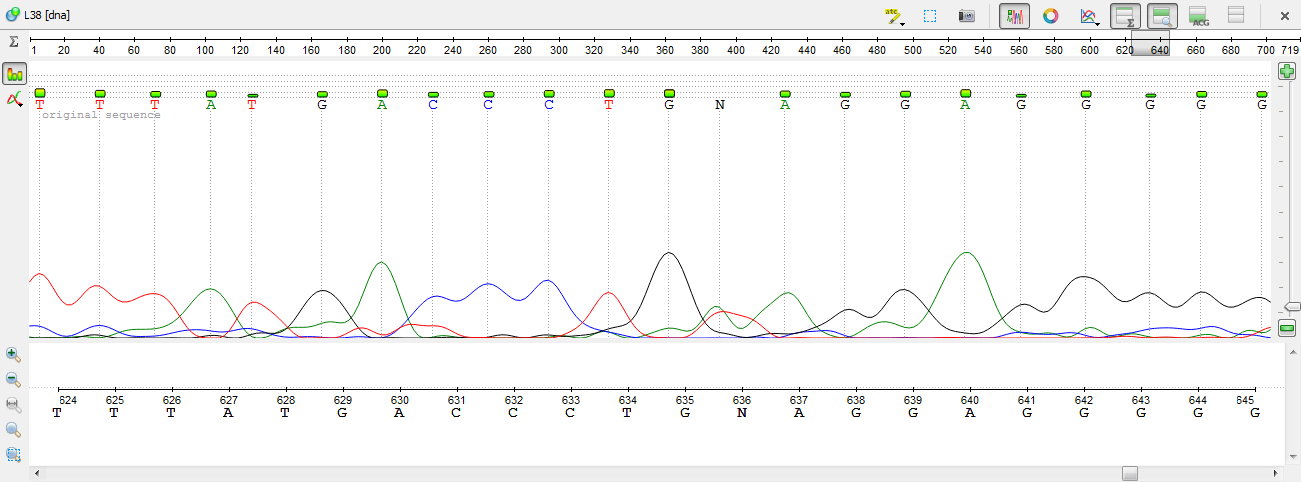
11)Пик на 669 пизиции несколько смещен, поэтому определен неправильно. Меняем N на Т.
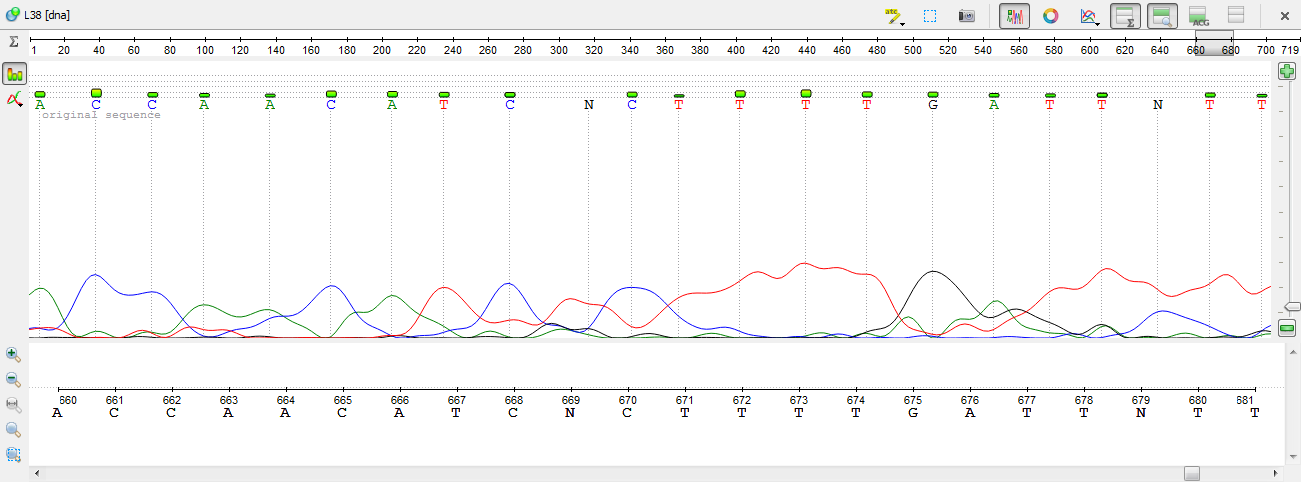
Нечитаемый фрагмент:
Пример нечитаемого фрагмента хроматограммы приведен на картинке ниже. Здесь представлено характерное для начала хроматограммы большое количество пиков всех цветов. Это может быть обусловлено выходом из колонки несвязанных флюоресцирующих ддНТФ.
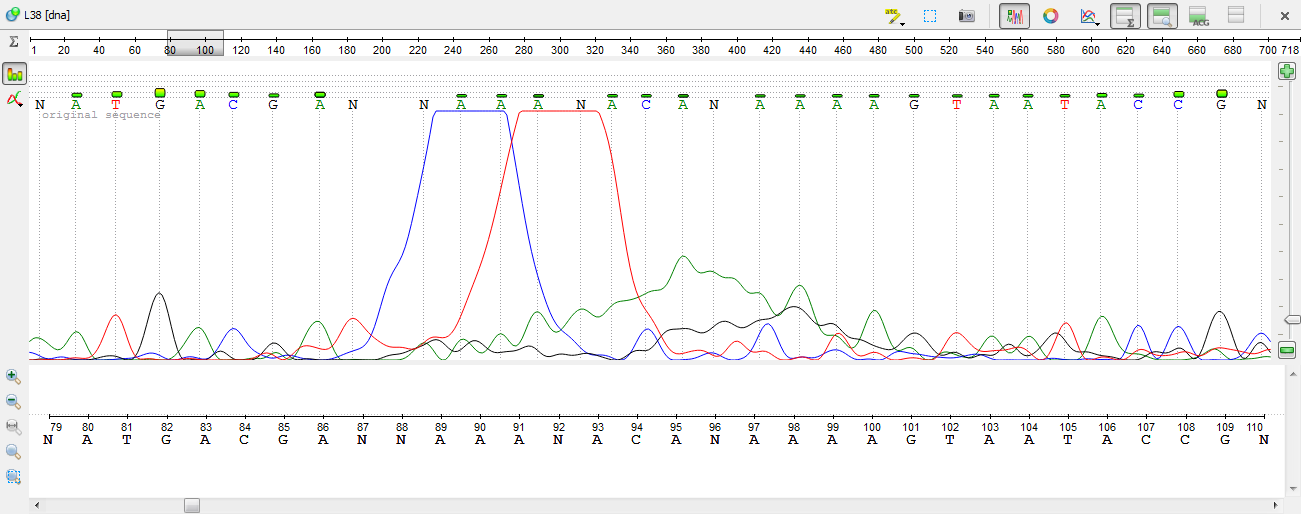
Стоит отметить, что существует страничка на сайте центра коллективного пользования "ГЕНОМ", на которой грамотно описаны возможные проблемы чтения хроматограмм секвенирования по Сэнгеру и способы их устранения.
С обратной последовательностью проделываем все то же самое. Результатом должны быть консенсусная последовательность, которую я получила в Jalview, и итоговое выравнивание.

