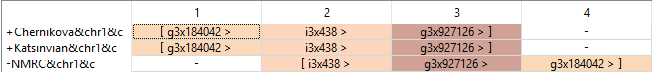
Выравнивание (qnpge):
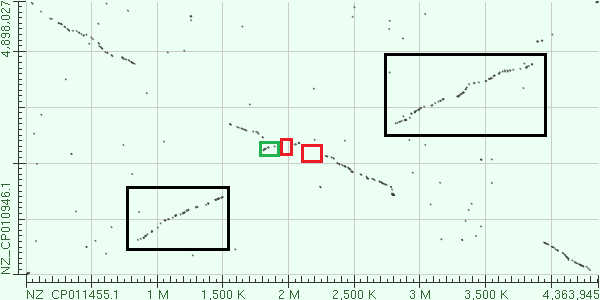
На рисунке приведена карта локального сходства бактерий Mycobacterium bovis (query - Ox) и Mycobacterium chelonae (subject - Oy).
Черным выделены 2 куска, претерпевших транслокацию и инверсию (0.8 - 1.5M и 2.8 - 3.85M по Ox), зеленым - кусок, претерпевший инверсию (1.8 - 1.9M), красным показаны вставки (1.9 - 2M и 2.1 - 2.15M) в последовательность Mycobacterium bovis.
В таблице описаны выбранные штаммы
| Штамм | Описание |
| Rickettsia prowazekii str. NMRC Madrid E | CP004888.1 |
| Rickettsia prowazekii str. Chernikova | CP003391.1 |
| Rickettsia prowazekii str. Katsinyian | CP003392.1 |
Число g-блоков - 3
Выравнивание (qnpge):

Число s-блоков - 15
Суммарная длина - 1105998 (99.63%)
Процент консервативных позиций в объединенном выравнивании s-блоков - 99.93%
Число r-блоков - 7
Описание двух из них:
| Блок | Число фрагментов | Число генов | Длина | identity, % |
| r7x165 | 7 | 7 | 165 | 95.75 |
| r6x140 | 6 | 6 | 140 | 93.92 |
h-блок всего один, его и опишу (его нет у str. Chernikova):
| Блок | Число фрагментов | Число генов | Длина | identity, % |
| h2x1812 | 2 | 2 | 1812 | 100 |
u-блок всего один - u1x246 - принадлежит str. Chernikova:
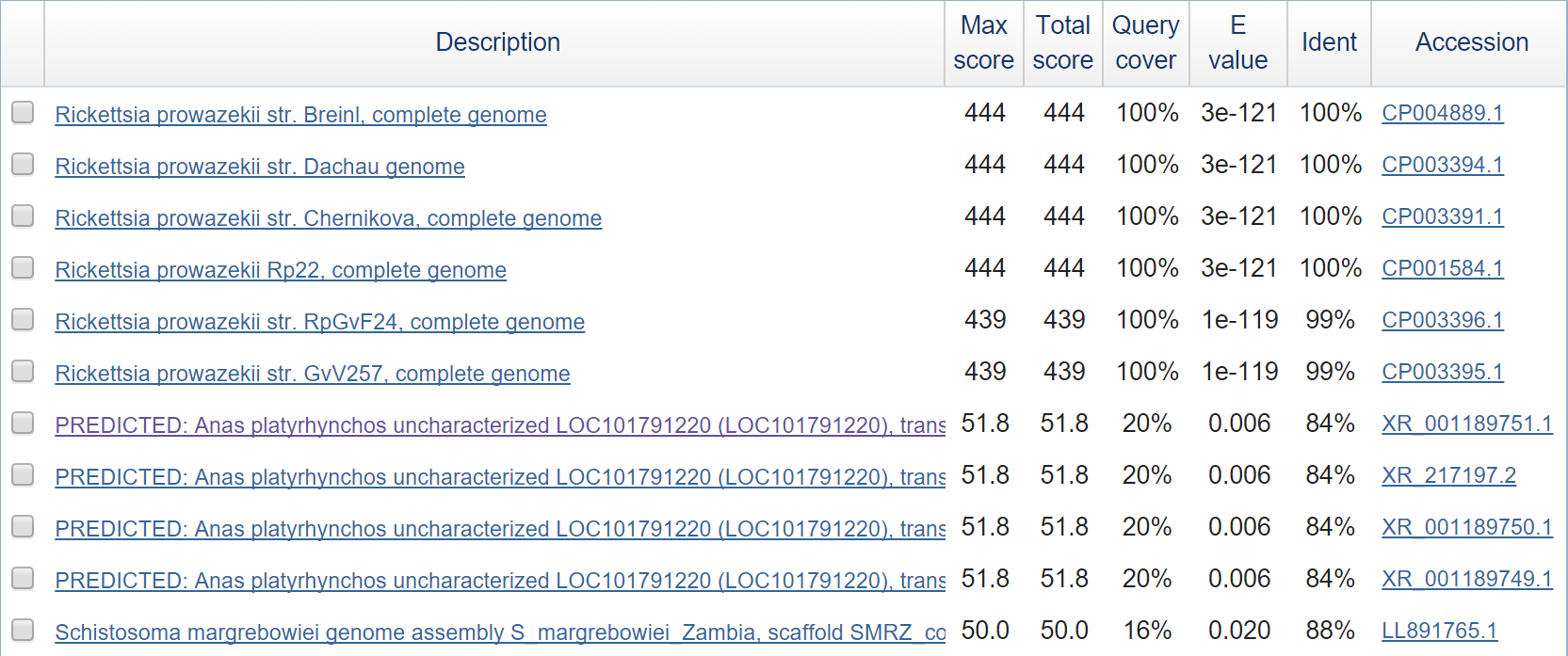
Эта последовательность присутствует у кучи других штаммов Rickettsia, но у организмов других видов ее, похоже, нет (Query cover маленький слишком, а последовательность и так небольшая).
Видимо, не было никакого горизонтального переноса.
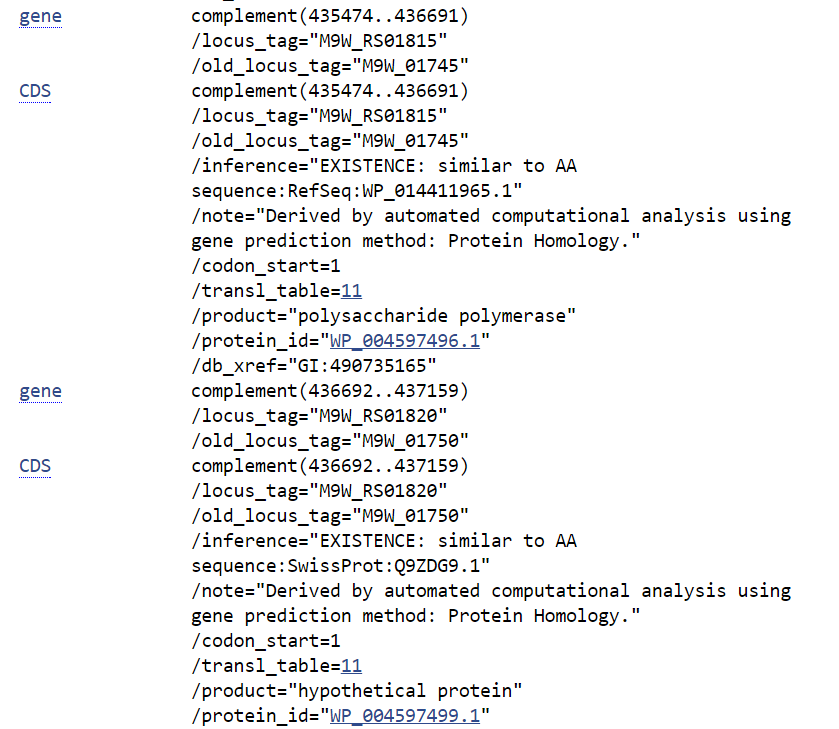
s3x133: только у str. NMRC аннотирован ген (хотя выравнивание - полное)
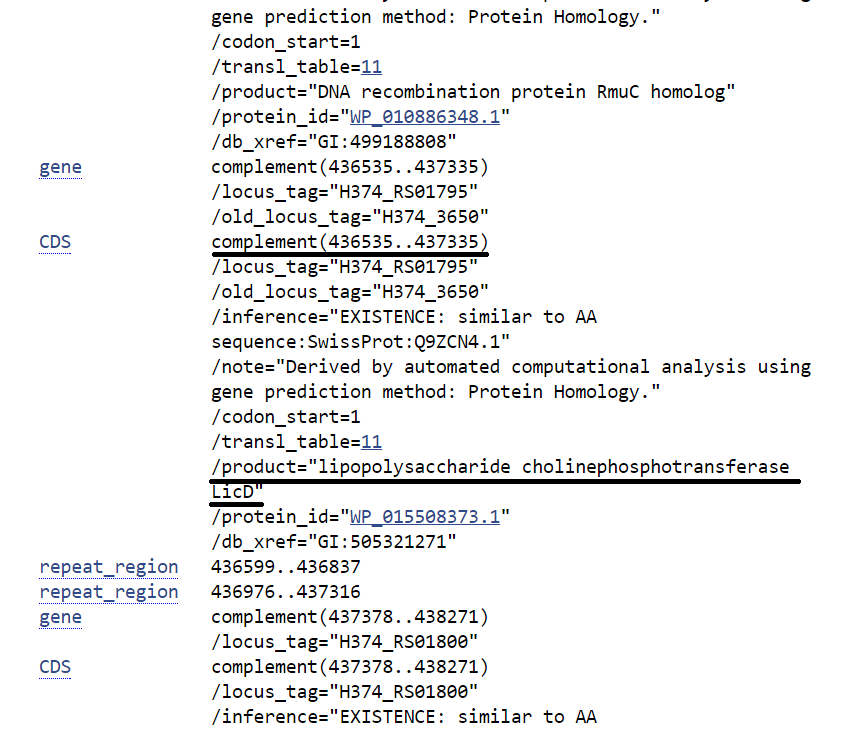
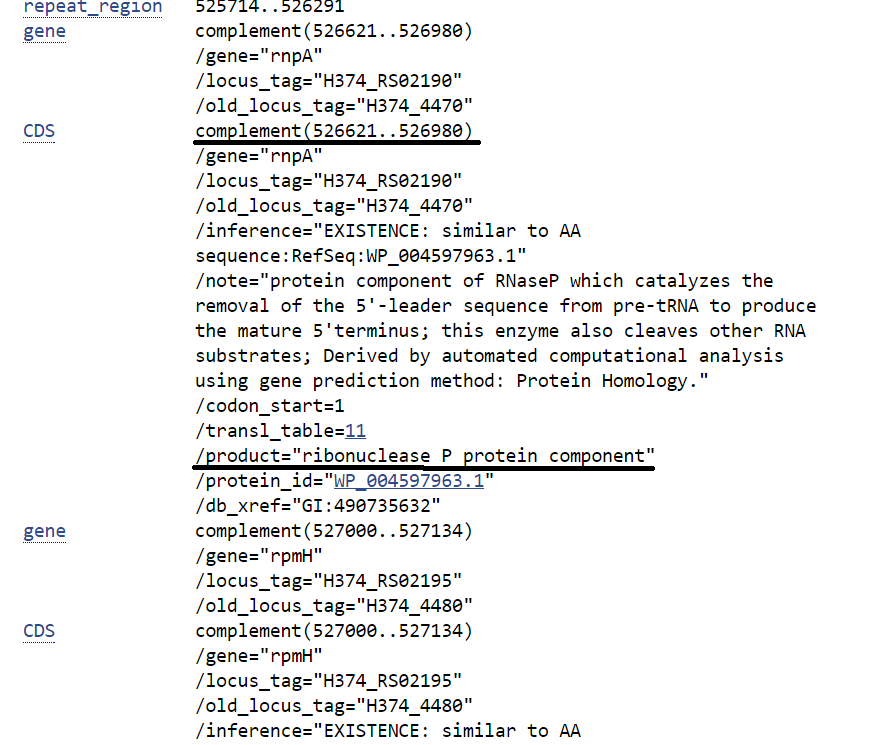
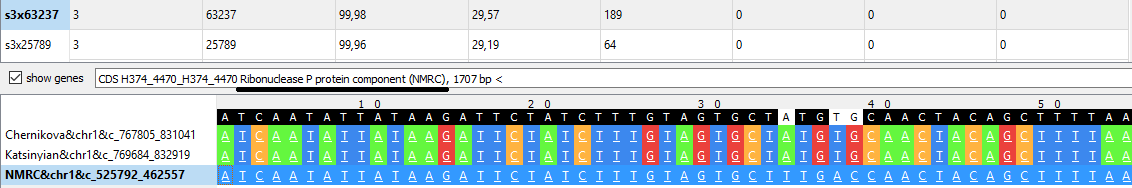
s3x63237: только у str. NMRC аннотирован ген (хотя выравнивание - полное)

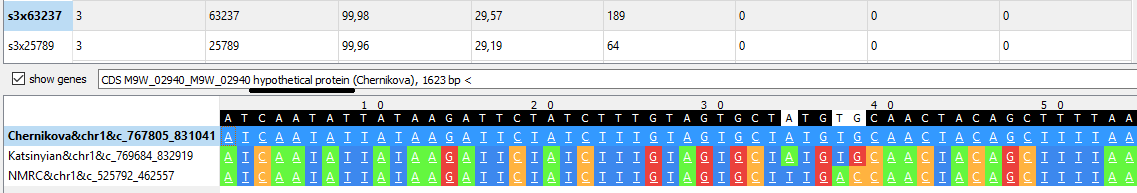
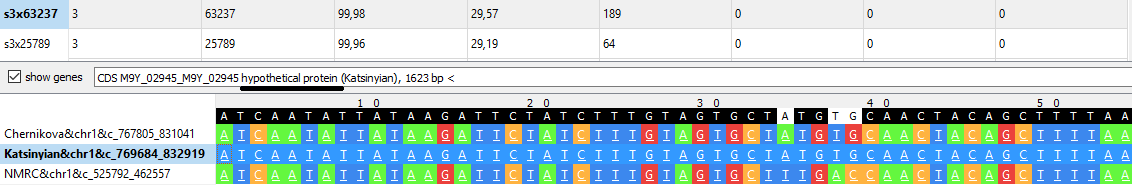
Все это было очень удобно находить с помощью qnpge:
Пакет npge достаточно удобен в работе, особенно для исправления ошибок в аннотациях, определения синтеничных участков и глобальных событий в эволюции штаммов.