Ссылки на исходные файлы:
Прямая последовательностьВ прямой последовательности не читаются: 5'-1-31, 3'-375-378
В обратной последовательности не читаются: 5'-1-67, 3'-409-413
Таким образом, после удаления нечитаемых концов первый читаемый нуклеотид в прямой последовательности соответствует 62 нуклеотиду обратной цепи, а последний читаемый нуклеотид обратной цепи соответствует 302 нуклеотиду прямой.
В среднем, шум в 5-8 раз менее интенсивен, чем сигнал. Сила сигнала и шума довольно равномерна. Пики у нуклеотидов разные, могут различаться не более, чем в 5 раз. В целом, качество хроматограмм хорошее (возможно, это связано с не очень большой длиной)

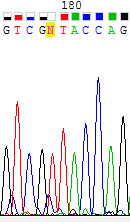
Рис. 1,2 Участки прямой и обратной цепи соответственно. Отчетливо видно, что на месте N должен стоять T.
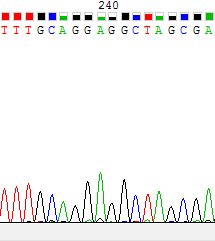
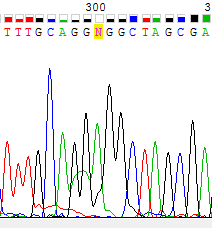
Рис. 3,4 Участки прямой и обратной цепи соответственно. Отчетливо видно, что на месте N должен стоять A
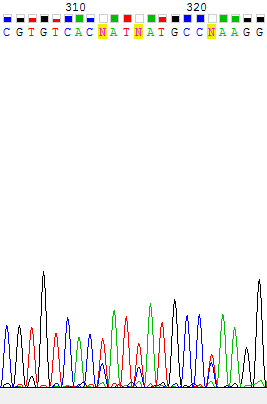
Рис. 5 Участок прямой цепи. Отчетливо видно, что на месте первого N должен стоять T, на месте второго - T. Решить, что поставить вместо третьего N, не представляется возможным
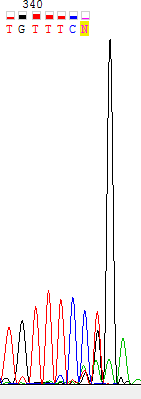
Рис. 6 Участок прямой цепи. Отчетливо видно, что на месте N должен стоять С
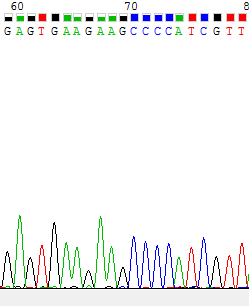
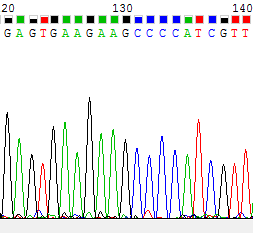
Рис. 7,8 Участки прямой и обратной цепи соответственно. Здесь все норм.
Не очень хорошая хроматограмма:
Ссылка на файл с хроматограммой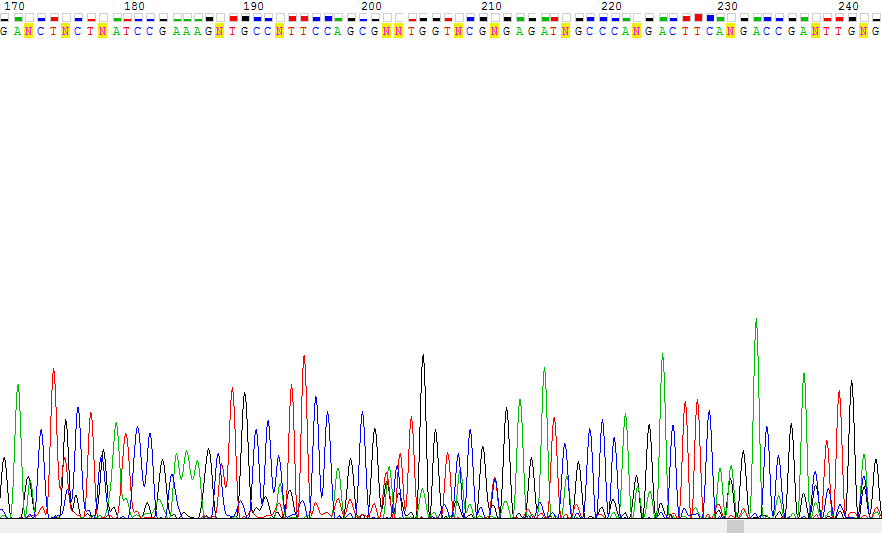
Рис. 6 Пример нечитаемой хроматограммы. Большой уровень шума.