Предсказание генов эукариот
Предсказание генов с помощью AUGUSTUS
Из сборки генома X5 я выбрал scaffold-266, длина - 47841 п.о.
blastx показал, что из списка организмов AUGUSTUS наибольшее сходство имеет Candida tropicalis.
Этот организм я и использовал для работы.
Файлы, которые выдал AUGUSTUS
- augustus.aa - аминокислотные последовательности для предсказанных генов (транслированные)
- augustus.cdsexons - нуклеотидные последовательности экзонов(стоп-кодон не указывается)
- augustus.codingseq - кодирующие нуклеотидные последоавтельности для предсказанных генов
- augustus.gbrowse - вся информация (начало, конец генов, интроны, экзоны и др.) в формате GenBank
- augustus.gff - то же только в формате таблицы с разделением на гены и приведенной для каждого гена CDS
- augustus.gtf - то же только в формате обычной таблицы
Проверка предсказания с помощью blastp
Генов было предсказано 14 - augustus.aa
- Ген 1 - BLAST совсем ничего не нашел - скорее всего, находка ошибочна
- Ген 5 - BLAST нашел что-то такое:
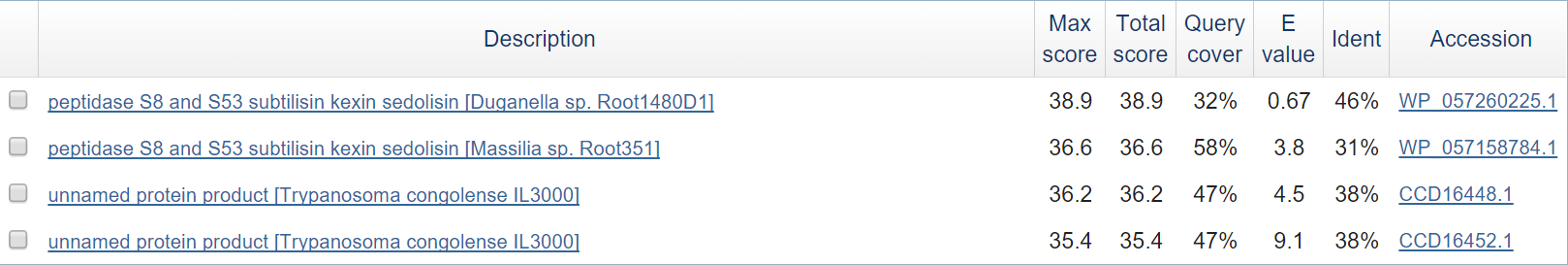
О гомологах речи, видимо, нет.
- Подобная ситуация и с генами 2,3,4,7 (совсем нет находок)
- Ген 8
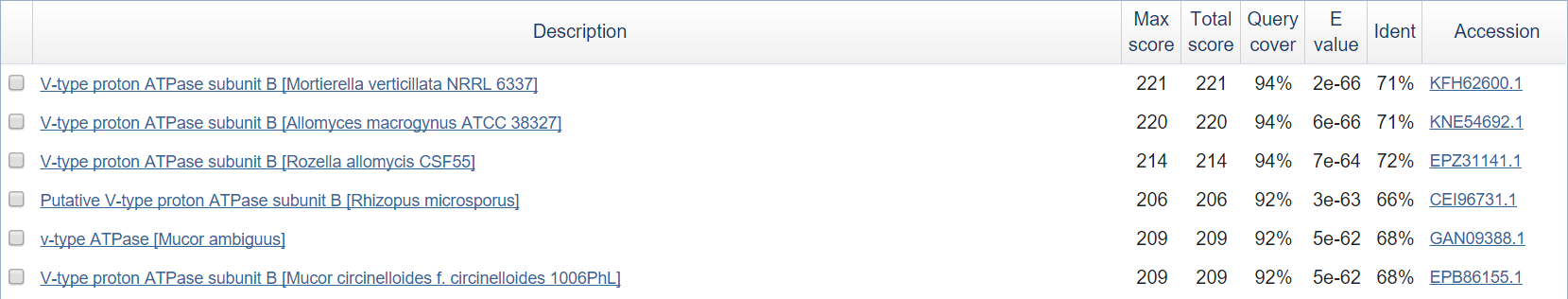
Восьмой ген кодирует субъединицу АТФазы. Сходство и Query cover высокие - находка, скорее всего, верная.
Экзоны не все найдены, т.к гомологичные белки гораздо длиннее (~500 aa) и имеют не меньше 5 экзонов.
- Ген 9
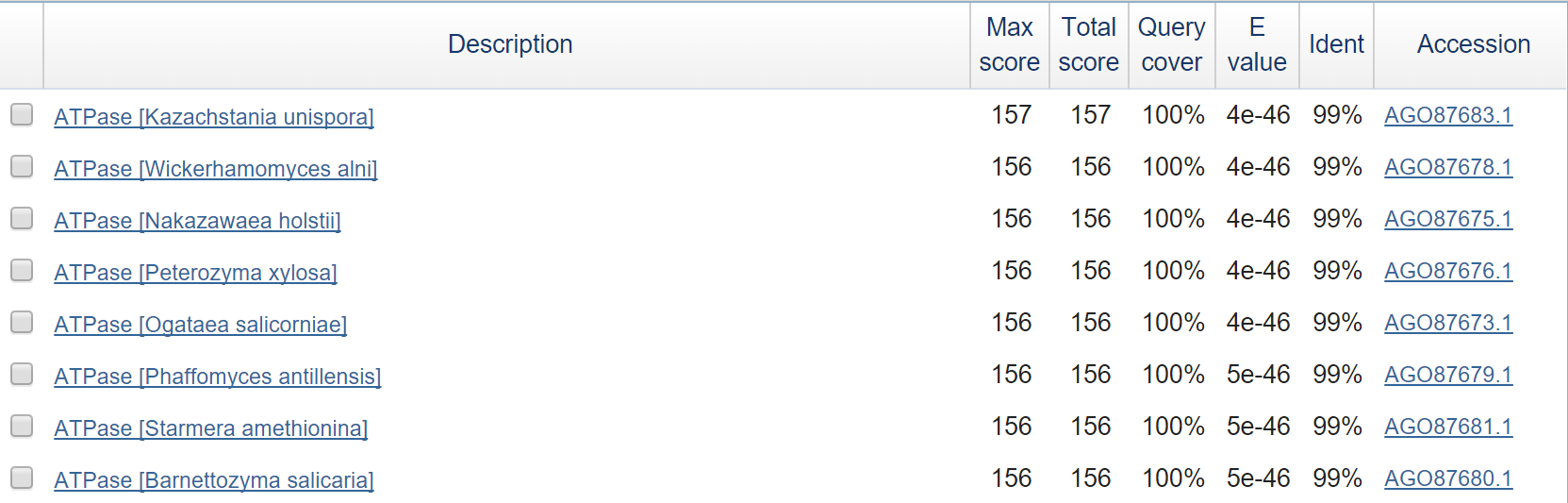
Девятый ген также кодирует часть субъединицы АТФазы. Сходство и Query cover очень высокие - находка верная.
- Ген 10
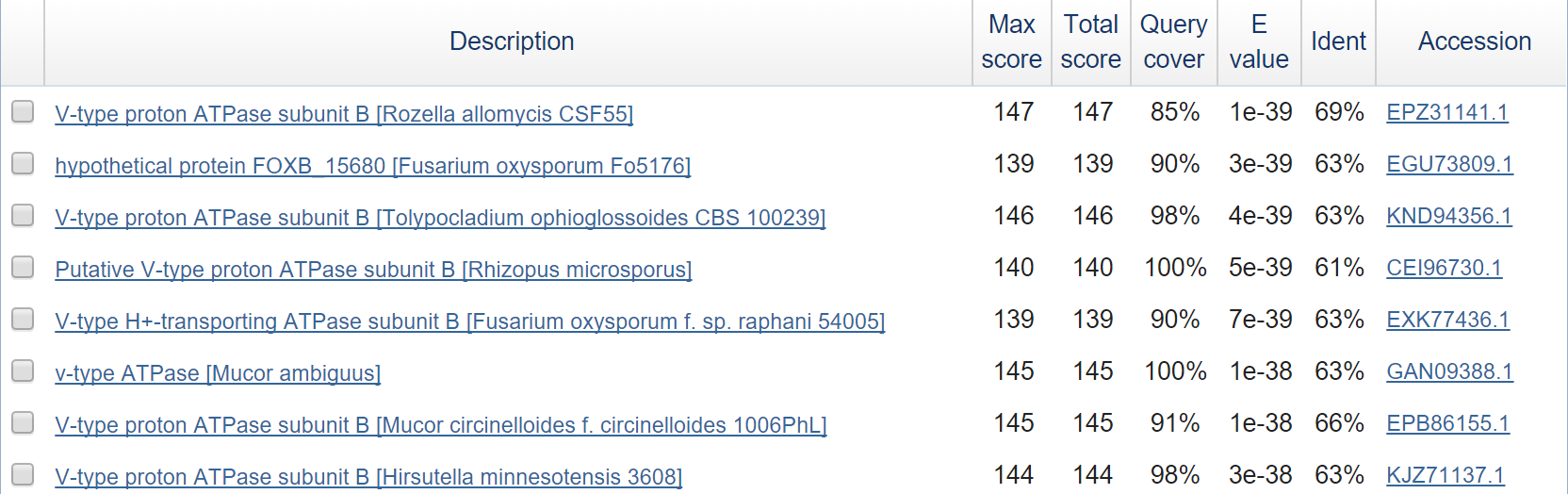
Десятый ген также кодирует часть субъединицы АТФазы. Сходство и Query cover очень высокие - находка верная.
Скорее всего, в 8-10 предсказанниях закодированы разные экзоны гена АТФазы. Однако точно есть не найденные экзоны, т.к. они вместе взятые не покрывают ген АТФазы.
- Ген 12:
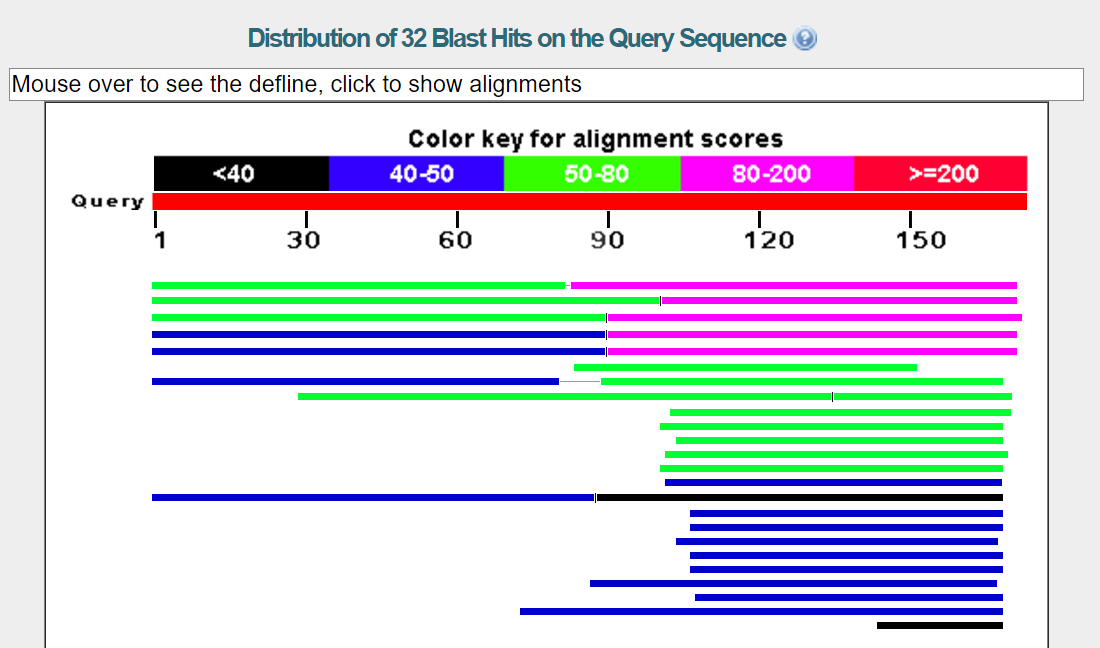
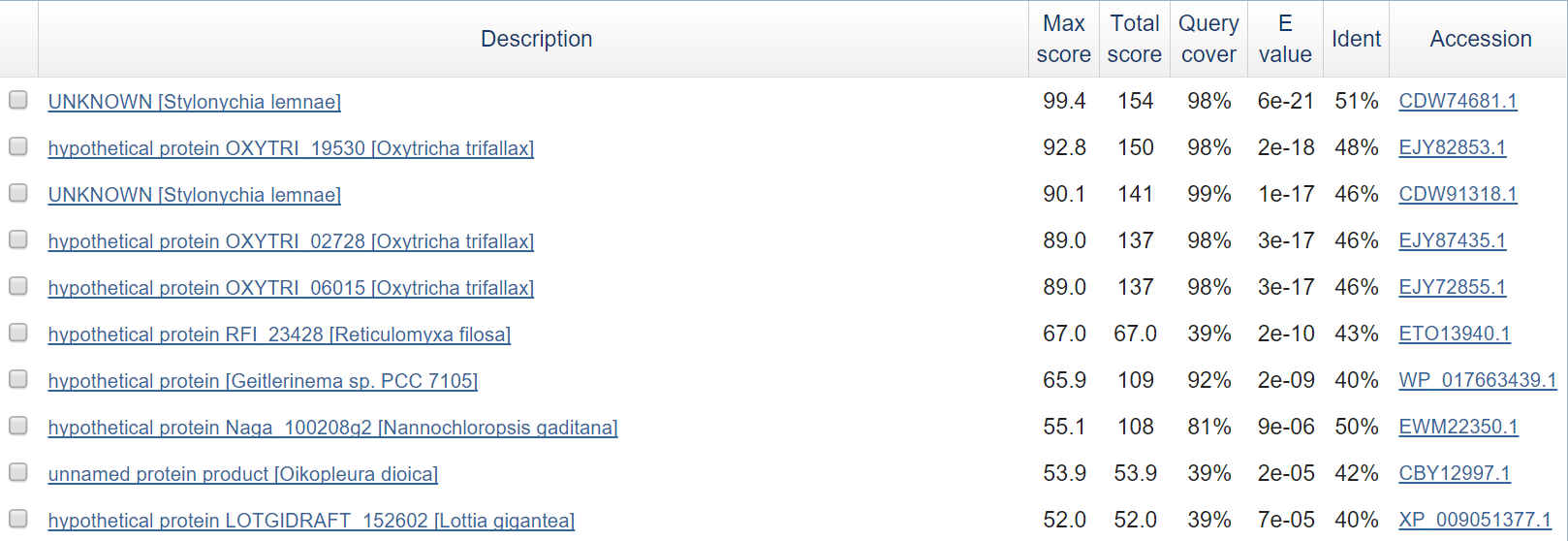 Ген 12 кодирует белок, функция которого неизвестна.
Ген 12 кодирует белок, функция которого неизвестна.
Скорее всего, находки соответствуют двум экзонам 12 гена. Однако у гипотетических белков экзоны вообще не указаны, из чего можно предположить, что предсказание неверно.
Сравнение аннотаций Refseq и AUGUSTUS гена DDIAS (Homo sapiens DNA damage-induced apoptosis suppressor)
Сравнение аннотаций в таблице (экзон - интронная структура)
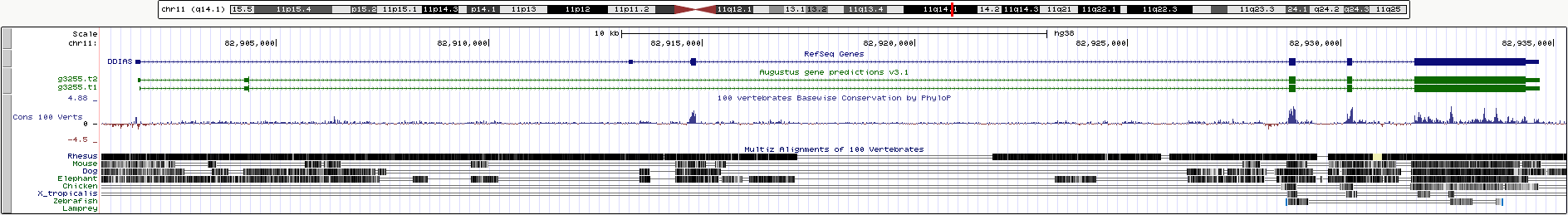
Ген DDIAS расположен на 11 хромосоме, его координаты chr11:82901695-82934657, ориентация +
Как видно из таблицы и визуализации Genome Browser, AUGUSTUS выдал две очень похожие аннотации - в обеих нашел "лишний" второй экзон, не нашел 2 экзона в середине, немного ошибся с началом гена (укоротил) и с его концом (удлинил).
Моя главная страница
© Sergey Starikov, 2015





 Ген 12 кодирует белок, функция которого неизвестна.
Ген 12 кодирует белок, функция которого неизвестна. 