Последовательности получены из файлов .frn с ftp NCBI. Так был создан файл
Выравнивал с помощью JalView
Дерево строил методом Neighbor-joining:
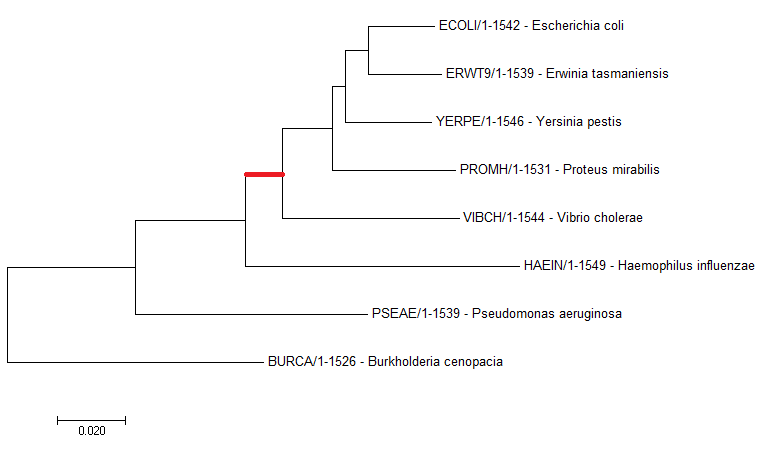
Правильное дерево:
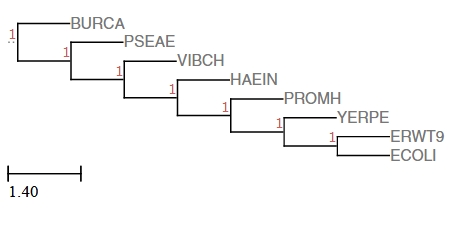
Полученное дерево не совпадает с правильным - отличается одной нетривиальной ветвью, выделенной красным цветом.
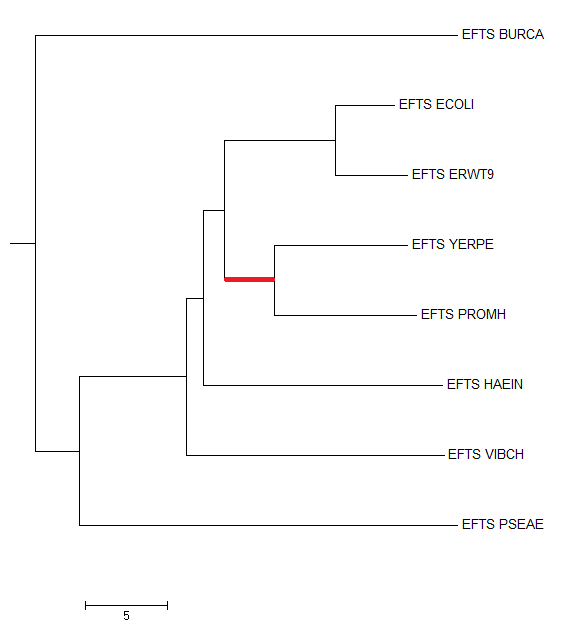
Дерево, построенное по белкам, также отличается от правильного одной нетривиальной ветвью (красная). Так что качество деревьев сравнимое.
Файл с протеомами
Результат работы blastp (E-value>0.001)
Выравнивание белков, полученных в результате работы blastp
Дерево полученных белков строил методом Neighbor-joining:
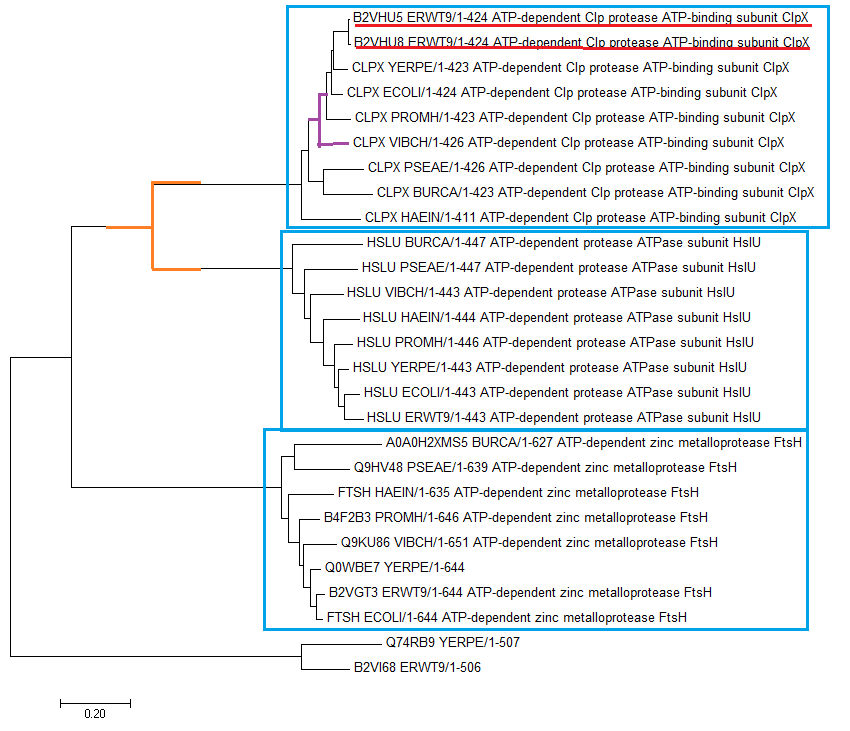
Группы попарно ортологичных белков - голубой цвет.
Пример паралогов - красный цвет.
Дупликация гена - оранжевый цвет.
Расхождение в ходе видообразования - фиолетовый.