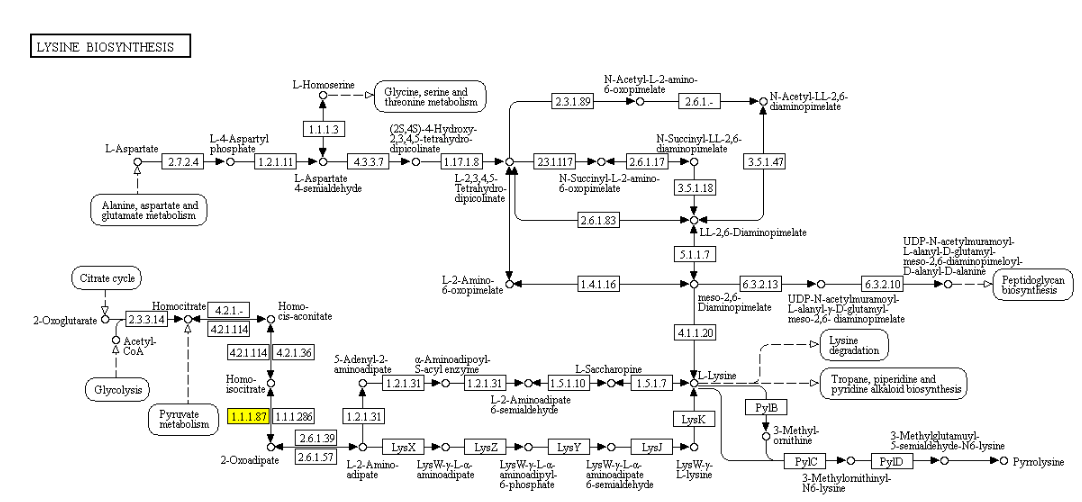
Для анализа выбран путь биосинтеза лизина (а в нем 1.1.1.87):
Ортологические ряды: K05824 (122 последовательности), K10978 (70 последовательностей)
Проект Jalview с множественным выравниванием fasta (alignment) и выравниванием после проверки (modified)
Отличное выравнивание получилось - внутри ортологичных рядов и между ними видна явная гомология (много блоков консервативных, почти у всех последовательностей внутри блоков нет гэпов). После проверки пришлось удалить всего 2 последовательности: A5DKS4_PICGU|K05824 и A5DKS5_PICGU|K05824 - уж больно короткие, с кучей гэпов внутри блоков. "Плохих", совсем плохо выровненных последовательностей не неаблюдал.
Для построения дерева использовалось множественное выравнивание
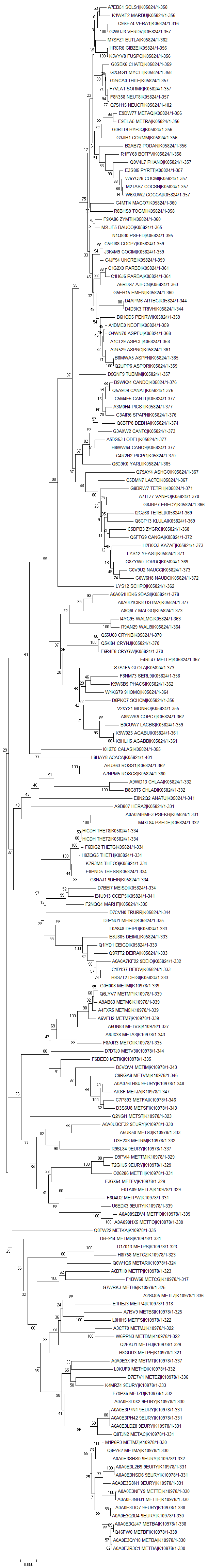
Рис. 1. Филогенетическое дерево (NJ) для белков, катализирующих выбранную реакцию.
Дерево действительно распадается на клады, соответствующие ортологическим рядам, это поддерживается 29 репликами бутстрэпа. Нет тривиальных ветвей с резко отличающимися длинами. Результат поддерживает ортологичность рядов в БД KEGG.