Практикум 15
В этом практикуме мы делали сборку de novo бактерии Buchnera aphidicola str. Tuc7. Для этого я воспользовалась ридами c кодом доступа SRR4240359.
Для того, чтобы их скачать я воспользовалась командой:
wget ftp://ftp.sra.ebi.ac.uk/vol1/fastq/SRR424/009/SRR4240359/SRR4240359.fastq.gz
Триммирование
Для начала я триммировала риды с помощью программы trimmomatic. Для этого я подготовила файл со всеми адаптерами, которые были в папке /mnt/scratch/NGS/adapters с помощью команды:
cat /mnt/scratch/NGS/adapters/* > adapters.fasta
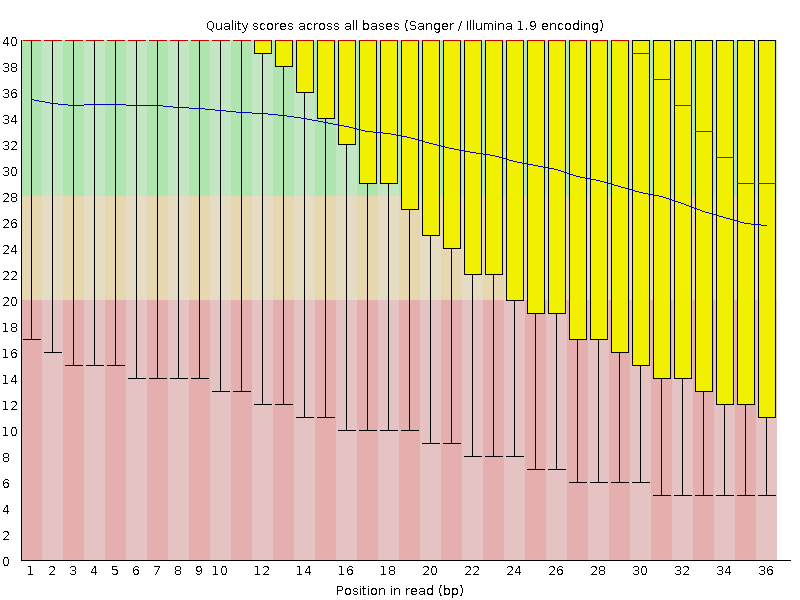
Перед тем как удалить остатки адаптеров, я посмотрела качесво ридов при помощи программы fastqc:
fastqc SRR4240359.fastq.gz
(Риды плохие, надо триммировать)
Вот такое качество получилось:
После чего запустила программу trimmomatic с следующими параметрами:
java -jar /usr/share/java/trimmomatic.jar SE -phred33 SRR4240359.fastq.gz SRR4240359_-_adapters.fq.gz ILLUMINACLIP:adapters.fasta:2:7:7
В результате 0.41% (55872 штук) последовательностей оказались остатками адаптеров.
После этого я удалила риды, у которых качество с конца было меньше 20 и длина которых меньше 32 пн:
java -jar /usr/share/java/trimmomatic.jar SE -phred33 SRR4240359_-_adapters.fq.gz SRR4240359_trimmed.fq.gz TRAILING:20 MINLEN:32
На этом этапе было отброшено 9.76% ридов (1317986 штук). После этого я снова проверила качество ридов. Вот что получилось:
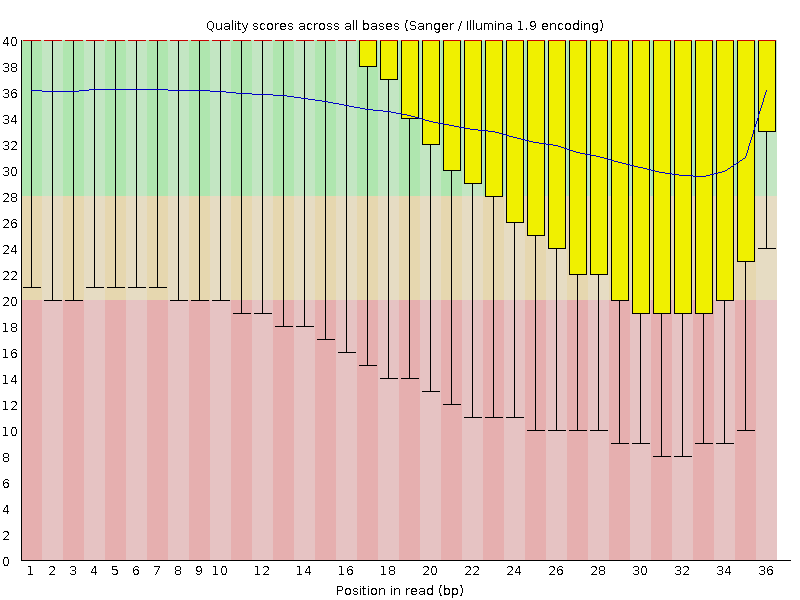
До чистки файл весил 445 Мб а после — 385 Мб (файлы я не распаковывала, ибо fastqc и trimmomatic умеют с таким работать). Всего было удалено 1373858 ридов.
Подготовка k-меров
Для того, чтобы собрать геном de novo, используя алгоритм, который использует граф де Брёйна, нужно подготовить k-меры из ридов. Для того, чтобы получить k-меры я использовала команду:
velveth SRR4240359_k_mers 31 -short -fastq.gz SRR4240359_trimmed.fq.gz
Длина k-меров — 31;
Чтения короткие непарные, параметр — -short;
Формат входного файла с чтениями fastq.gz, параметр — -fastq.gz;
SRR4240359_k_mers — папка в которой окажутся k-меры.
Сборка de novo
Дальше, для сборки de novo из получившихся k-меров, я запустила программу velvetg:
velvetg SRR4240359_k_mers
SRR4240359_k_mers — директория с k-мерами, получившаяся после работы программы velveth.
Из лог-файла, я узнала, что N50 = 70607, а из файла со статистикой ( используя команду sort -nk 2 stats.txt ), что самые длинные контиги имеют размеры 71403, 108447, 125674 пн,
а их покрытия составляют 39.4, 42.0, и 42.56 соответственно.
Есть аномальные контиги, например с покрытием 411220 и длиной 1 или с покрытием 1 и длиной 1 (таких контигов получилось несколько).
Анализ
Далее с помощью megablast я посмотрела куда в референсном геноме ложаться самые большие контиги моей сборки, информацию об этом я поместила в таблицу ниже.
| ID | Длина контига | Покрытие контига | Количество выравниваний | Координаты участка хромосомы | Средний % идентичных нуклеотидов | Средний % гэпов | Сколько % контига суммарно выровнялось |
|---|---|---|---|---|---|---|---|
| 1 | 125674 | 42.56 | 10 | 126623:127815, 127825:140555, 144368:151796, 153752:161738, 161898:166752, 166750:173180, 187938:192665, 192777:193984, 194042:196061, 198467:199381 | 79% | 1.8% | 46% |
| 11 | 108447 | 42.0 | 19 | 11103:2004, 14465:13994, 17919:14727, 20182:17962, 22183:20358, 28363:23067, 44693:35124, 46776:44768, 55420:47158, 59462:64632, 73310:70970, 86404:86174, 93683:88200, 94696:93821, 611524:611229, 611633:613671, 613658:620926, 621055:627104 | 78.6% | 2.5% | 60% |
| 14 | 71403 | 39.4 | 11 | 202390:207661, 209294:212243, 215717:218384, 219625:223720, 224057:228137, 228944:232057, 232358:236859, 248967:252161, 253223:257546, 260224:263784, 266073:273028, | 77.7% | 2.4% | 64% |
Контиги выравнились на геном с разрывами. Чем обусловлены эти "дырки" между выровненными кусками контигов? Есть вероятность, что в этих местах контиг плохо собрался из-за повторов, например. Еще одна возможность: геном бактерий достаточно изменчив. Вот почему например два конига выровнялись на обратную цепь и один на прямую? Я бы предположила, что у данного штамма произошла инверсия. Вообще то, что контиги легли на геном не целиком напрягает, есть вероятность, что сборка прошла неправильно.