Практикум 10
Выбраный домен
Я выбрала домен T-box. Этот домен связывается с ДНК и обеспечивает регуляцию развития той или иной ткани.
Мотивы T-box
Затем я скачала выравнивание seed. Исходно в выравнивание было 87 последовательностей. После удаление практически идентичных последовательностей (90% и выше) их осталось 84.
Из литературы [1] я узнала, что аминокислоты, которые непосредственно распознают нуклеотидную последовательность, находятся далеко друг от друга в последовательности домена, кроме того
они не были одинаковыми во всех последовательностях, вероятно, распознаваемый мотив ДНК немного отличается от белка к белку.
Каких-то известных мотивов я не нашла для данного домена, поэтому просто покрасила аминокилоты с высокой идентичностью (96% и выше) в выравнивание и выбрала мотив с наиболее высоким IC.

Паттерн мотива (синтаксис Jalview):F.[AVTS]V[TCS].Y
По этому паттерну затем был произведен поиск во всем выравнивание, всего находок получилось 84. Столько же, сколько и последовательностей, значит это мотив с хорошим IC.
Затем я произвела поиск данного мотива в базе данных Prosite.
Паттерн в формате Prosite:F-x-[AVTS]-V-[TCS]-x-Y
Всего находок получилось 708, во всех них паттерн встретился один раз. Паттерн оказался не уникален для домена T-box, в выравнивание было много последовательностей, не содержащих домен.
Затем последовательности были выровнены при помощи mafft. Мотив выравнялся с гэпами в большинстве последовательностей, что ожидаемо, поскольку выравнивание содержит множество последовательностей
не содержащих T-box домена.
Мотив специфичный для клады
В Jalview было построено выравнивание алгоритмом NJ. Затем я взяла кладу, выделенную красным на картинке:
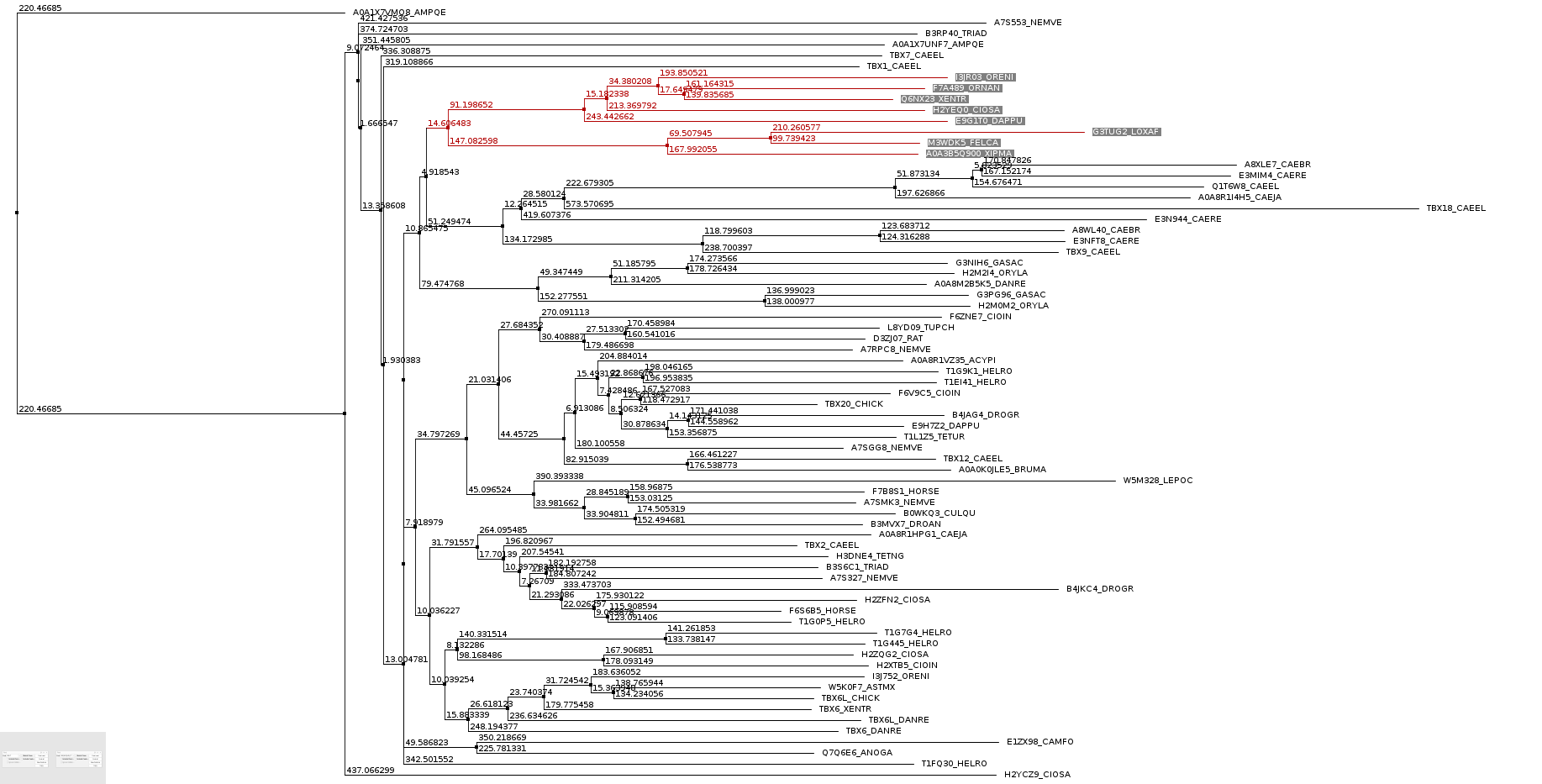
Я выделила следующий мотив: F[KPTQ]ET[RQ]FIAVTAYQN, который был во всех белках и не встречался больше ни в каком месте последовательности. Оказалось что данный мотив встречается только
в этой кладе, следовательно данный мотив является для нее специфичным.

PSI-BLAST
Выбраный AC: Q7VDL2
Этотбелок ингибирует клеточное деление, блокируя образование Z-колец за счет предотвращения полимеризации FtsZ.
Белок принадлежит бактерии Prochlorococcus marinus (strain SARG / CCMP1375 / SS120)
Итерации произвдились до стабилизации результатов. На 5 итерации новых результатов не появилось, разниа E-value между надпороговой и подпороговой находками также значительно увеличилось
Семейство (MinC) выделилось хорошо, однако на 4 итерации добавился один белок из другого семейства.
| № итерации | Число находок выше порога (0,005) | Идентификатор худшей находки выше порога | E-value этой находки | Идентификатор лучшей находки ниже порога | E-value этой находки |
|---|---|---|---|---|---|
| 1 | 146 | Q9AG20.1 | 0.005 | A8GFG7.1 | 0.005 |
| 2 | 188 | B6JKX0.1 | 7e-08 | - | - |
| 3 | 188 | Q9ZM51.1 | 2e-12 | A7H8E6.1 | 0.014 |
| 4 | 189 | A8MHK8.1 | 0.001 | A7H8E6.1 | 0.013 |
| 5 | 189 | A8MHK8.1 | 4e-10 | A7H8E6.1 | 0.009 |
Литература
1. Wilson, V., Conlon, F.L. The T-box family. Genome Biol 3, reviews3008.1 (2002). https://doi.org/10.1186/gb-2002-3-6-reviews3008