Практикум 11
HMM-профиль
Отбор последовательностей
Для создания профиля я выбрала подсемейство белков, содержащих два домена: T-box (PF00907) и TBX (PF12598). Подсеейство представлено белком O73717.
Затем я скачала fasta-файл с последовательностями подсемейства (всего 53 последовательности) и выровняла их с помощью программы mafft.
Затем два выровненных домена я сохранила в отдельный fasta-файл, после чего убрала подозрительные (имеющие много гэпов в местах доменов) и высокосхожие последовательности.
Финальный файл содержит 39 последовательностей.
Далее, для формирования калибровочной выборки я добавила к своему подсемейству еще одну архитектуру, содержащую домены: T-box и T-box_assoc (PF16176). В данном подсеемйстве 1821 белков, оно представлено белком P79944.
Калибровочная выборка
Построение профиля
Затем использовав пакет HMMER. Чтобы построить профиль я использовала команду
hmm2build -f t_box_tbx.hmm seed_final.fa
Чтобы уточнить некоторые константы модели, была проведена калибровка при помощи команды:
hmm2calibrate --cpu 1 t_box_tbx.hmm
Файл с профилем
Поиск по профилю
Затем я произвела поиск среди исходных последовательностей при помощи команды:
hmm2search --cpu 1 -T 200 t_box_tbx.hmm all.fasta > hmm_search.txt
Файл с находками
Результаты поиска в таблице.
Далее для обработки данных я использовала следующий код. (там же можно посмотреть суммарную таблицу, где отмечена принадлежность к позитивному/негативному контролю и seed)
Оказалось, что нашлись исключительно последовательности из подсемейства (52 штуки, те все кроме одной). Распределение весов для обучающей выборки и всего позитивного контроля:
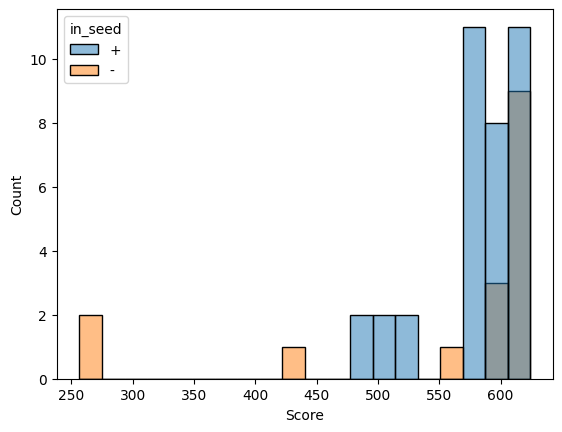
AUC ROC, очевидно, равен 1, поэтому кривую не прилагаю. Соответственно профиль идеально находит данную двудоменную архитектуру, однако, вероятно это связано с тем,
что подсемейство маленькое и для построение профиля я выкинула относительно немного последовательностей, скорее всего наблюдаемый результат — переобучение.