Практикум 4
Составление списка гомологичных белков
Протеомы бактерий из предыдущего практикума были собраны в один файл db.fa при помощи команды cat. Затем этот файл я проиндексировала:
makeblastdb -in db.fa -dbtype prot -parse_seqids
Затем я нашла гомологи белка CLPX_ECOLI в сформированной базе данных с помощью команды:
blastp -query clpx_ecoli.fa -db db.fa -evalue 0.001 -out clpx_out.txt
Вот такая выдача получлась:
Score E
Sequences producing significant alignments: (Bits) Value
P0A6H1 ATP-dependent Clp protease ATP-binding subunit ClpX OS=Esc... 860 0.0
Q8ZC66 ATP-dependent Clp protease ATP-binding subunit ClpX OS=Yer... 805 0.0
B4EU54 ATP-dependent Clp protease ATP-binding subunit ClpX OS=Pro... 769 0.0
Q21KA8 ATP-dependent Clp protease ATP-binding subunit ClpX OS=Sac... 645 0.0
Q5P160 ATP-dependent Clp protease ATP-binding subunit ClpX OS=Aro... 619 0.0
Q8UFY5 ATP-dependent Clp protease ATP-binding subunit ClpX OS=Agr... 596 0.0
Q165G0 ATP-dependent Clp protease ATP-binding subunit ClpX OS=Ros... 582 0.0
Q21H71 ATP-dependent protease ATPase subunit HslU OS=Saccharophag... 99.8 7e-23
B4F171 ATP-dependent protease ATPase subunit HslU OS=Proteus mira... 96.7 9e-22
Q8ZJJ5 ATP-dependent protease ATPase subunit HslU OS=Yersinia pes... 95.1 2e-21
P0A6H5 ATP-dependent protease ATPase subunit HslU OS=Escherichia ... 93.6 7e-21
Q16CY2 ATP-dependent protease ATPase subunit HslU OS=Roseobacter ... 93.6 9e-21
Q5P503 ATP-dependent protease ATPase subunit HslU OS=Aromatoleum ... 93.6 9e-21
Q8UJ87 ATP-dependent protease ATPase subunit HslU OS=Agrobacteriu... 92.0 2e-20
A0A5P8YGZ0 ATP-dependent Clp protease ATP-binding subunit OS=Yers... 51.2 5e-07
B4EV83 ATP-dependent Clp protease ATP-binding subunit OS=Proteus ... 50.1 1e-06
B4F2B3 ATP-dependent zinc metalloprotease FtsH OS=Proteus mirabil... 46.6 2e-05
P0AAI3 ATP-dependent zinc metalloprotease FtsH OS=Escherichia col... 46.2 2e-05
A0A5P8YB42 ATP-dependent protease OS=Yersinia pestis OX=632 GN=YP... 46.2 2e-05
A0A5P8YCE6 ATP-dependent zinc metalloprotease FtsH OS=Yersinia pe... 45.8 3e-05
Q168A2 Holliday junction branch migration complex subunit RuvB OS... 45.4 3e-05
Q7CT50 ATP-dependent zinc metalloprotease FtsH OS=Agrobacterium f... 45.4 4e-05
P0ABH9 ATP-dependent Clp protease ATP-binding subunit ClpA OS=Esc... 43.1 2e-04
Q16C81 Chaperone protein ClpB OS=Roseobacter denitrificans (strai... 41.2 8e-04
Q167Z2 ATP-dependent zinc metalloprotease FtsH OS=Roseobacter den... 41.2 8e-04
Всего 25 находок, все они являются АТФазами, и, почти все протеазами.
Реконструкция и визуализация
Находки затем были объединены в один fasta файл при помощи нехитрых команд:
blastp -query clpx_ecoli.fa -db db.fa -evalue 0.001 -out file.txt -outfmt '6 sseqid'
awk '!seen[$0]++' file.txt
seqtk subseq db.fa file.txt
awk -F "|" '/^>/{split($3, a, " "); print ">" a[1]; next} {print}' file.fasta
Дерево в Newick формате
| Ортологи | Паралоги |
|---|---|
| HSLU ECOLI и HSLU YERPE | A0A5P8YB42 YERPE и A0A5P8YGZ0 YERPE |
| CLPX ROSDO и CLPX AGRFC | CLPX AGRFC и HSLU AGRFC |
| CLPX YERPE и CLPX ECOLI | B4EV83 PROMH и B4F2B3 PROMH |
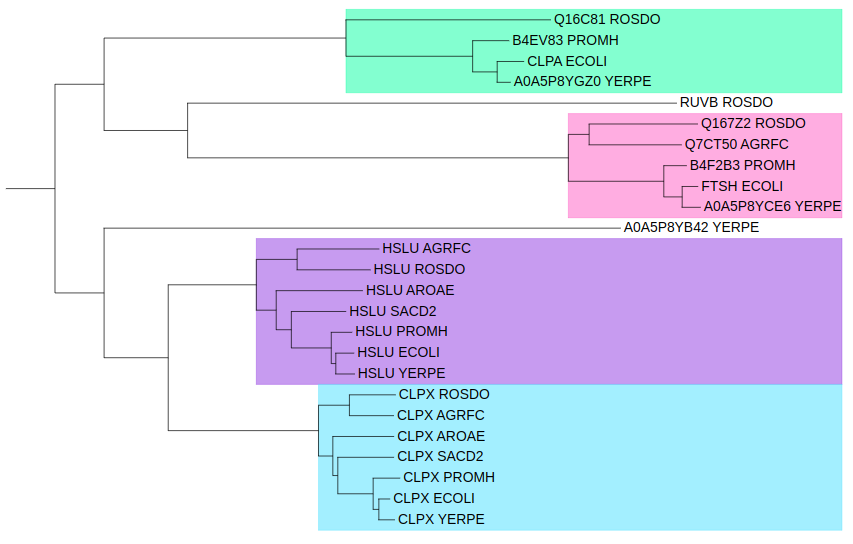
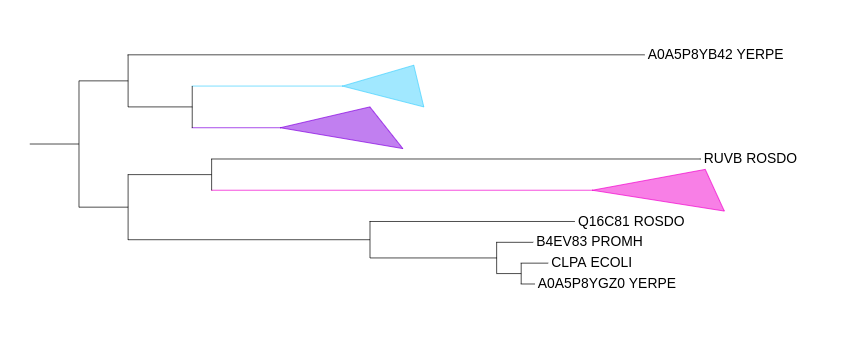
Члены розовой клады — металлопротеазы FtsH, а зеленой — субъединицы Clp протеазнго комплекса (но не ClpX). Обе клады также соответвуют филогении бактерий. Особняком стоят белок RuvB из Aromatoleum и некая АТФ-зависимая протеаза из чумной палочки.