Практикум 6
Сравнение предсказаний трансмембранных участков в бета-листовом белке
Для работы я выбрала аэролизин (aerolysin).
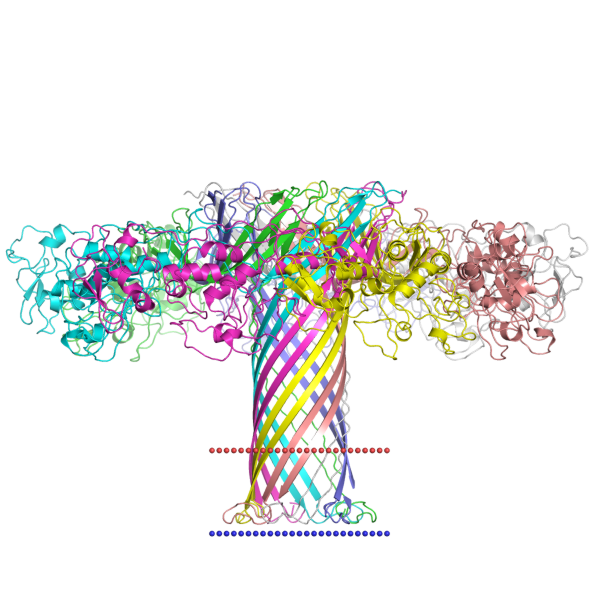
Идентификатор PDB: 5jzt
Идентификатор UniProt: AERA_AERHY
Данный белок был выделен из грамотрицательной бактерии Aeromonas hydrophila, обитающей в водной среде. Аэролизин — токсин данной бактерии, после удаления C-концевого домена, он
встраивается в мембрану клетки хозяина, вызывая её гибель.
Координаты трансмембранных участков белка: 238-245 и 254-259 в каждом из 7 сегментов.
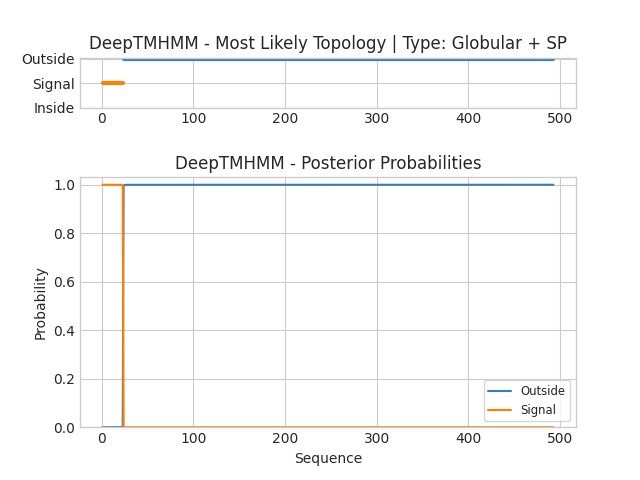
Затем я загрузила в DeepTMHMM последовательность аэролизина с сайта UniProt. Программа предсказала сигнальный участок и внешний участок, те секретируемый белок, который никак не заякорен
в мембране. Это произошло потому, что одиночный бета-лист не может находиться в мембране, только в группе с другими, образуя бета-боченок. Потом я попробовала загрузить все 7 сегментов одним fasta-файлом,
но трюк не удался.
Текстовая выдача
Сравнение предсказаний трансмембранных участков в альфа-спиральном белке
Мой белок: Y2029_ALISL (Putative transport protein VSAL_I2029)
Это потенциальный переносчик калия из бактерии Aliivibrio salmonicida.
Для данного мелка была запущена программа DeepTMHMM. Координаты предсказанных трансмембранных спиралей: 14-28, 37-53, 64-82, 92-112, 164-184, 386-403, 416-429, 447-463, 478-498,
539-559.
Текстовая выдача
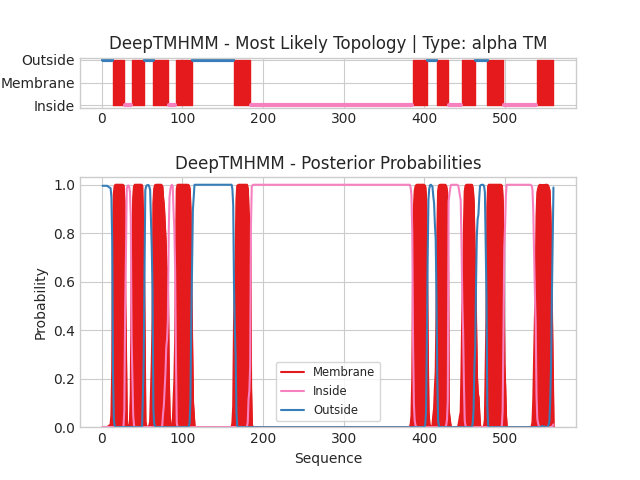
Количество мембран: 1
Положение N-конца: снаружи (по графику, приведенному выше)
Тип мембраны: внутренняя мембрана грамотрицательной бактерии
Предсказанные координаты: 14-34, 37-57, 66-86, 94-114, 161-181, 386-406, 409-429, 451-471, 478-498, 539-559.
Количество трансмембранных участков, предсказанных обоими методами совпадает, однако координаты немного разные. Если посмотреть на достоверность AlphaFold модели, то можно отметить,
что в последних двух трансмембранных альфа спиралях она уверена, в вот в предшествующей им спирали нет. Предсказания для двух моделей согласуются для последних двух спиралей,
а для предшествующей уже нет. Похожие наблюдения можно сделать и для других трансмембранных участков. Вероятно, достоверность модели могла оказать влияние на качество предсказания алгоритмом PPM.