Практикум 9
Подготовка данных
Для поиска сигнала de novo я выбрала сигнал посадки сигма-субъединицы РНК-полимеразы бактерий.
Для работы я выбрала бактерию Ideonella dechloratans (родственница Ideonella sakaiensis, разлагающей пластик:>). Сначала я скачала геномную последовательность, которую загрузила в сервис Operon-mapper, чтобы получить список оперонов.
Опероны доступны тут. Затем я использовала замечательный ноутбук Серёжи Бушуева для того,
чтобы получить три выборки промоторов: обучающую, тестовую и негативный контроль(участки ДНК, не пересекающиеся с промоторами).
MEME
Затем я запустила Meme локально на сервере kodomo:
meme train.fa -dna -nmotifs 3 -minw 6 -maxw 50
В результате было получено три мотива:

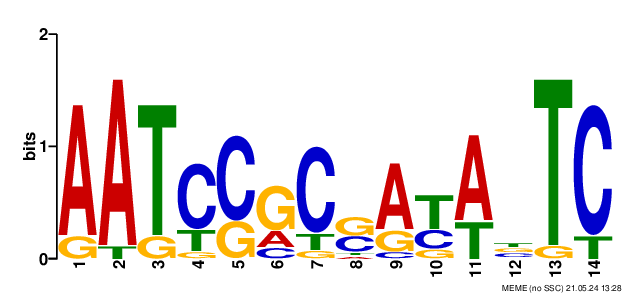

FIMO
Для поиска первого мотива в положительном и отрицательном контролях были запущены следующие команды:
fimo --norc -motif RCCBTTDTGCTAHAVTVCSCACCCYTTT -thresh 0.01 ./meme_out/meme.txt test.fa
Выдача для положительного контроля
fimo --norc -motif RCCBTTDTGCTAHAVTVCSCACCCYTTT -thresh 0.01 ./meme_out/meme.txt neg_contr.fa
Выдача для отрицательного контроля
Для положительного контроля нашлось 25 последовательностей из 50 (50%), а для негативного контроля 30 из 50 (60%). Как это можно прокомментировать? Мотив нашелся очень плохо, я пробовала
Увеличить обучающую выборку, добавляя ключевые слова для генов домашнего хозяйства, E-value уменьшилось, но результат все равно плохой.