Выравнивание геномов.
Для тренировки выравнивания геномов с помощью программы blast2seq я взяла два штамма бактерии:
Escherichia coli IAI39 и Escherichia coli str. K-12 substr. MG1655
E.coli - представитель грам-отрицательных бактерий, является широко распрастраненным модельным организмом.
Штамм К-12 является наиболее изученным для данного вида.
К данным организмам применяется широкий набор генетических манипуляций. На модели E.coli можно синтезировать нужные для эксперимента
белки (например GFP-Lamin A), либо белки нужные в комерческих целях и для медицины (производство инсулина).
Кроме синтеза продуктов, на модели бактерии можно изучать функции некоторых генов направлено выключая, увеличивая или наоборот снижая урвоень их экспресси.
При построении выравнивания двух последовательностей я использовала blast2sec, в основе которого лежит
алгоритм blastn, которому я дополнительно задала паратемтры E=1e-10.
Число находок: 5670
Максимальная длина находки:86581
Max.score:1.482e+0.5
Identity(для всего выравнивания):98%
Таблица с полной информацией по данному выравниванию.
Визуализация выранивания - карта локального сходства, на которой сразу видны крупные геномные перестройки между этими двумя организмами.
По оси абсцисс штамм IAI39, по оси ординат К-12
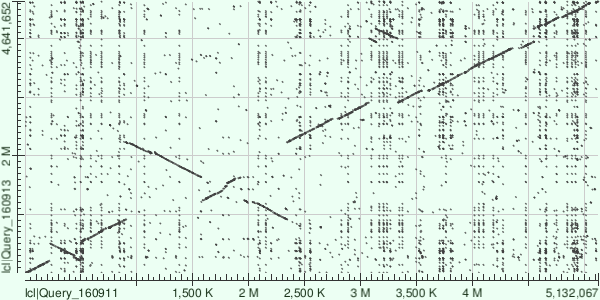
Рис.1
На полученной карте мы видим следующие перстройки:
- Инверсия пар оснований (по первому штамму) 0.25М - 0.5М.
- Инверсия пар оснований 0.9М - 1.6М
- Инверсия пар оснований 1.95М -2.35М
- Инверсия пар оснований 3.1М - 3.4М
Интеренсо отметить: инверсии 2 и 3 наталктивают на мысль, что сначала произошла инверсия целиком
участка 0.9М - 2.35М и позже еще раз участок 1.6М 1.95М изменил свое положение. Я делаю такие выводы,
потому что эти инверсии располагаются на данной карте на одной прямой, а участок соответствующей прямой последовательности расположен чуть ниже, то есть как бы
выбивается из диагонали.
На участке, соответствующем 4-ой инверсии, происходит еще транслокация.
|


