Анализ хроматограмм, полученных после секвенирования по Сэнгеру.
Редактирование хроматограмм.
Для анализа были взяты следующие последовательности: последовательность1, последовательность2.
В прогармме Chromas вручную были отредактированы обе последовательности - последовательность 1 и с переводом в комплиментаруную ей последовательность 2.
При поиске ошибок в определении нуклеотидов чаще всего встречалось наложение пиков, в таком случае решалось, являются ли какие-то из возникающих пиков шумом или мы имеем дело с племорфизмом.
Вот некоторые трудные моменты в хроматограммах:
Первая последовательность:
- Пик нуклеотида А в позиции 29 явно выделяется над фотом, поэтому я посчитала правильным вставить его в последовательность.
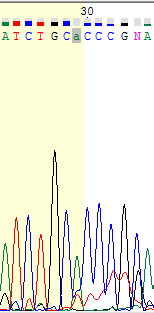
- Пик нуклеотида G растянут на две позиции 88 и 89, но в позиции 88 пик нуклеотида А выше и явно отличим от шума.
- В позициях 83 и 85 возможно явление полиморфизма, т.к. один из пиков в обоих случаях
выделяется сильнее, но второй достаточно высок относительно фона.
В целом хроматограмма этой последовательности довольно хорошая - шум присутствует, но его фон достаточно стабилен на протяжении всей последовательности (высота шумовых пиков составляет около 1/5 от высоты нормальных пиков) и незначительно возрастает только под конец. Высота всех пиков возрастает ближе к концу.
С 5' конца последовательности понадобилось
удалить 50 нуклеотидов. Широкие пики, затрагивающие неколько позиций и затрудняющие интерпретацию хроматограммы, здесь практически не встречаются.
Вторая последовательность:
- В позиции 142 практически слиты пики двух нуклеотидов. Скорее всего это полиморфизм.
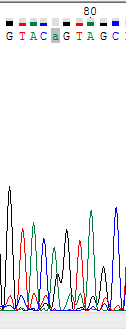
- В позиции 163 пик нуклеотида Т выделяется довольно явно, но пик С нельзя однозначно назвать шумом,
т.к. он выше соседнего пика, который учитывается в последовательности.
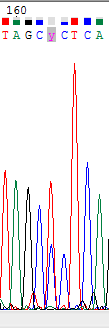
- Пик нуклеотида Т, который читывается в позиции 297, "расползсь" на несколько позиций, загородив собой соседние пики,
которые имеют достаточно большую высоту. Поэтому я отмечаю их в последовательности на соответствующих позициях.
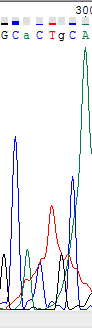
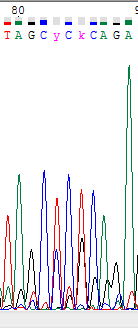
Вторая хроматограмма хуще первой - в ней чаще встречаются широкие пики, занимающие несколько позиций.
В районе позиции 300 выделяются резкие скачки пиков двух нуклеотидов, возможно в этом месте произошел какой-то
сбой в работе детектора сигнала. Фон шума также составляет около 1/5 от основного сигнала. Высота пиков увеличивается незначительно к концу хроматограммы.
С 5' конца удален 51 нуклеотид нечитаемой последовательности.
Полученные после редактирования последовательности в формате FASTA: последовательность1,
последовательность2.
Выравнивание последовательностей.
Выравнивания выполнены программой needle из пакета EMBOSS. Далее полученные данные импортированы в JalView
для удобства изучения. Все измененные вручную нуклеотиды напечатаны строчными буквами. Ссылка на проект выравнивания.
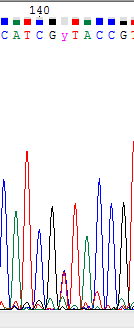
Нечитаемый фрагмент хроматограммы.
Нечитаемый фрагмент хроматограммы вязт из данного файла.
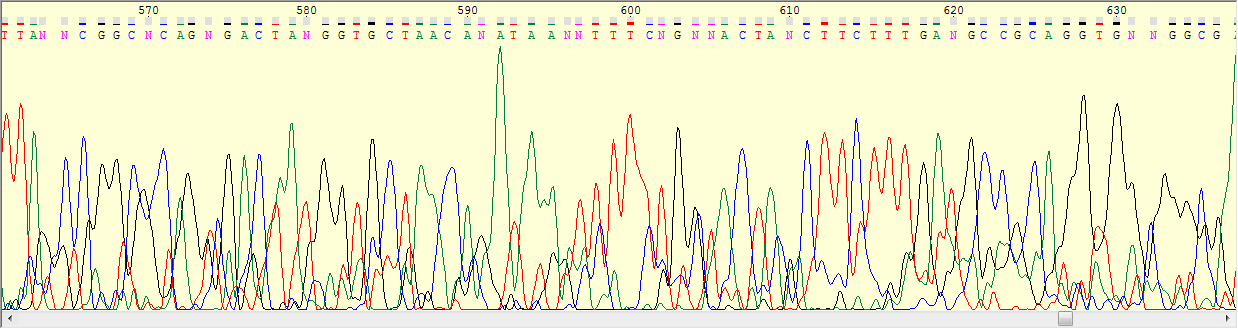
Интерпретация такого фрагмента очень затруднена поскольку присутствует множество находящих друг на друга пиков,
изза большой высоты которых невозможно точно сказать, какие из них являются фоновым шумом.
Очень часто встречаются широкие пики, контур которых растягивается на многие позиции. Некотыре пики резко выдаются по высоте на фоне окружения.
Кроме того сами пики не правильной конусовидной формы, а со скошенными краями, будто при образовании еще одной вершины.
Возможно при определении данной последовательности в образец попали в большом количестве фрагменты другой ДНК.
Возможно также через детектор проходили флюорофоры в большом количестве, т.е. не только связанные с нуклеотидами, что может объяснить
резкие высокие или растянутые пики.
|


