Поиск эволюционных доменов.
Для поиска мотивов в программе МЕМЕ было предложено выбрать штамм бактерии и отобрать у него 8-10 генов,
участвующих в биосинтере пуринов (поиск проводился в Uniprot по ключевому
слову "Purine biosynthesis").
Далее работа будет проводиться с: Escherichia coli UTI89 (ECOUT), для которой найдено 8 аннотированных записей по keyword "Purine biosynthesis".
AC записи EMBL, описывающей геном: CP000243.
| Protein |
Gene |
Coordinates |
| Phosphoribosylformylglycinamidine synthase |
purL |
complement(2833402..2837445) |
| Formate-dependent phosphoribosylglycinamide formyltransferase |
purT |
1957569..1958747 |
| Adenylosuccinate synthetase |
purA |
4676499..4677797 |
| Bifunctional protein FolD |
folD |
complement(567893..568759) |
| GMP synthase [glutamine-hydrolyzing] |
guaA |
complement(2766782..2768359) |
| Bifunctional purine biosynthesis protein PurH |
purH |
3698818..3700407 |
| HTH-type transcriptional repressor PurR |
purR |
1773602..1774627 |
| Phosphoribosylformylglycinamidine cyclo-ligase |
purM |
2757020..2758057 |
По координатам генов, взятых из файла с полным геномом были определены координаты 100 нуклеотидов,
предшествующих самим генам. Программой deascseq последовательности перед генами были переданы в один файл
prot.fasta.
На сервере kodomo была запущена программа ememe с параметрами -nmotifs 3 и -revcomp. Данные параметры
позволяют искать заданное число мотивов и производить поиск на прямой и обратной цепях. В рамках данной
задачи это необходимо, так как гены некоторых белков расположены на комплиментарной цепи.
Результат выдачи МЕМЕ meme.html.
Теперь разберем некоторые полученные результаты.
Для каждого найденного мотива было выдано LOGO -
диаграмма в которой высота каждого столбца предполагаемого мотива равна информационному содержанию данного столбца,
а высота отдельной буквы столбца - вероятность встечи этой буквы умноженная на информационное содержание данной позиции. Иными словами -
данная диаграмма показывает как вероятнее всего (в большинстве случаев) выглядит искомый мотив.
Для каждого мотива выдано егог Инвормационное содержание - критерий того, насколько целесообразно считать данную находку мотивом.
При грубом подсчете слово длины I/2 встретиться в геноме один раз на 4^(I/2) пар нуклеотидов.
Кроме того, для каждого мотива посчитано E-value - математическое ожидание - показатель того, сколько раз находки с таким же или большим весом встречаются в
последовательностях.
 |
- E-value = 2.3
- I= 22.2 bit
- 6 последовательностей содержат мотив
- Удивительно, что E-value больше единицы, что свидетельствует в пользу плохой находки мотива,
при этом информационное содержание высокое и по logo видно, что находится 8 консервативных позиций. При этом
p-value каждой находки низкое. Для некоторых последовательностей найденный мотив похож на сайт посадки рибосомы (например в purM).
|
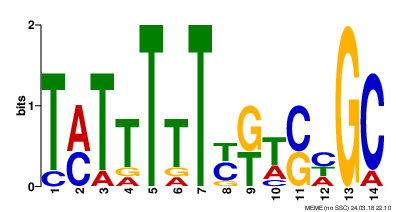 |
- E-value = 1.4*10^3
- I= 16.6 bit
- 7 последовательностей содержат мотив
|
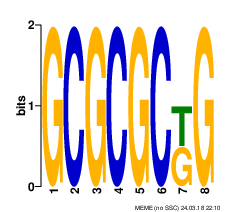 |
- E-value = 7.5*10^3
- I= 15.0 bit
- 2 последовательности содержат мотив
|
|

