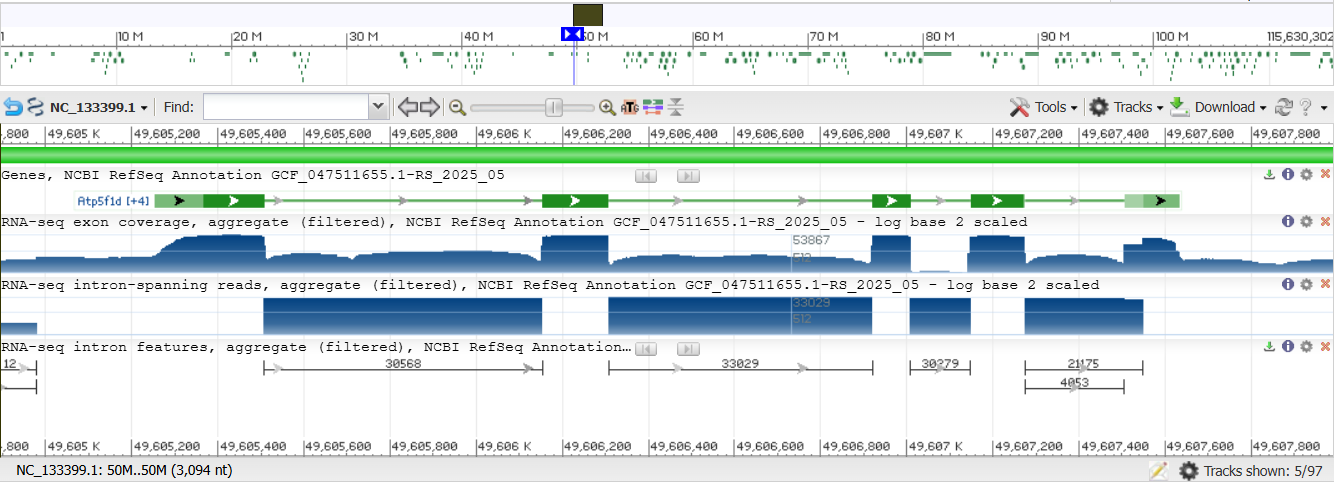
 Рис.1. Ген Atp5f1d и его окрестность. Координаты кодирующей части: 49605253 - 49607632.
Рис.1. Ген Atp5f1d и его окрестность. Координаты кодирующей части: 49605253 - 49607632.
С помощью текстового поиска в файле с последовательностями белков Castor canadensis был найден идентификатор последоательности белка дельта-субъединицы АТФ синтазы - XP_020026319.1. Идентификатор нуклеотидной записи соответствующего гена - NC_133399.
 Рис.1. Ген Atp5f1d и его окрестность. Координаты кодирующей части: 49605253 - 49607632.
Рис.1. Ген Atp5f1d и его окрестность. Координаты кодирующей части: 49605253 - 49607632.
Для поиска по последовательностям геномов был выбран таксон Araneae. В качестве базы данных использовалась refseq_genomes. Поиск проводился двумя методами: blastn и tblastx. Алгоритм megablast на использовался, т. к. нужен для поиска почти идентичных последовательностей. В blastn все параметры выравнивания я оставил по умолчанию, за исключением длинны слова: выставил на 7.
Алгоритм blastn не нашел схожих последовательностей. Ввиду особенностей работы алгоритма такой результат ожидаем: происходит сравнение нуклеотидных последовательностей, количество находок в таком случае если и будет, то очень низким из-за вырожденности генетического кода.
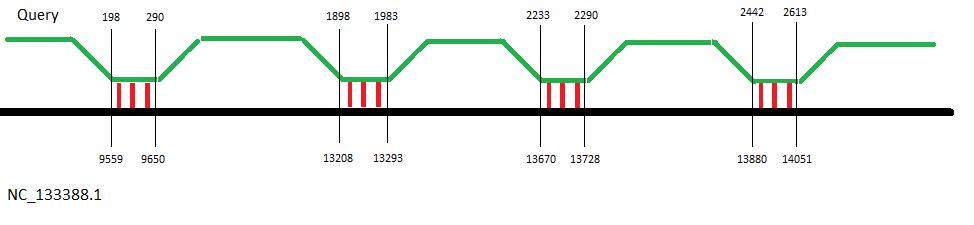
Алгоритм tblastx обнаружил 14 находок: происходит сравнение не нуклеотидных последовательностей, а результатов их трансляции.
 Рис.2. Выдача tblastx.
Рис.2. Выдача tblastx.
Для поиска гомологов последовательности генома Castor canadensis были проиндексированы с помощью локального BLAST:
makeblastdb -in "C:\FBB_PC\3_sem\Bio_inf\pr7\Genome_ncbi_dataset\ncbi_dataset\data\GCF_047511655.1\GCF_047511655.1_mCasCan1.hap1v2_genomic.fna" -dbtype nucl -out BOBR_db
Поиска гомологов 16s и 23s рРНК у E. coli по базе данных Castor canaedensis производился с помощь алгоритма blastn:
blastn -task blastn -query "C:\FBB_PC\3_sem\Bio_inf\pr8\Ecloi_pr8.txt" -db BOBR_db -out 16s_pr8.out -evalue 0.01 -outfmt 7
Поиск гомологов для 16s рРНК дал 41 результат, содержащий 1 участок (1494 - 1536).
В случае 23s рРНК получилось столько же находок, совпавших участков получилось 4.
Для построения карты локального сходства были выбраны геномы бактерий Streptomyces albidoflavus strain S20 и Streptomyces sp. WZ-12.
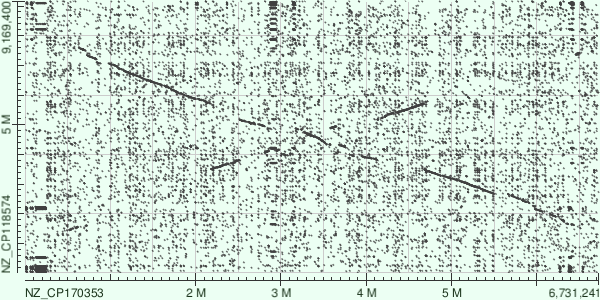
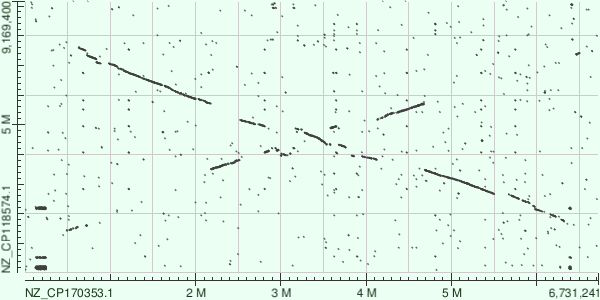
Как видно из рисунков 3 и 4, оба алгоритма обнаружили такие наиболее крупные события как две инверсии с транслокациями (в центре), длинные индели (тоже в центре), а также blastn обнаружил множество коротких сходств (дупликации?).
При попытке применить алгоритм tblastx выдается ошибка.