-изолейцин-тРНК
-изолейцин-тРНКсинтетаза.
Для исследования была выбрана цепь T, представляющая изолейциновую тРНК со следующей последовательностью:
[1] 5' -GGGCUUGUAGCUCAGGUGGUUAGAGCGCACCCCUGAUAAGGGUGAGGUCGGUGGUUCAAGUCCACUCAGGCCCAC- 3' [74],
где 1 и 74 - номера первого и последнего нуклеотида.В последовательности на 3'-конце отсутствует триплет CCA, взяимодействующий с аминокислотным остатком.
-акцепторный стебель состоит из участка 1-7 и комплементарного ему участка 66-72.
-Т-стебель состоит из участка 49-53 и комплементарного ему участка 61-65.
-D-стебель состоит из участка 10-13 и комплементарного ему участка 22-25.
-антикодоновый стебель состоит из участка 26-32 и комплементарного ему 38-44.
На полученном изображении акцепторный стебель выделен красным, Т-стебель - зеленым, D-стебель - синим, антикодоновый - жёлтым.
Рис.1. Вторичная структура изолейциновой тРНК из Staphylococcus aureus: |
Скрипт для получения изображения: restrict none select all backbone 150 select 1-7 or 66-72 color red select 49-53 or 61-65 color green select 10-13 or 22-25 color blue select 26-32 or 38-44 color yellow |
Пример неканонической пары, поддерживающих структуру стеблевых дуплексов (G26- A44):
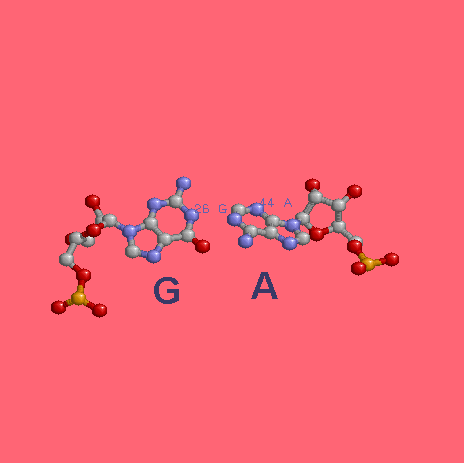
Также стоит отметить:
а)Oтсутствие вариабельной петли
б)Отсутствие остатка тимидина в Т-петле
в)Отсутствие дигидроуридинов в D-петле
В антикодоновой петле найден триплет, кодирующий изолейцин (A35-U-36-A37). На рис.1 эти нуклеотиды показаны в шарнирной модели.
В пользу стекинг-взаимодействия говорят данные о ненулевой площади перекрывния, равной 3.57 A^2, в выходном файле 1qu2_old.out .
Картинка, иллюстрирующая наложение оснований:
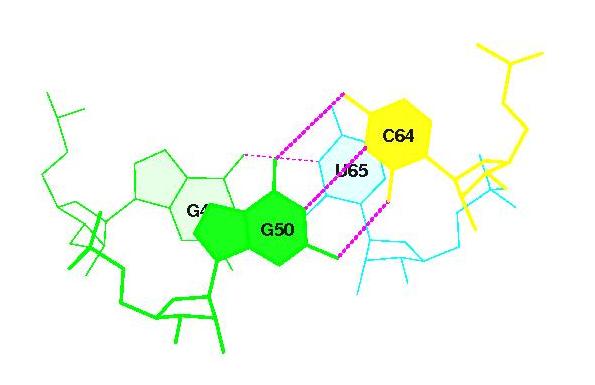
2. Дополнительные водородные связи между основаниями D- и Т-петель отсутствуют
Реальная и предсказанная вторичная структура тРНК из файла 1qu2.pdb
| Участок структуры |
Позиции в структуре (по результатам find_pair) |
Результаты предсказания с помощью einverted |
Результаты предсказания по алгоритму Зукера |
| Акцепторный стебель | 5' 1-7 3' 5' 66-72 3' Всего 7 пар |
предсказано 6 пар | предсказано 7 пар |
| D-стебель | 5' 10-13 3' 5' 22-25 3' Всего 4 пары |
предсказано 0 пар | предсказано 4 пары |
| T-стебель | 5' 49-53 3' 5' 61-65 3' Всего 5 пар |
предсказано 0 пар | предсказано 5 пар |
| Антикодоновый стебель | 5' 26-32 3' 5' 38-44 3' Всего 7 пар |
предсказано 2 пары | предсказано 5 пар |
| Общее число канонических пар нуклеотидов | 23 | 8 | 21 |
Программа einverted наилучший результат выдаёт при параметрах: Gap penalty = 6, minimum score threshold = 1. Изменение параметров Match score и Mismatch score не приводят к новым значимым результатам. При снижении Gap panalty увеличивается число гэпов, не приводящее к появлению новых стеблей, при увеличении - снижается число найденных стеблей. Для увеличения количества найденных водородных связей параметр Minimum score threshold выбран наименьшим.
Программа mfold выдаёт самую удачную картинку при заданном параметре Р=15, при увеличении параметра структура всё меньше похожа на канонический вид тРНК, уменьшение параметра приводит к изображению всего одного стебля.
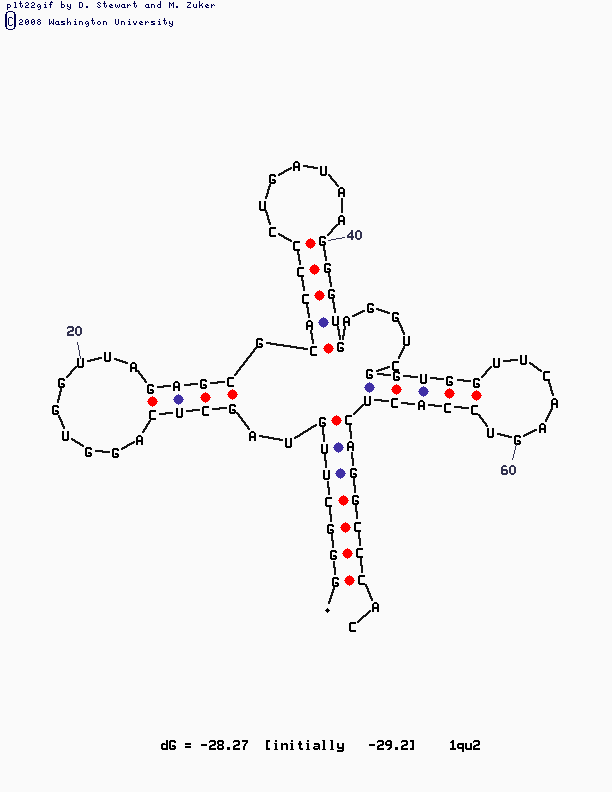
Вывод: Программа einverted хуже справляется с предсказанием вторичной структуры ДНК, находя только акцепторный и антикодоновый стебель (будто представляет структуру тРНК в виде двух шпилек); для данной задачи лучше использовать алгоритм Зукера. Программа mfold способна предсказать структуру с максимальной точностью и предоставить пользователю на выбор тот визуализированный вариант, который по его мнению наиболее точно отвечает реальной структуре.