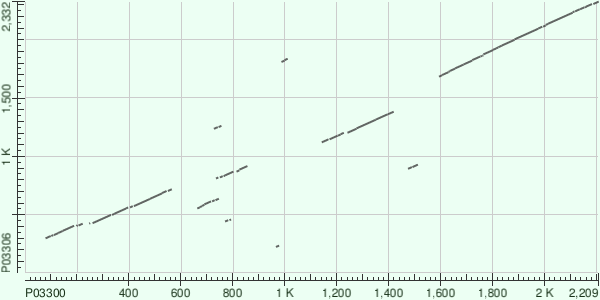
Карта локального сходства 2 полипротеиновКарта локального сходства геномных полипротеинов Poliovirus type 1 (strain Mahoney) (по оси абсцисс) и Foot-and-mouth disease virus (strain A10-61) (Aphthovirus A) (по оси ординат):
Характеристики 2 лучших блоков локального выравнивания:
* 1 - относится к вирусу полиомиелита, 2 - относится к вирусу ящура | ||||||||||||||||||||||||||||||||||||||||||||||||||||
Сравнение веса выравнивания со случайным
Оценка локального выравнивания предположительно гомологичных факторов сопряжения транскрипции и репарации Escherichia coli и Bacillus subtilis:
Оценка локального выравнивания предположительно негомологичных декарбоксилирующей 6-фосфоглюконатдегидрогеназы Escherichia coli и пантотенаткиназы Bacillus subtilis:
Проверка формулы для перевода веса в биты Формула B = 1 + (S - M)/(Q1 - M) даёт оценку (через p-value) случайности появления выравнивания данного веса. Соответсвенно, если вес исходного выравнивания составляет уровень верхней 1/8 (O1) весов любого количества случайных выравниваний, то вероятность получить случайное выравнивание с таким же или больше весом должна быть p = 1/8 = 2^(-3), значит, этот вес соответствует 3 битам. Проверим формулу для 1000 случайных последовательностей и веса гипотетического локального выравнивания (исходной последовательности со специально подобранной случайной последовательностью), равному уровню верхней 1/8 весов локальных выравниваний исходной последовательности с этой 1000 случайных последовательностей:
Полученный счёт в битах заметно отличается от теоретического значения, хотя p-value = 16.6%, не так уж сильно отличается от теоретического p = 1/8 = 12.5% - формула для перевода веса выравнивания в биты даёт погрешность, но всё же достаточно точная (особенно, если учитывать то, что p-value позволяет дать лишь приблизительную оценку гомологичности белков, и часто важен только порядок его значения). Методы: Последовательности были перемешаны программой shuffleseq. Локальное выравнивание исходной последовательности с перемешанными осуществлялось программой water с параметрами по умолчанию. Сортированный список весов выравниваний со случайными последовательностями составлялся конвейером grep "Score" [input_alignments.fasta] | sed -r "s/[^.0-9]//g" | sort -g > [score_list.scores]. Нужные веса извлекались из списка программой sed по номеру строки. Все промежуточные файлы лежат в директории ~stepan_puhov/term2/block3/pr10. | ||||||||||||||||||||||||||||||||||||||||||||||||||||
BLAST: поиск гомологов в банке2 белка из банка Swiss_Prot, наиболее сходных с предположительной аминометилтрансферазой Pseudomonas putida strain ATCC 47054 (Name: Putative Aminomethyltransferase; UniProt AC: Q88LI8):
Замечание: Исходный белок (Query) принадлежит Pseudomonas putida, относящейся к классу Gammaproteobacteria отдела Proteobacteria группы дидермных ("грамотрицательных") бактерий. Однако среди представленных 50 находок первые 36 являются аминометилтрансферазами монодермных ("грамположительных") бактерий (в основном отдел Firmicutes, реже отдел Thermotogae и отдел Actinobacteria), почти для всех этих находок процент идентичности около 22% и довольно большое покрытие исходного белка - около 93%. Только с 37-находки начинают появляться аминометилтрансферазы дидермных бактерий (в основном отделы Cyanobacteria и Proteobacteria, а также отдел Spirochaetea), также присутствуют регуляторная суб'единица митохондриальной фосфатазы пируват дегидрогеназы 3 видов позвоночных (предки митохондрий относятся к классу Alphaproteobacteria отдела Proteobacteria), тем не менее хотя процент идентичности этих находок даже несколько больше, покрытие исходного белка значительно уменьшается: менее 73%, в среднем около 55-60%, E-value также больше такового для белков монодермных бактерий, начиная с 9e-08 и достигает значений порядка единиц. Можно предположить, что фермент псевдомонады имеет больше гомолгичных доменов с аминометилтрансферазами монодермных бактерий, чем с ферментами протеобактерий, и, возможно, был получен от монодермных бактерий путём горизонтального переноса генов. | ||||||||||||||||||||||||||||||||||||||||||||||||||||
| Главная страница |