|
Описание дерева
|
Отобранные виды бактерий
| Название вида | Мнемоника |
| Bacillus anthracis | BACAN |
| Clostridium tetani | CLOTE |
| Enterococcus faecalis | ENTFA |
| Geobacillus kaustophilus | GEOKA |
| Lactobacillus acidophilus | LACAC |
| Listeria monocytogenes (serovar 1/2a) | LISMO |
| Moorella thermoacetica | MOOTA |
| Staphylococcus aureus | STAAR |
|
|
Скобочная формула дерева
Дерево отобранных бактерий, полученное по заданному дереву,
в Newick формате:
((CLOTE,MOOTA),((ENTFA,LACAC),(STAAR,(LISMO,(BACAN,GEOKA)))));
|
|
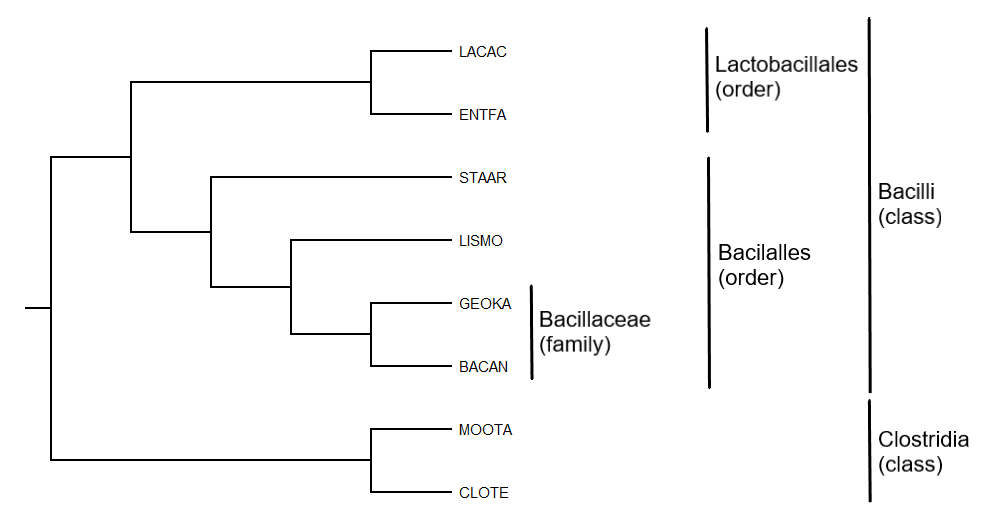
Изображение дерева
Изображение построено программой MEGA по записи в Newick формате
и далее отредактировано программой Paint.
Указанная таксономия соответствует таксономии NCBI:
|
|
Ветви дерева
Неукоренённую топологию полученного дерева можно задать следующим списком
разбиений множества листьев дерева, где каждое разбиение соответствует
нетривиальной ветви неукоренённого дерева (список охватывает все
нетривиальные ветви дерева):
- {MOOTA, CLOTE} vs {LACAC, ENTFA, STAAR, LISMO, GEOKA, BACAN},
- {LACAC, ENTFA} vs {STAAR, LISMO, GEOKA, BACAN, MOOTA, CLOTE},
- {LACAC, ENTFA, MOOTA, CLOTE} vs {STAAR, LISMO, GEOKA, BACAN},
- {LACAC, ENTFA, STAAR, MOOTA, CLOTE} vs {LISMO, GEOCA, BACAN},
- {LACAC, ENTFA, STAAR, LISMO, MOOTA, CLOTE} vs {GEOKA, BACAN}.
Дерево имеет n = 8 листьев, и поэтому 2n - 3 = 13 ветвей для
неукоренённой топологии, из которых (2n - 3) - n = n - 3 = 5
нетривиальных − перечислены в списке.
Первая из них разделяет выбранных бактерий на классы Clostridia и Bacilli,
вторая ветвь выделяет порядок Lactobacillales, третья ветвь выделяет порядок
Bacillales, пятая нетривиальная ветвь выделяет семейство Bacillaceae. Для
клады, выделяемой четвёртой ветвью, в таксономии NCBI название отсутствует.
|
|
Реконструкция филогении
|
Поиск гомологов белка
CLPX_ECOLI
в выбранных бактериях
Белок CLPX_ECOLI имеет
сложную доменную структуру
— составлен 3 доменами. Его N- и С-концевые домены, видимо, специфичны
для семейства шаперонов CLP, тогда как центральный домен белка относится к
подсемейству Cdc48 очень обширного семейства AAA+ АТФаз.
Поиск гомологов этого белка в протеомах выбранных бактерий производился при
помощи локального blastp с порогом E-value = 1E-3, длиной
якорного слова = 6 aa и матрицей BLOSUM62.
В выдаче программы
находки можно разбить на 4 группы по длине выравнивания:
>400 aa (находка с самым коротким выравниванием),
[300; 400) aa (находка с самым коротким выравниванием),
[200; 300) aa (находка с самым коротким выравниванием)
и <200 aa за исключением выравниваний находок, имеющих другие
выравнивания в одной из предыдущих групп (находка с самым длинным выравниванием).
На граничных представителях этих групп показано (и верно для каждой из групп в
целом из-за похожих внутри групп паттернов покрытия выравнивания), что белки
первой группы имеют все 3 домена искомого белка, белки второй и третьей групп
обладают центральным и C-доменом искомого белка, а для длинных белков четвёртой
группы гомология с искомым белком выражается лишь наличием AAA+ АТФазного
домена. Гомологами CLPX_ECOLI считались белки первой, второй и третьей групп
(имеющие гомологичные центральный и C-концевой домены), что согласуется с
причислением их к суперсемейству Clp в UniProt.
Таким образом, было найдено 19 гомологов белка CLPX_ECOLI:
| Название вида | Мнемоника | UniProt ID гомолога |
| Bacillus anthracis | BACAN | CLPX_BACAN, HSLU_BACAN |
| Clostridium tetani | CLOTE | CLPX_CLOTE |
| Enterococcus faecalis | ENTFA | CLPX_ENTFA, HSLU_ENTFA |
| Geobacillus kaustophilus | GEOKA | CLPX_GEOKA, HSLU_GEOKA, Q5L436_GEOKA |
| Lactobacillus acidophilus | LACAC | Q5FKR6_LACAC, HSLU_LACAC, Q5FM98_LACAC |
| Listeria monocytogenes | LISMO | CLPX_LISMO, HSLU_LISMO, Q8Y8B1_LISMO |
| Moorella thermoacetica | MOOTA | CLPX_MOOTA, Q2RJP5_MOOTA |
| Staphylococcus aureus | STAA8 | CLPX_STAA8, HSLU_STAA8, CLPL_STAA8 |
|
|
Выравнивание и реконструкция филогении гомологов CLPX_ECOLI
Для выравнивания найденных последовательностей использовалась программа
MAFFT. Поскольку последовательности, исходя из выравниваний в выдаче
blastp, имеют один общий для всех них гомологичный участок, но
АТФазный домен имеет сильно вариабельную длину (т.е., видимо, имеются большие
неконсервативные инсерции), а также имеются протяжённые негомологичные участки
с N-концевой стороны, использовался алгоритм E-INS-I:
Команда:
mafft --ep 0 --genafpair --maxiterate 1000 --bl 45 proteins.fasta > proteins_einsi.msa
Результат: выравнивание
Для удаления неконсервативных участков было проведено триммирование
выравнивания программой BMGE с удалением колонок, содержащих более
30% гэпов:
Команда:
java -jar $BMGE -i proteins_einsi.msa -t AA -h 1 -g 0.3 -of proteins_einsi_gap30.msa
Результат: триммированное выравнивание
Филогенетическое дерево было построено по триммированному выравниванию
программой IQTREE2 методом ML с моделью LG+G4+C10 (модель LG+G4
подобрана автоматически в соответствии с Bayesian information criterion):
Команда:
iqtree2 -s proteins_einsi_gap30.msa -m LG+G4+C10
Результат: отчёт программы
Полученное дерево в Newick формате, переукоренённое по ветви, разделяющей
семейства ClpA/B и ClpX, и без указания длин ветвей:
ссылка
Примеры пары ортологов на дереве:
Q8Y8B1_LISMO и Q5L436_GEOKA,
HSLU_BACAN и Q2RJP5_MOOTA,
Q5FKR6_LACAC и CLPX_LISMO
Примеры пары паралогов на дереве:
Q8Y8B1_LISMO и HSLU_LISMO,
Q8Y8B1_LISMO и CLPX_LISMO,
HSLU_LISMO и CLPX_LISMO
Визуализация (проведена сервисом iTOL):
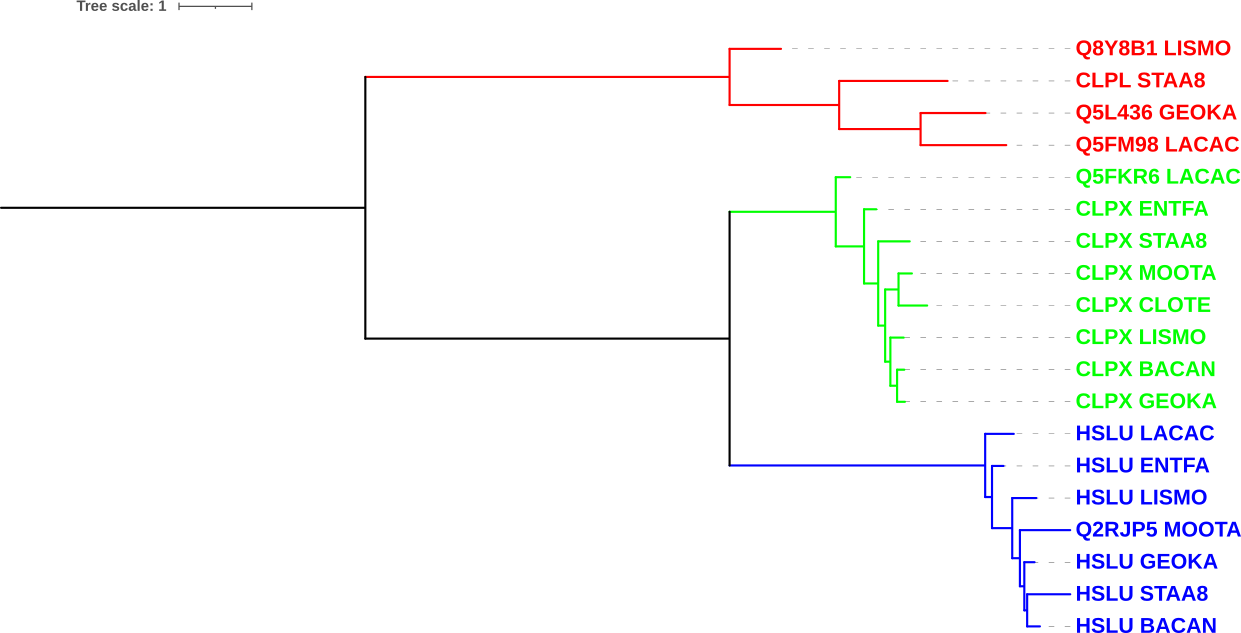 |  |
Раскраска по ортологичным группам:
красный — семейства ClpA/ClpB,
зелёный — семейство ClpX sensu stricto,
синий — семейство ClpX подсемейство HslU
|
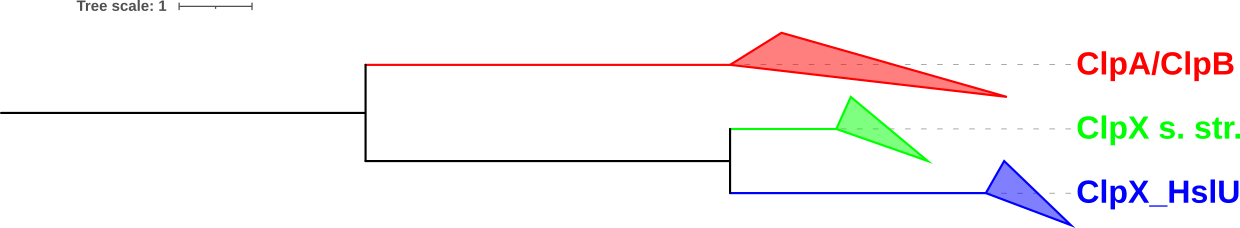
Ортологичные клады сжаты с сохранением раскраски:
ClpA/ClpB включает белки бактерий LISMO, STAA8, GEOKA и LACAC,
не совместимо с деревом видов;
ClpX s. str. включает белки всех 8 бактерий,
не совместимо с деревом видов;
ClpX_HslU включает белки всех выбранных бактерий, кроме CLOTE,
не совместимо с деревом видов
|
|
|
|
|