Мини-обзор генома бактерии Paenibacillus durus
Автор: Степанова Софья Михайловна
Факультет биоинженерии и биоинформатики, Московский Государственный Университет имени М.В.Ломоносова, Москва; stepanova.sofia@fbb.msu.ru
Резюме
Данная статья содержит краткий обзор генома и протеома Paenibacillus durus с использованием биоинформатического пакета EMBOSS, программирования на языке Python и функционала электронных таблиц. Описан геном бактерии, GС-состав плазмиды и хромосомы, нуклеотидный состав геномных ДНК, проанализированы длины белков, их распределение белок-кодирующих генов, представлены данные о рибосомальных, гипотетических и транспортных белках и данные об РНК бактерии.
1 Введение
Бактерия Paenibacillus durus выделенна из повы и корней различных растений [5].
Медицинское значение. Виды Paenibacillus описаны как "золотая жила кандидатов в антибиотики" [3]. Paenibacillus производят широкий спектр липопептидов, из которых можно разработать антибактериальные, противогрибковые, противораковые и противовирусные препаратов. Также известно, что развитие антибиотикорезистентности к липопептидам происходит гораздо медленнее. Помимо липопептидов, Paenibacillus также производят два из трех типов бактериоцинов [4]. А некоторые исследования показывают, что их экзополисахариды имеют потенциал в качестве антиоксидантов, противоопухолевых препаратов и, возможно, даже профилактических средств против кариеса.
Сельскохозяйственное значение. Многие виды производят противогрибковые препараты и инсектициды, которые защищают растения от патогенов и насекомых-травоядных[2]. Они также могут стимулировать собственные механизмы сопротивления растений. Некоторые виды производят ферменты, которые убивают личинок жуков.. Кроме того, известно, что они способствуют росту культур посредством фиксации азота, солюбилизации фосфатов и усвоения железа. Исследования показывают, что Paenibacillus могут быть использованы в качестве менее дорогого и более экологически чистого удобрения, чем многие из используемых сегодня химических фосфорных удобрений [1].
Промышленное значение. Хотя в настоящее время Paenibacillus не используется ни в одной отрасли промышленности, некоторые виды Paenibacillus производят ферменты, которые могут быть использованы в производстве моющих средств, биотоплива, бумаги, текстиля и продуктов питания [1]. Их потенциальная польза как более стабильного, более продуктивного и менее дорогого источника ферментов для промышленного применения остается неизученной.
2 Материалы и Методы
Данные бактерии взяты и с сайта NCBI [3]. Анализ данных проводился с помощью электронной таблицы Google Sheets [5], электронной таблицы Excel [8] и программы на языке Python [4]. Хромосомная таблица[1], последовательность генома [2].
3 Результаты
3.1 Описание генома бактерии
Геном Paenibacillus durus содержит безымянную кольцевую плазмиду(15151 п.н.) и одну хромосому(6038347 п.н.) [6]. В хромосоме GC составляет 50,8%, а в плазмиде 49,05%.| ДНК | Длина (п.н.) | GC состав |
|---|---|---|
| Хромосома | 6038347 | 50,8 % |
| Плазмида | 15151 | 49,05% |
В Таблице 2 представлен нуклеотидный состав ДНК. Можно заметить, что число адениловых оснований примерно равно числу тиминовых (1485270 и 1492462 соответственно), а число гуаниловых примерно равно цитозиновых (1545693 и 1530073 соответственно) в геноме, то есть выполняется второе правило Чаргаффа[7].
| ДНК | A | T | G | C |
|---|---|---|---|---|
| Хромосома | 148172 | 1488287 | 1542335 | 1525999 |
| Плазмида | 3544 | 4175 | 3358 | 4074 |
| Всего | 1485270 | 1492462 | 1545693 | 1530073 |
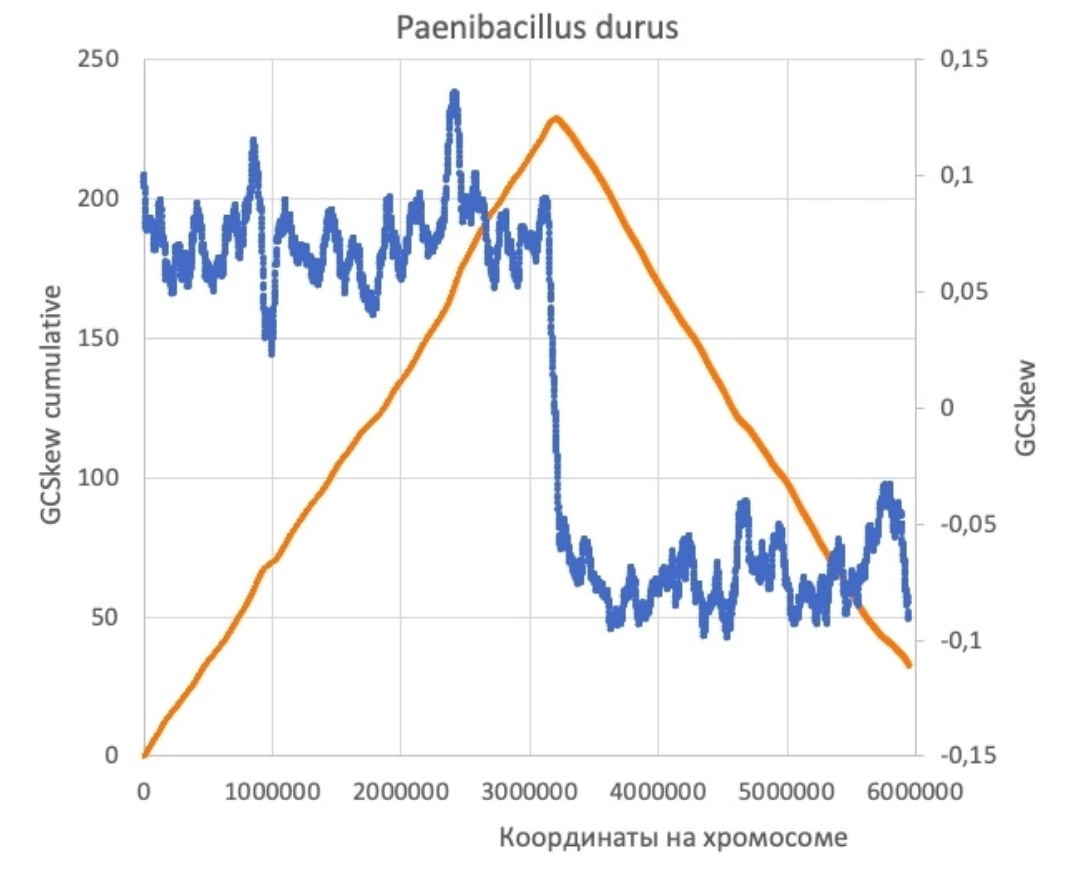
На рис. 1 представлен график cumulative GC-skew нуклеотидной последовательности хромосомы Paenibacillus durus . Известно, что минимум соответствует местоположению начальной точки репликации, а максимум - местоположению конечной точки репликации в прокариотическом геноме.
3.2 Статистические данные о белках протеома
Распределение длин белков показано на гистограмме на рис. 2.
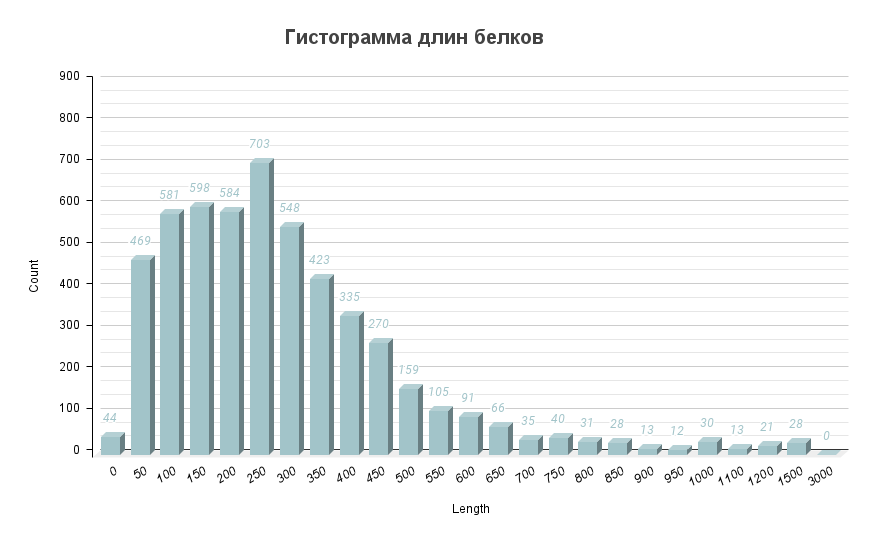
| Средняя длина | 326 |
|---|---|
| Среднее квадратичное отклонение | 314 |
| Медиана | 274 |
| Минимальное значение | 16 |
| Максимальное значение | 6649 |
С помощью электронных таблиц [8, таблица GC, лист “+и-”] было установлено распределение белок-кодирующих генов по цепям ДНК (Таблица 4). По статистической значимости мы можем сделать вывод, что гены по цепям распределяются случайно (уровень значимости >0.01).
| ДНК | «+» цепь | «-» цепь | Статистическая значимость | |||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Хромосома | 2516 | 2694 | 0,014191142 | |||||||||||||||||
| Плазмида | 0 | 29 | - | |||||||||||||||||
| Белки | Количество | Процент от всех белков |
|---|---|---|
| Рибосомальные белки | 60 | 2.13 % |
| Гипотетические белки | 839 | 29.72 % |
| Транспортные белки | 97 | 3.44 % |
| РНК | Количество | Процент от всех РНК |
|---|---|---|
| тРНК | 83 | 72,8 % |
| рРНК | 27 | 23,7% |