В мини-обзоре рассмотрены некоторые особенности генома и протеома бактерии Sphingomonas melonis, в частности, в сравнении с тремя другими видами этого же рода. Опробована программа для поиска потенциальных шпилек в ДНК или РНК-продукте. Применены два интернет-ресурса для оценки возможной вторичной структуры ДНК и РНК при предсказании шпилек в геноме и для определения антикодона у тРНК.
Sphingomonas melonis — грамотрицательная лишённая жгутиков бактерия, относящаяся к типу Proteobacteria. Известна как фитопатоген, вызывающий появление бурых пятен на плодах дыни (Cucumis melo var. inodorus), из которых и была впервые выделена и описана в 2002 году. Длина генома S. melonis составляет около 3,65 Mbp (миллионов пар нуклеотидов), из которых ~ 65% — Г+Ц пары (мольная доля) [1]. Помимо сельскохозяйственного значения бактерия интересна тем, что способна разлагать никотин, используя его в качестве единственного источника углерода, азота и энергии [4], а также метаболизировать иные потенциально опасные для человека и окружающей среды вещества, например, имидаклоприд и метомил (широко применяемые в сельском хозяйстве инсектициды) [2], [3]. Таким образом, изучение данного вида может оказаться полезным для сельского хозяйства как с точки зрения предотвращения ущерба, наносимого S. melonis урожаю, так и с точки зрения биоремедиации ядовитых веществ, применяемых для обработки агрикультур.
1) Файлы, содержащие геномы, кодирующие последовательности и расшифрованные кодирующие последовательности, а также таблица особенностей генома скачаны с интернет сайта NCBI.
2) Данные по содержанию различных нуклеотидов, длине геномов и GC-составу получены при помощи программы “скрипт1” из сопроводительных материалов, написанной на python в сервисе Google Colab.
3) Данные для построения графиков GC-skew получены при помощи программы “скрипт2” из сопроводительных материалов, написанной на python в сервисе Google Colab, и обработаны в сервисе Google sheets при помощи функции построения диаграмм. В качестве параметров “window” и “step” программы были выбраны числа 100000 и 1000 соответственно.
4) Для поиска последовательностей потенциальных шпилек использована программа “скрипт3” из из сопроводительных материалов, написанная на python в сервисе Google Colab. В качестве параметров “lencompl” и “lengap” программы были выбраны числа 15 и (от 3 до 12) соответственно.
5) Для построения графиков распределения длин белков, позиции шпилек, позиций начала последовательностей тРНК и других графиков, не касающихся вторичной структуры полимеров, использованы встроенные функции сервиса Google sheets.
6) Для поиска оптимальной вторичной структуры шпилек и тРНК использованы интернет сервисы: rna.urmc.rochester.edu и RNAWebSuite соответственно.
7) Поиск антикодона по вторичной структуре мы проводили по следующему алгоритму: Определяли антикодоновую шпильку (противолежит свободным концам НК, в случае наличия двух противолежащих выбирали большую) Находили в петле антикодоновой шпильки три нуклеотида, равноудалённых от конца петли (начала стебля шпильки) Если п1 или п2 не могли быть выполнены, относили структуру к неоднозначно трактуемым
8) Количества кодонов для каждой из аминокислот и количества старт-кодонов получены при помощи программы “скрипт4” из сопроводительных материалов, написанной на python в сервисе Google Colab
В таблице 1 приведены базовые численные характеристики геномов перечисленных выше видов:
| вид, штамм | длина генома, mbp | A | T | G | C | содержание GC-пар, % |
|---|---|---|---|---|---|---|
| S. alpina DSM 22537 | 5197460 | 935219 | 937585 | 1658068 | 1666588 | 0,640 |
| S. sanxanigenens NX02 | 6205896 | 1031698 | 1029848 | 2075824 | 2068526 | 0,668 |
| S. melonis ZJ26 | 3653375 | 596177 | 595355 | 1234328 | 1227515 | 0,674 |
| S. panacis DCY99 | 5003808 | 859289 | 858823 | 1638255 | 1647441 | 0,657 |
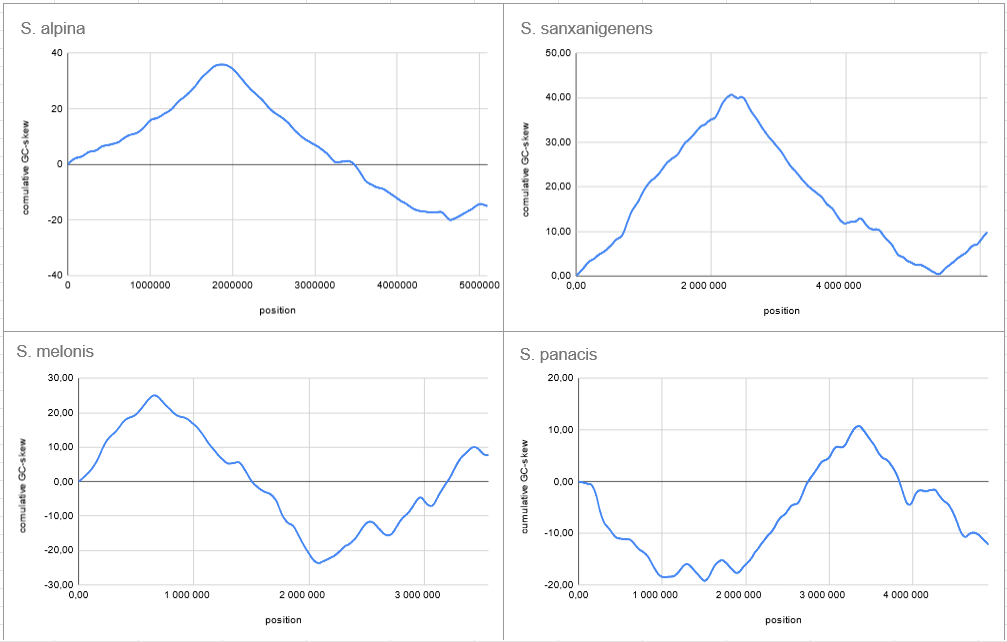
По данным таблицы видно, что наименьший процент GC-пар в геноме характерен для S. alpina, бактерии-психрофила [5]. Для всех видов количество аденинов очень близко (различия колеблются в пределах 1%), как и количество цитозинов и гуанинов, что говорит о практически одинаковом нуклеотидном составе на “+” и “-” -цепях. Длина генома S. melonis оказалась заметно меньше длины геномов других видов (~72,8% от общего среднего).
На рисунке 1 показаны графики GC-skew для четырёх видов. По ним можно приблизительно понять, где находится ориджин и точка терминации репликации (минимумы и максимумы на графиках соответственно). По рисунку видно, что даже в пределах рода форма графика GC-skew может значительно меняться.

Под потенциальной шпилькой мы подразумеваем такой участок цепи нуклеиновой кислоты, который может “склеить” своё начало и конец посредством образования водородных связей между комплементарными парами нуклеотидов.
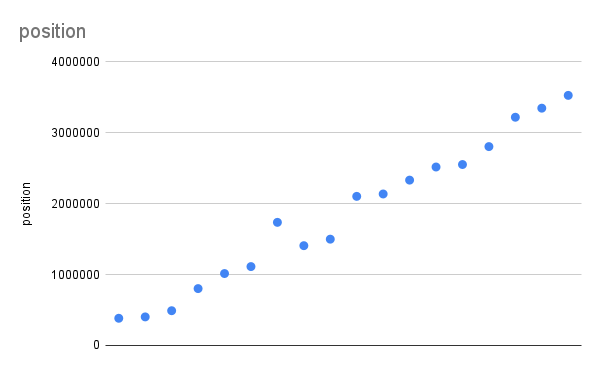
В геноме S. melonis найдено 18 различных потенциальных шпилек по заданным параметрам. Судя по рисунку 2, они распределены по геному более-менее равномерно:
Из них мы проанализировали произвольные 3. Результаты отражены на рисунке 3:
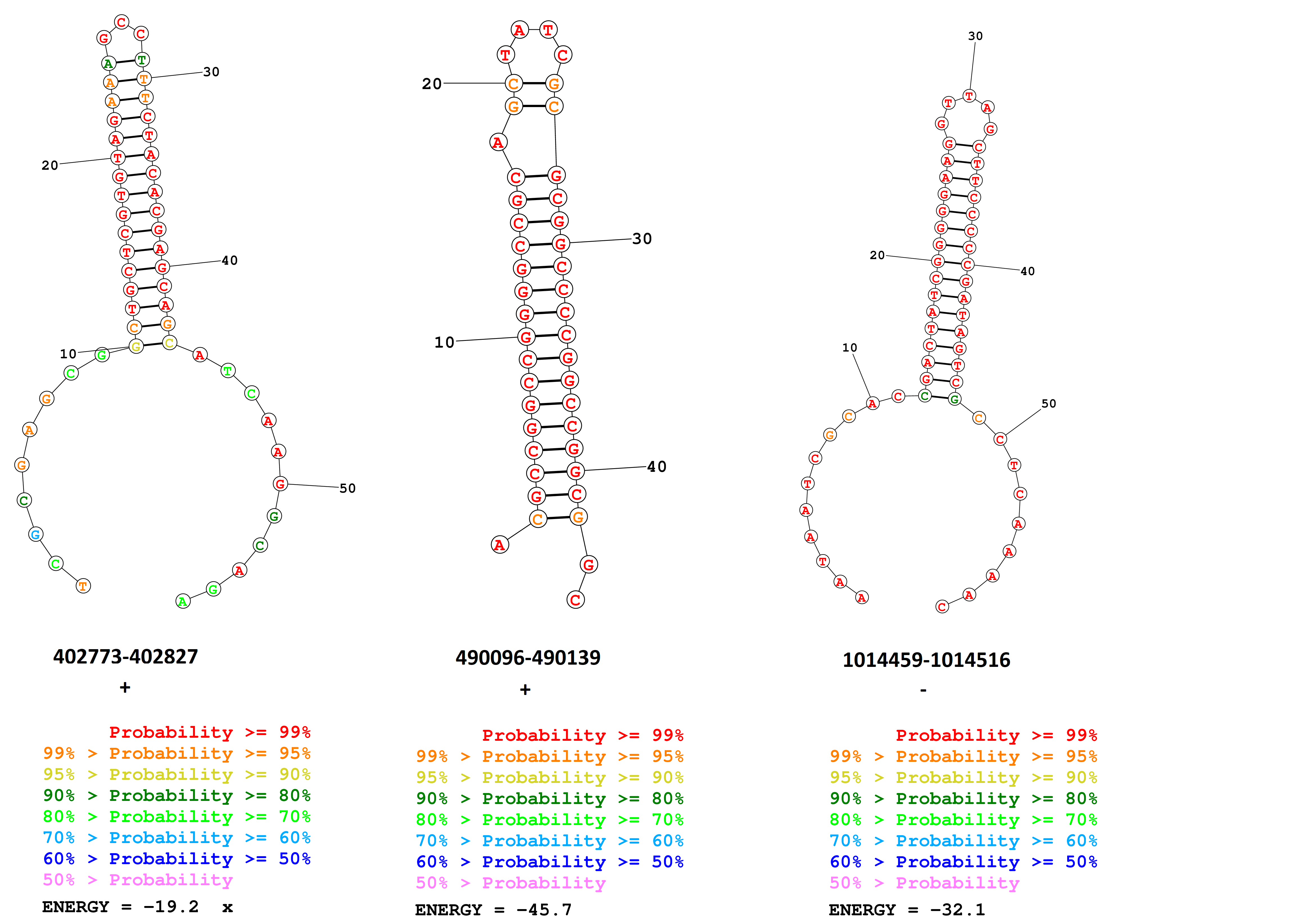
Все три последовательности не входят целиком ни в одну из кодирующих последовательностей генома, а лежат между ними, частично заходя в начала и концы.
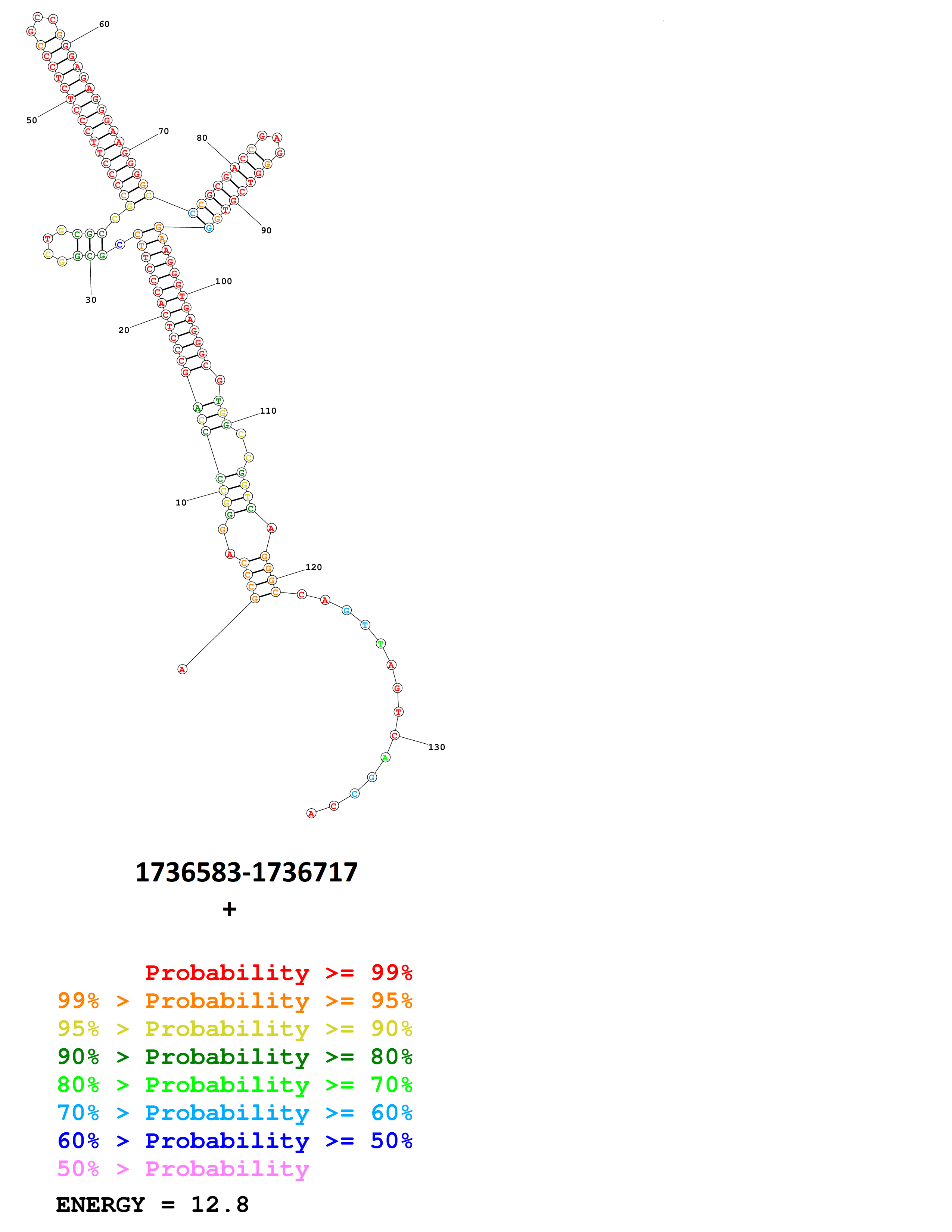
Для ещё одной последовательности мы также построили структуру, однако по ходу построения выяснилось, что она захватывает больший участок генома и включает в себя найденную нами по с помощью кода:
В геноме S. melonis найдено 50 тРНК. Из них по 25 расположены на “+” и “-” цепях. Рисунок 5 показывает их распределение по геному (мы брали координаты начала вне зависимости от цепи, поскольку длина последовательности тРНК не превышает 100 нуклеотидов, что пренебрежимо мало в сравнении с длиной генома):
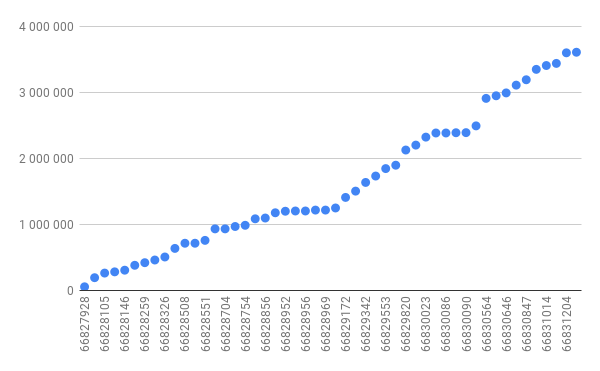
Для первых 10 (первых) из 50 последовательностей мы построили гипотетическую вторичную структуру, определили антикодон и соответствующую ему аминокислоту и сравнили её с реальной (из описания последовательностей в файле с РНК). Все 10 структур лежат в папке “tRNA” из сопроводительных материалов.
Пояснительный рисунок:
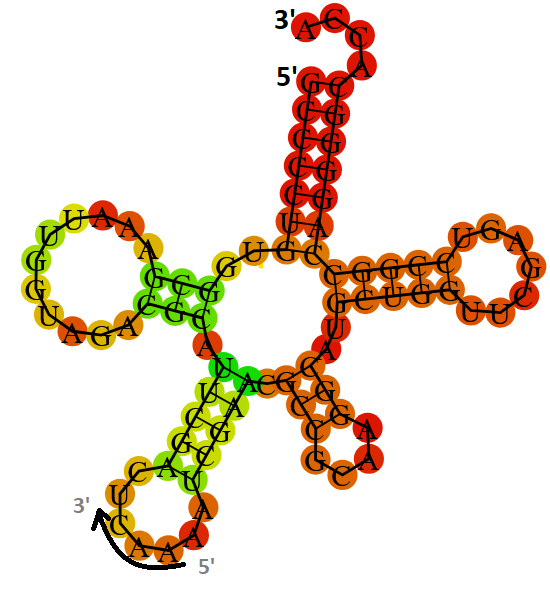
Для всех 10 тРНК на полученных структурах можно выделить CCA свободный “хвост” на 3’-конце. Для 7 из десяти получилась характерная 4 или 5 - шпильковая структура с явно выделяемой антикодоновой шпилькой (для всех из них найденный антикодон соответствует аминокислоте, данной в описании тРНК) Ещё три дали неоднозначно трактуемые структуры.
Количество тРНК в геноме меньше, чем 61 (число кодирующих аминокислоты триплетов). Поэтому мы посмотрели для каких аминокислот тРНК меньше, чем “должно быть”. Результаты представлены в таблице 2:
| аминокислота | тРНК | кодоны |
|---|---|---|
| Ala | 4 | 4 |
| Arg | 5 | 6 |
| Asn | 1 | 2 |
| Asp | 2 | 2 |
| Cys | 1 | 2 |
| Gln | 2 | 2 |
| Glu | 2 | 2 |
| Gly | 3 | 4 |
| His | 1 | 2 |
| Ile | 3 | 3 |
| Leu | 5 | 6 |
| Lys | 2 | 2 |
| Met | 3 | 1 |
| Phe | 1 | 2 |
| Pro | 3 | 4 |
| Ser | 4 | 6 |
| Thr | 3 | 4 |
| Trp | 1 | 1 |
| Tyr | 1 | 2 |
| Val | 3 | 4 |
Затем мы отобрали те аминокислоты, для которых в таблице2 стоят 1 и 2 во втором и третьем столбцах соответственно и по описанному выше алгоритму смоделировали для соответствующих тРНК структуру и нашли, какой именно кодон им соответствует. Результаты отражены на рисунке 8:
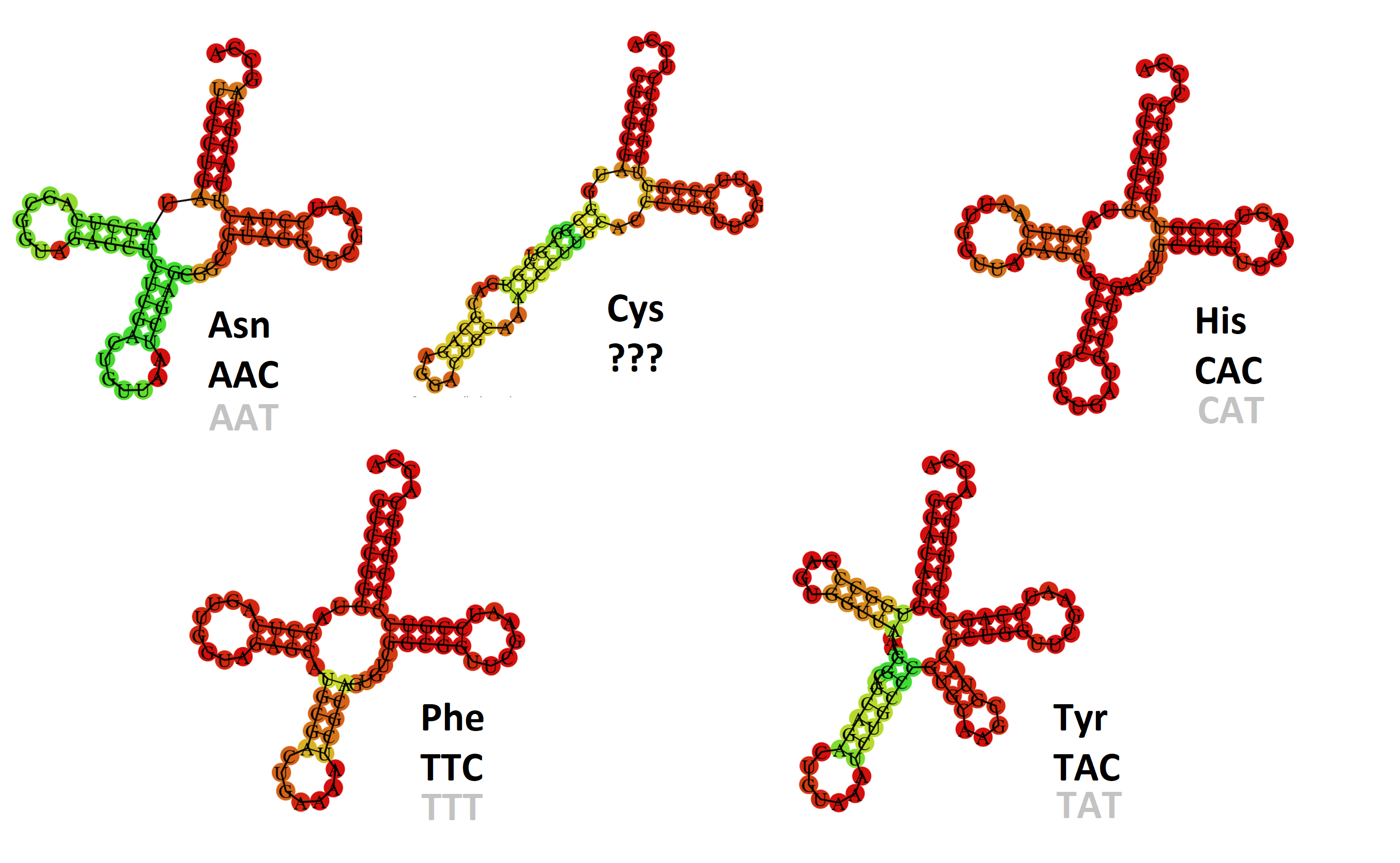
А в таблице 3 приведены частоты встречи каждого из двух кодонов для этих пяти аминокислот:
| аминокислота | кодон | встречаемость | % для всех кодонов к-ты |
|---|---|---|---|
| Asn | AAT | 7365 | 28,2 |
| AAC | 18772 | 71,8 | |
| Cys | TGT | 1067 | 13,7 |
| TGC | 6729 | 86,3 | |
| His | CAT | 9776 | 45,7 |
| CAC | 11610 | 54,3 | |
| Phe | TTT | 3846 | 10,6 |
| TTC | 32514 | 89,4 | |
| Tyr | TAT | 14285 | 59,0 |
| TAC | 9922 | 41,0 |
1) Сравнительно небольшой размер (относительно других видов) генома S. melonis можно объяснить а) нерепрезентативностью выборки видов; б) упрощением, вызванным переходом к фитопаразитизму или симбиозу с растениями (как штамм ZJ26 [6], который и изучали в этом обзоре)
2) Наименьший GC-процент в геноме оказался у психрофила, что логично: температура плавления ДНК повышается с ростом GC-содержания.
3) Код, применённый для нахождения шпилек, дал результаты, впоследствии подтверждённые построением вторичной структуры (рис.3 и рис.4), однако для доказательства его эффективности требуется проанализировать уж точно более 4 произвольно отобранных шпилек.
4) Построение вторичной структуры тРНК позволяет с высокой точностью назвать антикодон и соответствующую аминокислоту (в случаях, когда полученная структура однозначно трактуемая)
5) Неоднозначная трактовка вторичной структуры тРНК может быть результатом: того, что в наших последовательностях мы не учитывали возможность посттранскрипционной модификации некоторых нуклеотидов
того, что тРНК может существовать в нескольких формах, а интернет-ресурс не учитывает особенностей внутренней среды клеток исследуемого организма при фолдинге6) Найденные нами шпильки могут выполнять регуляторную функцию, так так находятся рядом с началами и концами открытых рамок считывания
7) Неполный набор тРНК может быть важным регуляторным механизмом трансляции: те кодоны, у которых нет соответствующей тРНК, должны связываться с частично комплементарными тРНК, при этом скорость и вообще возможность трансляции, а также состав белкового продукта может меняться.
Все сопроводительный матеориалы, упомянутые в тексте, можно найти в папке по ссылке: Сопроводительные материалы
Мы благодарим преподавателей кафедры информатики ФББ МГУ, проводивших занятия курса, в рамках которого выполнена эта работа.
1) Buonaurio R. et al. Sphingomonas melonis sp. nov., a novel pathogen that causes brown spots on yellow Spanish melon fruits //International journal of systematic and evolutionary microbiology. – 2002. – Т. 52. – №. 6. – С. 2081-2087.
2) Erguven G. O., Demirci U. Statistical evaluation of the bioremediation performance of Ochrobactrum thiophenivorans and Sphingomonas melonis bacteria on Imidacloprid insecticide in artificial agricultural field //Journal of Environmental Health Science and Engineering. – 2020. – Т. 18. – №. 2. – С. 395.
3) Tatar S. et al. Can toxicities induced by insecticide methomyl be remediated via soil bacteria Ochrobactrum thiophenivorans and Sphingomonas melonis? //Current Microbiology. – 2020. – Т. 77. – С. 1301-1307.
4) Wang H. et al. Characterization of a novel nicotine degradation gene cluster ndp in Sphingomonas melonis TY and its evolutionary analysis //Frontiers in microbiology. – 2017. – Т. 8. – С. 337.
5) Margesin R, Zhang DC, Busse HJ. Sphingomonas alpina sp. nov., a psychrophilic bacterium isolated from alpine soil. Int J Syst Evol Microbiol. 2012;62(Pt 7):1558-1563.
6)Matsumoto, H., Fan, X., Wang, Y. et al. Bacterial seed endophyte shapes disease resistance in rice. Nat. Plants 7, 60–72 (2021).