В качестве рассматриваемого семейства я выбрал семиспиральный домен, характерный для GPCR (рецепторы, сопряжённые с G-белками). Эта группа рецепторов встречается только у эукариот (выполняет множество функций: фоторецепторная, хеморецепторная и т.д.), хотя семиспиральные белки присутствуют и у прокариот, выполняя несигнальные функции (например, бактериородопсин) (Strotmann et al., 2011). В настоящее время GPCR - цель около 34% лекарственных препаратов (Hauser et al., 2017). Ниже представлена основная информация о домене из pfam:
1) ID: 7tm_1
2) ACC: PF00001
3) seed: 63 последовательности
4) full: 127566 последовательности (в 638 раз больше рекомендуемого...)
5) архитектур: 1078 (из них я выбрал 2 наиболее представленные: '7tm_1' (115509 sequences) и '7tm_1 x 2' (2444 sequences))
6) для 101 белка в PDB построены 650 структур
7) Домен найден у 628 видов. Из них к Metazoa относятся 452, к Fungi - 44, к некатегоризованным эукариотам - 78 (+3 вида эукариот с маркером No kingdom), к различным вирусам - 36, к Viridiplantae - 1, к Bacteria - 1, акже есть некоторые не имеющие никаких систематических категорий (вероятно, среди них и затерялись оставшиеся 13 видов, но разглядеть их невозможно, а tree не прогружается).
8) HMM-профиль был создан 19 октября 2021 года, в профиле 264 позиции
Для поиска локальных сходств двух белков я выбрал по первому белку из вышеназванных архитектур: X2JIG6_DROME (рецептор Drosophila melanogaster) и M7BHM6_CHEMY (хеморецептор черепахи Chelonia mydas). (указаны ID по Uniprot). При просмотре данных записи Uniprot в глаза бросилось наличие лишь 12 альфа-спиральных участков во втором белке, хотя, учитывая дупликацию семиспирального домена, я, умножив 2 на 7, ожидал увидеть 14 альфа-спиралей. Как выяснилось, недостача спиралей - в первом домене двухдоменного белка.
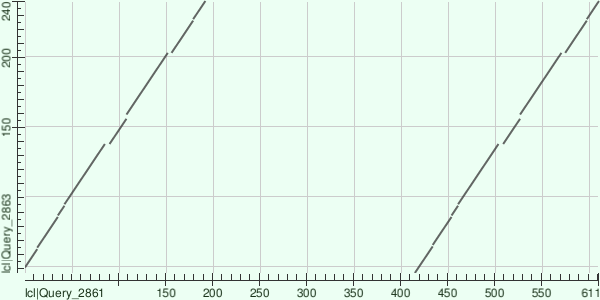
Ниже модно увидеть Dot plot, показывающий локальные сходства указанных последовательностей при пороге на E-value 0,05:

Затем я решил увеличить порог E-value до 15 и получил несколько дополнительных неожиданных прямых. Чтобы разобраться с этими неожиданностями и потерянными альфа-спиралями, я построил следующее изображение:
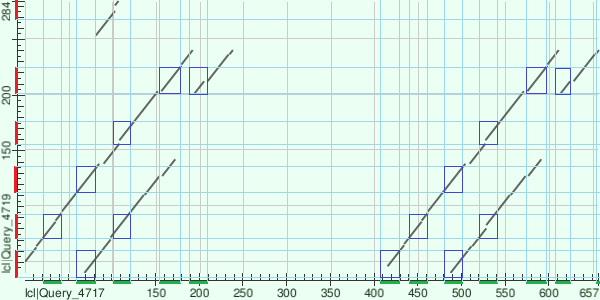
Из рисунка2 можно понять следующее: во-первых, в первом домене черепашьего белка отсутствует первая альфа спираль, и он начинается со второй; во-вторых помимо сходств соответствующих (по номеру) альфа-спиралей обнаружены сходства между 1 мушиной и 3 черепашьей (в первом домене из-за делеции первой альфа-спирали третья кажется второй) и между 2 мушиной и 4 черепашьей (такой же сдвиг в первом домене). А также слабое и, на мой взгляд, случайное, сходство 5 мушиной и 6 черепашьей. При этом седьмые альфа-спирали почти не попали на график ни у одного из белков. Возможно сходство 1 - 3 и 2 - 4 можно теоретически объяснить наличием двухспирального предка, претерпевшего дупликацию (⓵⓶ → ⓵⓶⓵⓶ → ⓵⓶⓷⓸). Интересно, что при попытке понять родство прокариотических и эукариотических семиспиральных белков (которое до сих пор не доказано) была выдвинута гипотеза дупликации трёхспирального предка с предварительно появившейся 4-ой спиралью (⓵⓶⓷ → ⓵⓶⓷⓸ → ⓵⓶⓷⓸⓵⓶⓷ → ⓵⓶⓷⓸⓹⓺⓻) (Agarwal et al., 1993). Что касается 6-ой и 7-ой спиралей, то, видимо, они подвержены наибольшим изменениям, поскольку не дали своих линий сходства.
Для выравнивания в Jalview я выбрал seed, поскольку 127566 (full) последовательностей - не то, с чем хочется работать...
Выделенные мной группы покрашены в зелёный и фиолетовый цвета (первые две группы в проекте Jalview). Созданы с помощью построения Tree.
Для первой группы характерны следующие участки: на 87 позиции выравнивания - всегда ароматическая аминокислота (во второй - никогда). Мотив D------SI (91 - 99, при этом на позициях 94 - 95 довольно консервативный участок CC, характерный почти только для этой группы), мотив V-P (50 - 52). По позиции 32 для первый группы обычен серин, для второй - скорее аспарагин (нечёткое различие). Для второй группы также характерен валин на 114 позиции, для первой - треонин. Интересно, что родство рецепторов выстроилоось скорее по мнемонике функции, чем по организмам (что объяснимо, т.к., вероятно, расхождение семиспиральных белков по функциям произошло значительно ранее, чем расхождение отдельных групп Bilateria, которыми в основном представлен seed).
Таблица с белками домена доступна по ссылке. Осторожно! 291719 белков! Из них содержат только исследуемый домен 274584 (~94%), а аннотированных - 1839 (жалкие 0,63%).
1) Strotmann R. et al. Evolution of GPCR: change and continuity //Molecular and cellular endocrinology. – 2011. – Т. 331. – №. 2. – С. 170-178.
2) Hauser, A. S., Attwood, M. M., Rask-Andersen, M., Schiöth, H. B., & Gloriam, D. E. (2017). Trends in GPCR drug discovery: new agents, targets and indications. Nature reviews Drug discovery, 16(12), 829-842.
3) Agarwal, A., Pearson, P. P., Taylor, E. W., Li, H. B., Dahlgren, T., Herslof, M., ... & Nelson, D. L. (1993). Three-dimensional quantitative structure-activity relationships of 5-HT receptor binding data for tetrahydropyridinylindole derivatives: a comparison of the Hansch and CoMFA methods. Journal of medicinal chemistry, 36(25), 4006-4014.