История началась с подбора организма. Я остановился на риккетсиях Rickettsia sp. и теперь могу постфактум обосновать свой выбор: риккетсии - облигатные внутриклеточные паразиты, поэтому гомологическая рекомбинация (при горизонтальном переносе) в них между видами рода может произойти только при козаражении этими видами одной клетки. При беглом гуглении кориккетсиозов я нашёл одну статью, в которой упомянуто о кориккетсиозах у клещей рода Amblyomma, однако я по-прежнему считаю что такое сильное ограничение на обмен участками геномной ДНК между видами облегчает восстановление последовательности инверсионных событий на дереве риккетсий.
Далее я поискал, какие полные геномы риккетсий доступны в базе NCBI Genomes и выбрал произвольные 12 полных геномов (где были - взял референсные). В таблице ниже указаны видовые эпитеты и соответствующие им идентификаторы последовательности хромосомы (плазмиды тут и далее вообще не учитывались):
| Эпитет | Идентификатор |
|---|---|
| akari | NC_009881.1 |
| asiatica | NZ_AP019563.1 |
| australis | NC_017058.1 |
| canadensis | NC_016929.1 |
| conorii | NC_003103.1 |
| rhipicephali | NC_017042.1 |
| slovaca | NC_016639.1 |
| rickettsii | NC_010263.3 |
| japonica | NZ_AP017600.1 |
| typhi | NC_017066.1 |
| prowazekii | NC_017049.1 |
| helvetica | NZ_CM001467.1 |
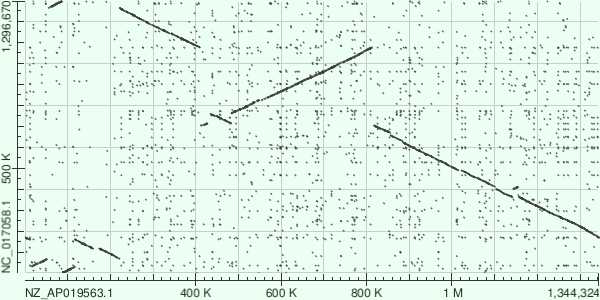
Затем я применил megablast и blastn к последовательностям R. asiatica (по оси x) и R. australis (по оси y):

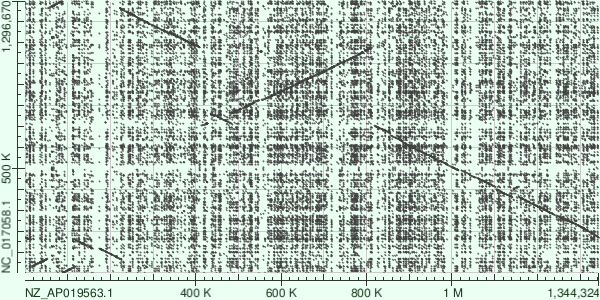
При сохранении общей картины выравнивания (например, крупной инверсии в положении 400к - 800к) в выдаче blastn оказалось огромное количество дополнительных точек, соответствующих многочисленным повторам небольших участков (очевидно, меньших, чем длина слова megablast (28)).
Но меня больше заинтересовала история с инверсиями, поэтому мне захотелось выровнять геномы многих риккетсий на геном какой-нибудь одной, чтобы можно было рассматривать покоординатное соответствие участков генома. Риккетсии разделяются на две группы: группы тифов и группы пятнистых лихорадок. Во второй группе значительно больше видов, поэтому я взял из группы тифов R. prowazekii, а всех остальных выравнивал на неё. Результатом этой деятельности стал рисунок 3:
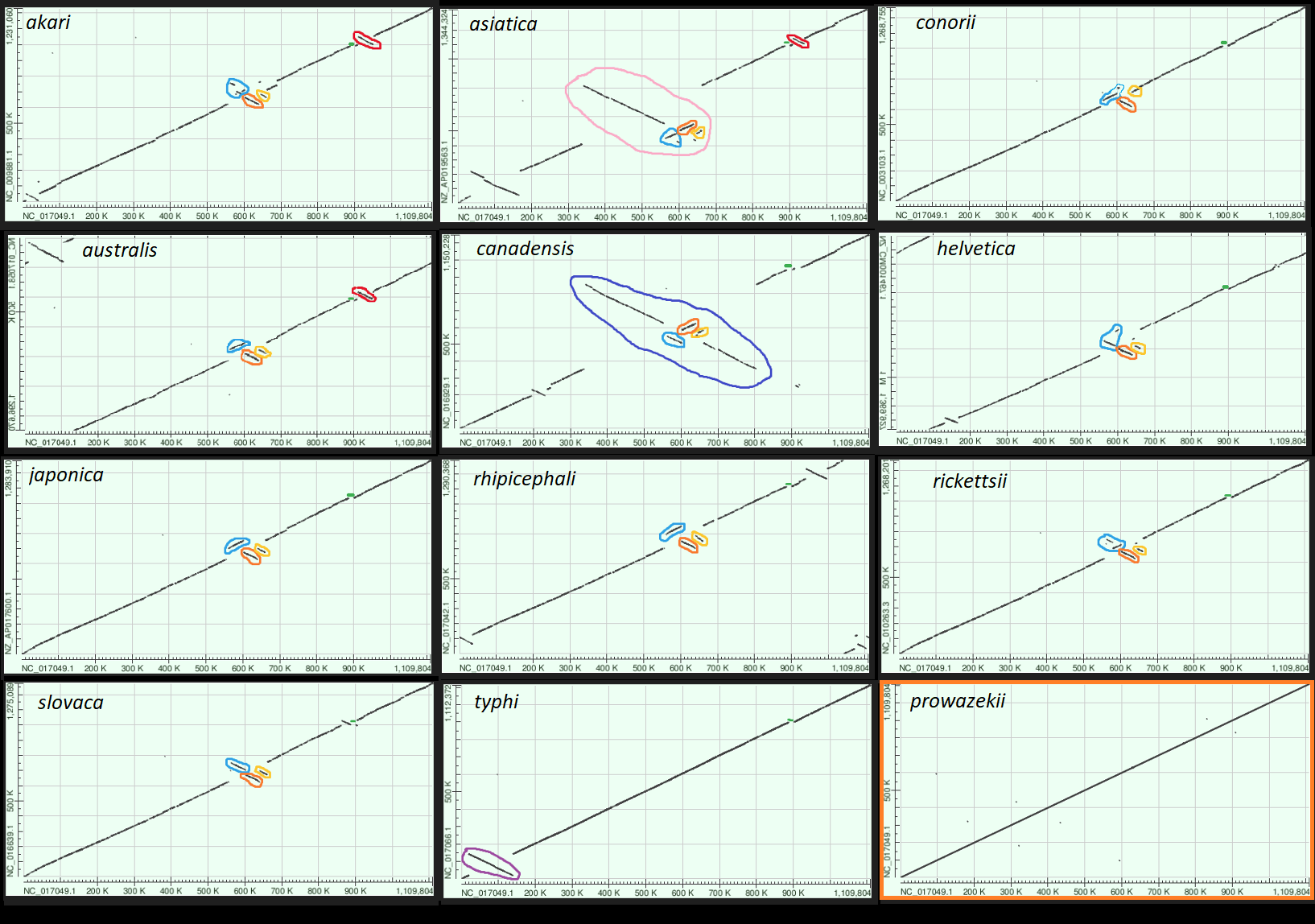
Из этого рисунка можно попробовать сделать несколько выводов:
1) Во-первых, комплекс инверсий (тёмно- и светло-оранжевая и голубая) по координатам ~550к - 670к в том или ином виде присутствует у всех, кроме R. prowazekii и R. typhi, что даёт основание предполагать, что последнии образуют сестринскую группу по отношению к остальным видам.
2) Во-вторых, маленькая зелёная "делеция" в позиции чуть менее 900 есть у всех кроме R. prowazekii, и я думаю, что, вероятнее всего, это не делеция, а вставка у R. prowazekii (иначе бы пришлось предполагать её независимое возникновение у R. typhi и всех остальных).
3) В третьих, красная инверсия после позиции 900 хорошо объединяет три вида в общую группу: R. akari, R. asiatica и R. australis.
4) Голубой участок много раз претерпевал инверсии и внутренние транслокации в разных ветвях рода.
5) Розовая и тёмно-синяя инверсии произошли независимо, поскольку имеют разные "правые" координаты, однако их "левые" координаты совпадают, возможно, указывая на положение горячей точки.
6) Ну и конечно же у R. australis неправильно выбрана точка начала генома, из-за чего кажется, будто там произошла транслокация (помимо инверсии).
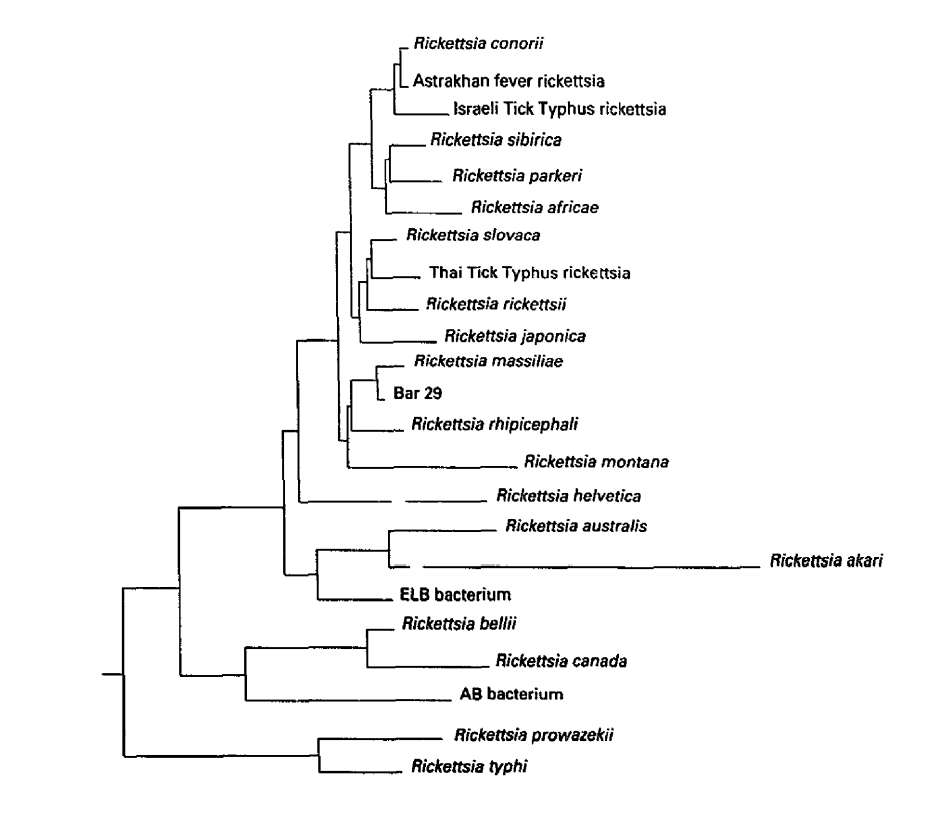
Ниже на картинке приведена филогения рода, полученная марсельскими учёными в 1995 году:
de Abreu D. P. B. et al. Two for the price of one: Co-infection with Rickettsia bellii and spotted fever group Rickettsia in Amblyomma (Acari: Ixodidae) ticks recovered from wild birds in Brazil //Ticks and tick-borne diseases. – 2019. – Т. 10. – №. 6. – С. 101266.
Roux V., Raoult D. Phylogenetic analysis of the genus Rickettsia by 16S rDNA sequencing //Research in Microbiology. – 1995. – Т. 146. – №. 5. – С. 385-396.