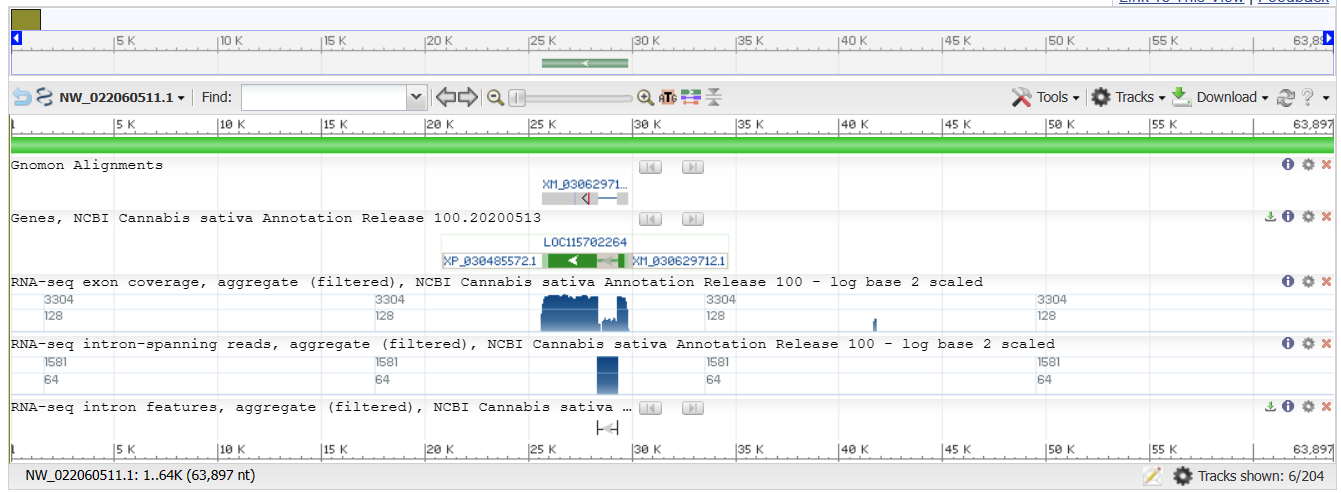
Для работы с контигом я выбрал небольшой участок, охарактеризованный как unplaced genomic scaffold (не отнесён ни к одной хромосоме), с идентификатором NW_022060511.1. На рисунке ниже можно увидеть схему расположения выделенных в нём участков:

Последовательность контига доступна для скачивания. Далее я работал с её участком по координатам (25000-30000), так как изначальная длина слишком большая (63897 bp), а что-то интересное есть только на выбранном участке.
По результатам различных видов бластования я составил следующую таблицу, содержащую основную информацию о входных параметрах и результатах:
| Вид BLAST | Зачем он нужен | Какие таксоны исключены | длина слова | сколько нашлось | что нашлось | Примечания |
|---|---|---|---|---|---|---|
| megablast | Поиск очень похожих нуклеотидных последовательностей на заданную по нуклеотидной БД (например, быстро сориетироваться, есть ли что-то очень похожее на вашу последовательность) |
Сначала пытался исключить все цветковые, но тогда не было ни одной находки. Поэтому я исключил только род Cannabis |
28 | 68 | ДНК-связывающие домены белков различных высших растений. На картинке справа видно, что почти все находки упали на доинтронную часть гена, при этом самая правая часть гена до интрона наиболее часто выравнивалась с чем-то из БД. |
|
| blastn | Более чувствителен, чем megablast, но работает дольше за счёт поиска менее похожих последовательностей (например, поиск гомологов в не самых близких родственниках) |
Исключил таксон angiosperms | 11 | 28 | С хорошей достоверностью нашлась последовательность в Physcomitrella patens, кодирующая тоже ДНК-связывающий домен, а также несколько находок у селагинелл. Хочется отметить, что только первые 11 из 28 находок заслуживают доверия (дальше очень сильно повышается e-value и падает процент покрытия) |
|
| blastx | Сравнивает транслированный запрос с белковой базой (например, поиск вероятных белковых продуктов в неаннотированной последовательности) |
искал только по Rhodophyta | 6 | 14 | В красных водорослях все находки оказались либо кодирующими белки репликации (связывают ДНК!) либо отнесены к hypothetical protein. Любопытно, что у красных водорослей часто есть маленький постинтронный участок, где последовательности тоже выровнялись с какой-то достоверностью |
|
| tblastx | Сравнивает транслированный запрос с транслированной нуклеотидной базой (например, поиск далёких гомологов последовательности) |
3 | blast ругался | Бласт на меня наругался, и я не захотел получить бан от NCBI (некоторые получили) |
Он просил меня поменять что-то в моём поведении... |
Выданные последовательности - 16S и 23S рРНК (в малой и большой субъединице соответственно) E. coli. Поскольку подготовленная мной база данных нуклеотидная, как и запрос, при этом транслировать рРНК можно, но непонятно зачем, я воспользовался алгоритмом blastn.
Подготовка нуклеотидной базы:
makeblastdb -in GCF_900626175.2_cs10_genomic.fna -dbtype nucl
Бластование (показано для 16S, аналогично для 23S):
blastn -task blastn -evalue 0.05 -query 16s.fasta -db GCF_900626175.2_cs10_genomic.fna -out blastn_16s.fasta -outfmt 7
Параметр e-value аналогичен такому же в веб-интерфейсе, параметр outfmt определяет формат выдачи.
При бластовании на оба вида РНК нашлось по 14 находок, причём на хромосоме 1,2 и 3, но по-разному разбитые между первой и второй хромосомой: 6/8 и 7/5/2 соответственно