
Для практикума мне был выдан идентификатор Y644_TROW8 - трансмембранный белок из бактерии Tropheryma whipplei , вызывающей странную болезнь ЖКТ Уиппла. В качестве бета-белка я нашёл в базе данных OPM белок перфорин мыши (PDB: 8a1d, UniProt: MPEG1_MOUSE). Этот белок вырабатывается иммунной системой для неспецифичной атаки на патогены у млекопитающих. Белок состоит из 16 одинаковых (почти) субъединиц, образующих кольцо для атаки на мембрану патогена.
В таблице ниже приведены основные характеристики, взятые из БД (трансмембранные участки и среднее кол-во остатков указаны для "средней" субъединицы, но они почти одинаковы):
| Толщина мембранной части | 29.4 Å |
|---|---|
| координаты трансмембранных участков | 113-120; 127-132; 235-244; 248-256 |
| среднее кол-во остатков на бета-тяж | 8.25 |
| локализация | внешняя мембрана патогена |
А на рисунке ниже изображён весь гексадекамер (простите). Изображение получено в PyMol.

Для прогнозирования трансмембранных участков я воспользовался сервисом DeepTMHMM и получил результаты в виде текстовой выдачи в формате .md:
А ниже изображена гарфическая выдача программы:
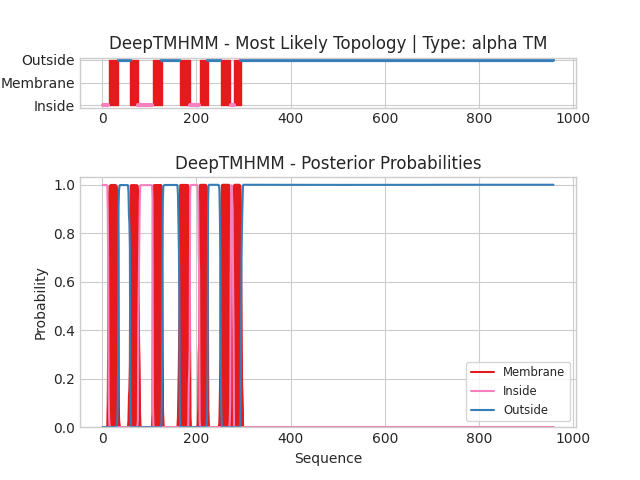
Далее для Предсказания положения выданного белка в мембране я воспользовался сервисом PPM. При запуске использовал следующие параметры: Number of Membranes: 1; Allow curvature: no; Topology (N-ter): in, потому что моя бактерия грамположительная, а расположение N-конца взял из предсказания TMHMM. В таблице ниже приведены основные характеристики, полученные с помощью модели:
| Толщина мембранной части | 32.3 ± 0.9 Å |
|---|---|
| координаты трансмембранных участков | 9-34; 56-81; 107-129; 161-185; 202-226; 251-273; 276-297 |
| среднее кол-во остатков на бета-тяж | 24.3 |
| локализация | мембрана грамположительной бактерии |
А на рисунке ниже изображена структура, визуализированная в PyMol:
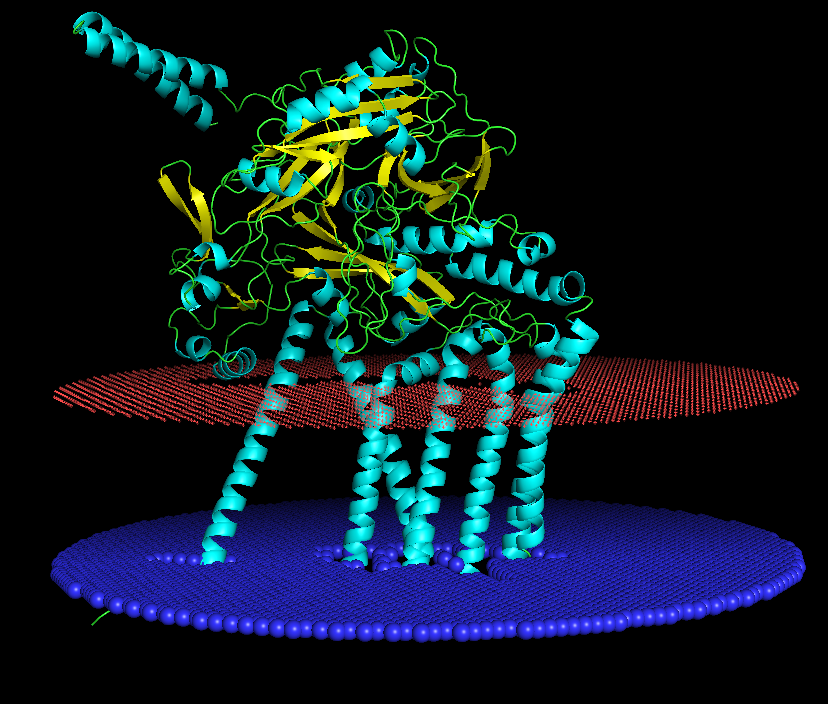
Все семь спиралей были найдены TMHMM, и определены как трансмембранные PPM. Координаты определённых трансмембранных участков отличаются незначительно, при этом участок, указанный PPM всегда не меньше и почти всегда содержит внутри себя участок (+2 - +3 а.к. с обоих концов), определённый TMHMM. Что касается структуры, предсказанной AlphaFold из Uniprot, то там всё неудивительно: альфа спирали определены с хорошей достоверности, а вот соединяющие их олигопептидные свободные нити - с плохой. Они и по жизни могут болтаться и не иметь строгого положения.