Для начала я собрал содержимое всех файлов с протеомами в один и назвал его db.fasta. Затем я проиндексировал базу с протеомами при помощи команды:
makeblastdb -in db.fasta -dbtype protПосле этого запустил локальный бласт файла с именем query.fasta, содержащего последовательность белка CLPX_ECOLI, против созданной ранее базы протеомов с порогом на e-value = 0.001:
blastp -query query.fasta -db db.fasta -evalue 0.001 > res.fasta
В таблице ниже приведены находки в "моих" бактериях:
| название (мнемоника) | e-value |
|---|---|
| CLPX_STRAW | 0.0 |
| Q1AVT0_RUBXD | 0.0 |
| CLPX_MYCTU | 0.0 |
| CLPX_COREF | 0.0 |
| CLPX_CORDI | 0.0 |
| CLPX_ACIC1 | 0.0 |
| CLPX_ARTS2 | 0.0 |
| Q47MU4_THEFY | 0.0 |
| A0K1M3_ARTS2 | 1e-07 |
| Q1AU05_RUBXD | 6e-07 |
| Q8FMH5_COREF | 2e-05 |
| Q6NFB1_CORDI | 7e-05 |
| A0LW31_ACIC1 | 8e-05 |
| Q47MZ2_THEFY | 2e-04 |
| A0LRB8_ACIC1 | 3e-04 |
| Q1AY82_RUBXD | 3e-04 |
| FTSH_ACIC1 | 4e-04 |
| RUVB_ARTS2 | 6e-04 |
| Q82QV8_STRAW | 7e-04 |
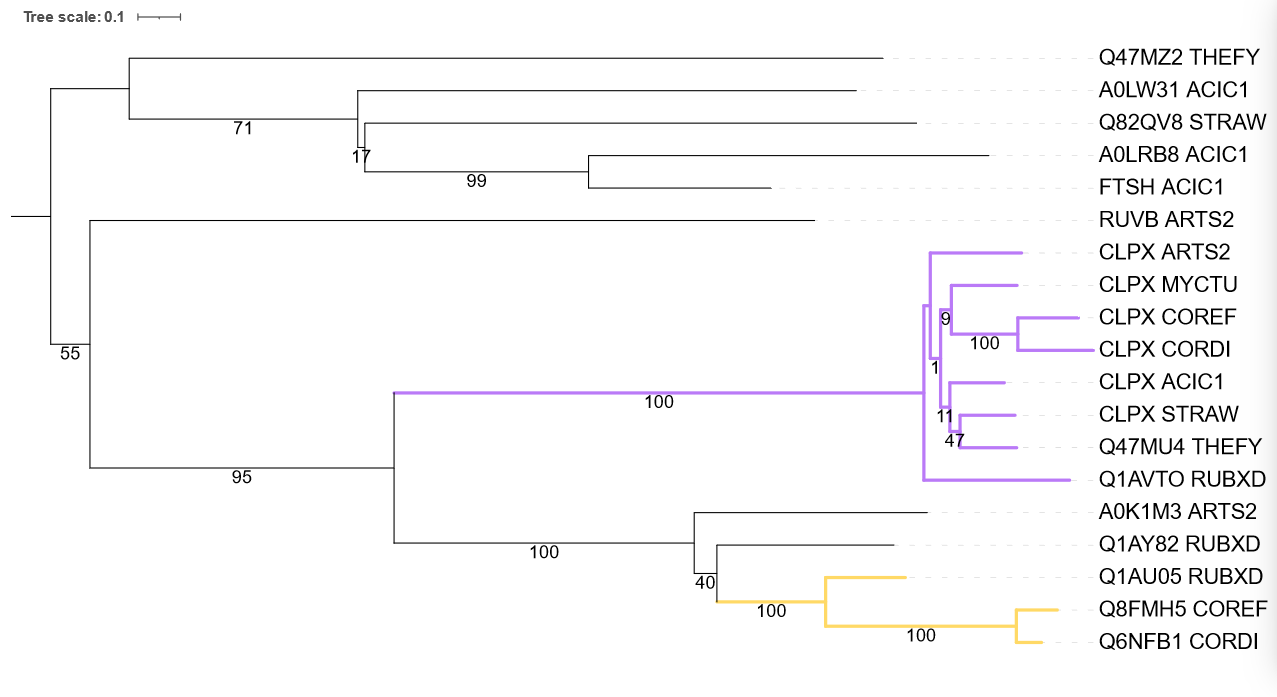
Все находки из таблицы выше я объединил в один файл, оставив только мнемонические названия. Затем я воспользовался французским ресурсом (VIVE LA FRANCE!!!) NGPhylogeny для построения дерева отобранных последовательностей. В качестве программы для множественного выравнивания я использовал MAFFT, алгоритм построения дерева - FASTME с рекомендованными в указаниях параметрами. В результате получился файл с деревом в формате Newick. На рисунке ниже показано получившееся в iTOL дерево:

По дереву можно указать три пары ортологов:
CLPX COREF - CLPX CORDI
Q8FMH5 COREF - Q6NFB1CORDI
Q47MU4 THEFY - CLPX STRAW
и паралогов:
A0LRB8 ACIC1 - FISH ACIC1
Q1AY82 RUBXD - Q1AU05 RUBXD
RUVB ARTS2 - A0K1M3 ARTS2
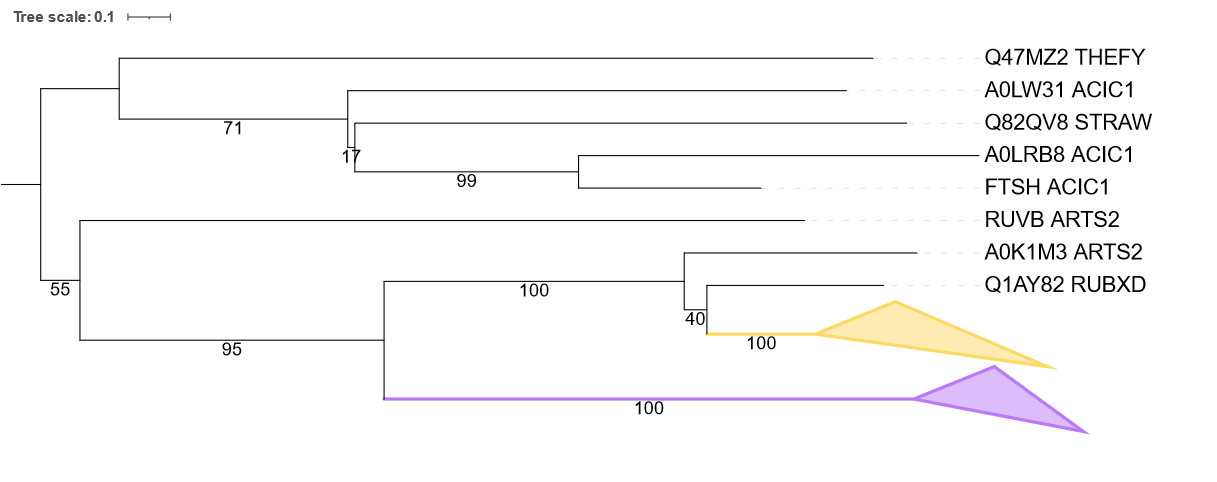
На рисунке 2 изображено дерево со "схлопнутыми" ортологическими группами:
Ниже для наглядности сравнения привожу дерево из первого практикума:
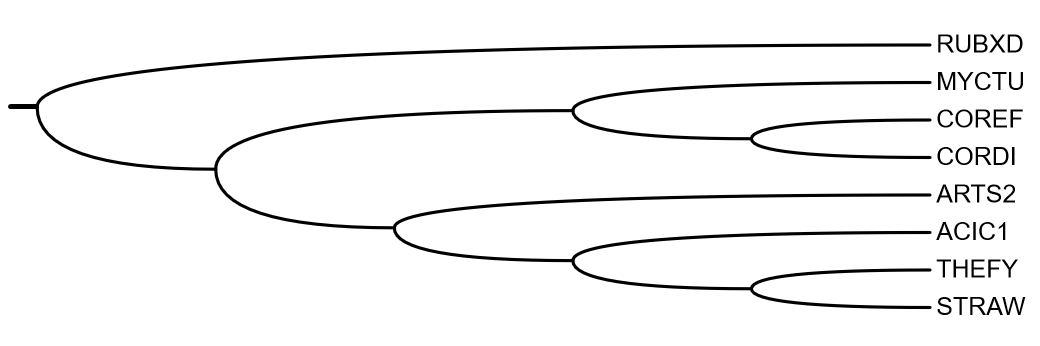
Фиолетовая группа содержит белки из всех 8 бактерий и строго по одному. E-value для них всех - "машииный" ноль. Это всё АТФ-зависимые Clp протеазы. Внутрення иерархия фиолетовой группы почти совпадает с деревом бактерий: всё так же, за исключением положения ARTS2, который теперь отходит вторым после корня (но бутстрэп поддержка этой ветви очень низкая, так что...). Что касается оранжевой группы, то она не противоречит филогении: хотя все эти бактерии не входят в группу {STRAW, THEFY, ACIC1, ARTS2}, среди них почему-то отсутствует ортологичный белок для MYCTU.