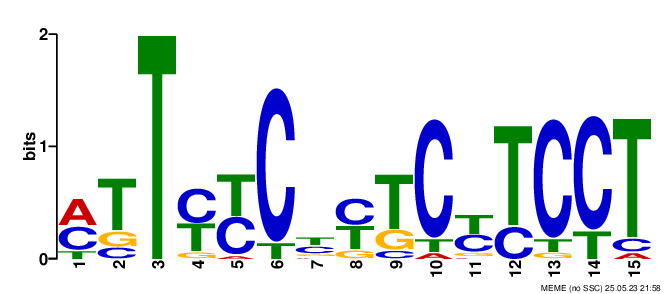
Для подготовки данных я придерживался следующего порядка действий:
1) Скачал геном Treponema pallidum по ссылке.
2) Воспользовался ресурсом Operon-mapper для получения файла с координатами оперонов.
3) Использовал слегка изменённый скрипт Георгия Муравьёва для получения файлов с выборкой обучения (околопромоторные области генов домашнего хозяйства длиной в 100 нуклеотидов), выборкой тестирования (промоторные области любых оперонов такой же длины) и выборкой негативного контроля (участки той же длины, вырезанные из рандомных мест генома).
Затем для поиска мотивов я запустил локальный MEME для обучающей выборки следующей командой (тип последовательности - ДНК, количество искомых мотивов - 3, минимальная длина мотива - 6):
meme housekeeping.fasta -dna -nmotifs 3 -minw 6
В результате нашлось 3 мотива, информация для которых показана на трёх Logo-иллюстрациях ниже:
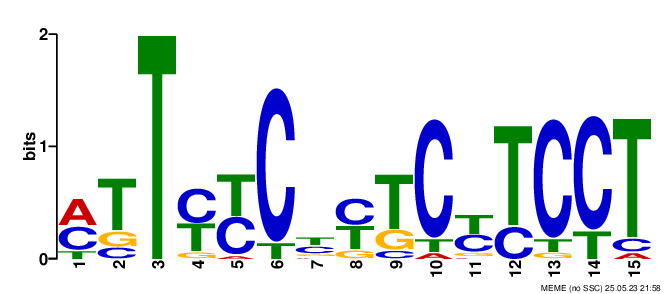

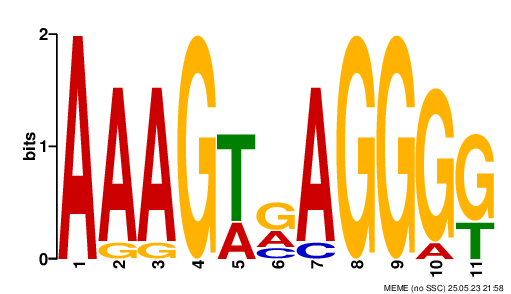
Вся текстовая выдача MEME доступна по ссылке. Для дальнейшей работы я выбрал первый найденный мотив №1, потому что он с наименьшим E-value и потому что мне показалось забавным, что он только из пиримидинов.
Для проверки осмысленности найденного мотива, я запустил локальный FIMO для негативного контроля и тестовой выборки соответственно двумя командами:
fimo --norc -motif MTTYYCYYTCYTCCT -thresh 0.001 meme_out/meme.txt negative.fasta
fimo --norc -motif MTTYYCYYTCYTCCT -thresh 0.001 meme_out/meme.txt promotors.fasta
В результате в негативной выборке оказалось 10 находок, а в тестовой - 422, что, в общем-то, говорит о том, что наш мотив специфичен для промоторной области. Результаты FIMO в формате html доступны для негативного контроля и тестовой выборки.