Для анализа я выбрал домен PF09191, внеклеточная часть рецептора CD4, потому что с ним связывается ВИЧ перед проникновением в клетку, а ВИЧ интересный. Основные характеристики выбранного домена:
AC: PF09191
Seed: 20
Full: 171
Средняя длина: 108.2
Средний процент идентичности: 50
Среднее покрытие: 24.14
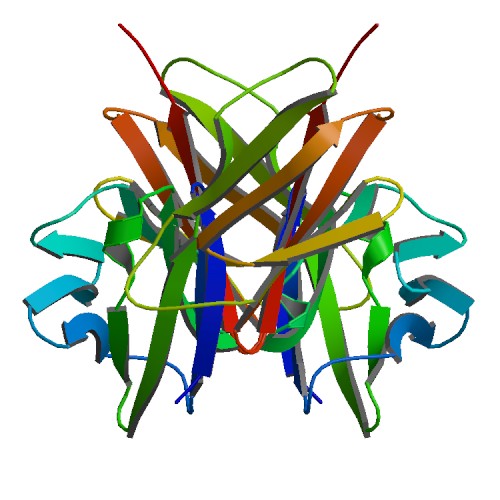
На рисунке ниже изображён человеческий CD4:

Для HMM-профиля я выбрал доменную архитектуру CD4-extracel, Tcell_CD4_C, содержащую 25 последовательностей. По ссылке доступен файл со всеми AC из full. 25 последовательностей с нужной архитектурой выровнял программой mafft с параметрами по умолчанию. В Jalview использовал функцию Remove redundancy с параметром 85%, при этом осталось 14 из 25 последовательностей. Затем отрезал C- и N- gap-богатые области, в результате чего получил файл для построения HMM-профиля. Сам профиль получил и откалибровал следующими командами:
hmm2build profile for_hmm.fasta
hmm2calibrate profile
Поиск по full сделал командой:
hmm2search --cpu=1 profile PF09191_full_length_sequences.fasta &> hmmsearch_log.txt
В результате получил выдачу программы.
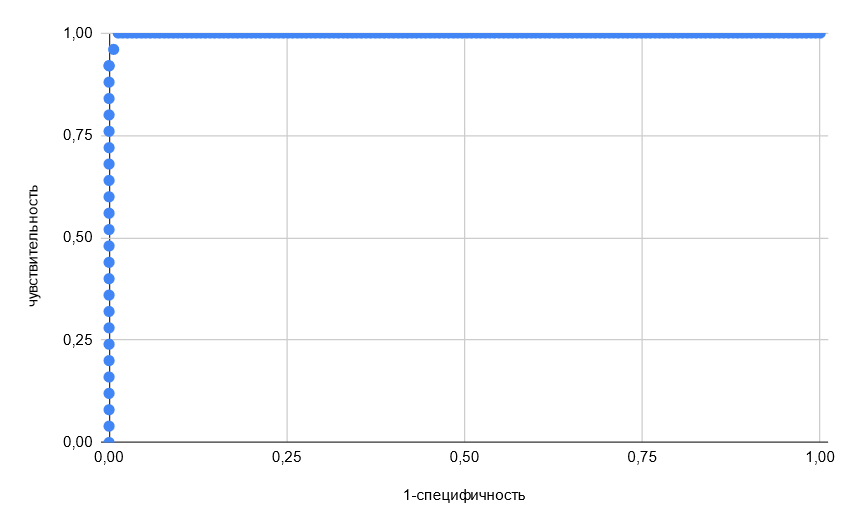
Вообще, для оценки профиля строить ROC-кривую в моём случае оказалось и необязательным: По выдаче было видно, что 25 "правильных" белков содержались в 26 верхних строчках. Всё же приведу картинку кривой, которая это наглядно иллюстрирует:
Мне показалось интересным посмотреть на группу находок с положительными весами, но не из списка "наших" 25 белков. Дело в том, что все, кроме наших 25 и этих 7 "неожиданных" белков имеют отрицательный вес. Для того, чтобы понять, в чём дело, я посмотрел на доменные архитектуры этих находок.
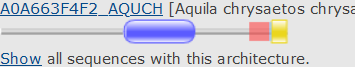
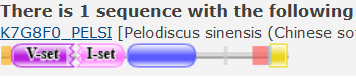
По рисунку 3 видно, что большинство из этих находок имеют архитектуру, "старшую" по отношению к нашей, т.е. содержат её в качестве элемента своей архитектуры. В таком случае, находя их с большим весом и низким E-value по HMM-профилю, мы не совершаем ошибки, а просто находим наш двойной домен в более сложных конструкциях.
Интересный факт: Один мой однокурсник, используя рандомизацию поиска домена, независимо выбрал домен Tcell_CD4_C, следующий за моим в нашей доменной архитектуре. Интересно, какова вероятность такого события в предположении о случайности выбора...