BLAST - инструмент для быстрого поиска последовательностей в банках данных по гомологииv.
Работа с программой Blastp.
Blast
Поиск белка GlpF по фрагменту его аминокислотной последовательности.
Результьтаты поиска по фрагменту длиной 30 аминокислот (MSQTSTLKGQCIAEFLGTGLLIFFGVGCVA):

Результьтаты поиска по фрагменту длиной 10 аминокислот (MSQTSTLKGQ):

Причины отличий.
Различия в e-value могут быть обяснены тем, что с уменьшением длины последовательности, по которой проводился поиск, возрастает
вероятность простого совпадения аминокислот. Т.е. скорее всего произойдет простое совпадение 10 аминокислот, чем 30.
Различия в величине score также объяcняется длиной заданной последовательности. При меньшей длине (10 аминокислот) программа создаст больше гэпов, чем с большей (30 аминокислот).
У найденных белков разные порядковые номера, т.к. чем меньшее количество
аминокислот мы используем для поиска, тем выше вероятность того, что большая
выборка белков будет обладать данной последовательностью.
Причины сходств.
Идентичность 100%, что указывает на адекватность поиска, т.е. найден тот белок,
который и был источником данных выборок аминокислот.
Является ли BLAST инструментом для поиска ортологов.
На примере поиска по последовательности репрессора рибозного оперона RbsR из Bacillus subtilis была проведена проверка: является ли
Blastp инструментом для поиска ортологов.
В данном случае ортологами считались белки из разных организмов, в названии
которых было RbsR.
Выборка получилась следующая (e-value до 10):
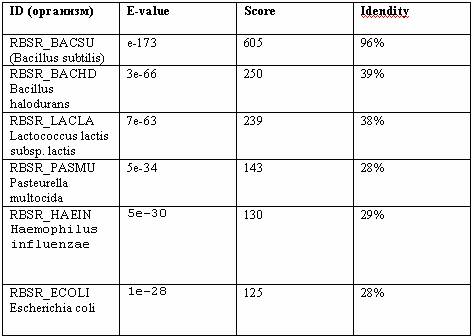
В результате поиска Blast способен находить ортологов, но не является
специализированным инструментом для подобной цели, т.к. помимо необходимых
белков (в названии которых было RbsR), он находит массу других, обладающих
сходством с заданной последовательностью, но не ортологов (с другим описанием
функций.) Доля последних составляет значительную часть. Следовательно, Blast
можно использовать для подобного поиска, но при этом результат необходимо
обрабатывать вручную.
Главная страница



