| Главная | Семестры | Обо мне | Ссылки |
|---|
Описание АТФ-связывающего сайта в структуре белка RadA (идентификатор PDB 3NTU)
Белок RadA является ДНК-зависимой АТФазой. Он образует филамент с одноцепочечной ДНК, после этого гидролизует молекулу АТФ и активизирует обмен участками между гомологичными цепями. Молекула AMP-PNP является аналогом АТФ, в котором атом кислорода, соединяющий бета- и гамма-атомы фосфора, замещен на атом азота. В PDB-файле и Jmol молекула имеет имя ANP. AMP-PNP - мощный конкурентный ингибитор белков, который также позволяет кристаллизовать АТФазы, образуя с ними комплексы вместо АТФ. По расположению AMP-PNP в трехмерной структуре белка на рис. 1 можно сделать вывод, где расположен АТФ-связывающий сайт белка RadA. Радужный градиент был создан командой color group в Jmol. На рис. 2 показана формула AMP-PNP.
В структуре белка RadA также есть атомы натрия и магния. Возможно, они играют роль кофакторов в белке, но есть и вероятность, что их добавили к белку для упрощения его кристаллизации. Здесь мы подробно рассмотрим взаимодействие RadA с AMP-PNP.
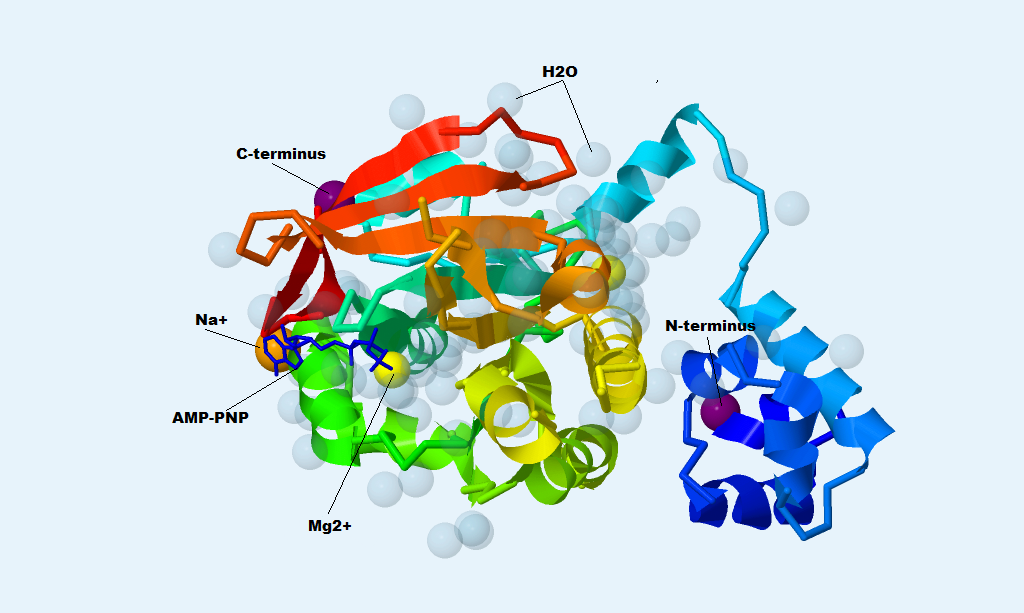
Рис. 1. Белок RadA со всеми лигандами. Подписаны и обозначены фиолетовым N- и С-концы белка. Цветами радуги от синего к красному показано, как следует цепь RadA от N-конца к C-концу

Рис. 2. Структурная формула AMP-PNP
Область контакта c AMP-PNP и ионом магния (АТФ-связывающего сайта) в структуре белка RadA
На рис. 3 подписаны аминокислоты, участвующие в создании АТФ-связывающего сайта. Это все остатки, которые образуют водородные или ионные связи с молекулой AMP-PNP либо с близким с ней ионом магния.
Скрипт Jmol для создания изображения: загрузить.
Внимание: для работы скрипта необходимо наличие скриптов из предыдущих практикумов Vasyutkina.spt (загрузить) и atom-types.spt (загрузить)!
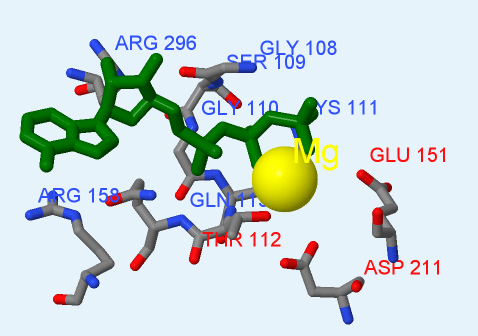
Рис. 3. Область АТФ-связывающего сайта белка RadA. Синим подписаны остатки, образующие связи с молекулой AMP-PNP, красным - с ионом магния. Зеленым цветом покрашена молекула AMP-PNP. Получено с помощью Jmol
Область контакта с молекулой AMP-PNP поддерживается ионными и водородными связями. Рисунок 4 иллюстрирует расстояния между аминокислотами в области связывания и лигандом. На рис. 5 показаны расстояния от аминокислот в сайте связывания до атома магния. Также атом магния удерживается ионными связями с молекулой AMP-PNP.
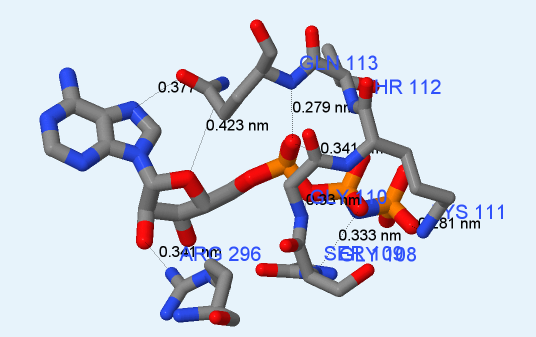
Рис. 4. Расстояния между атомами AMP-PNP и аминокислотами АТФ-связывающего сайта белка RadA. Раскраска молекул по типу атомов (cpk). Получено с помощью Jmol
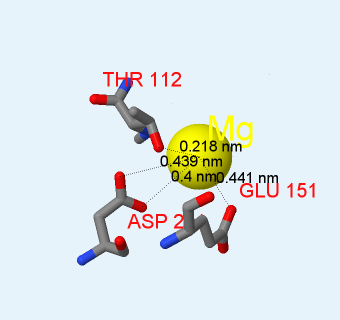
Рис. 5. Расстояния между атомом магния и связанными с ним аминокислотами АТФ-связывающего сайта белка RadA. Раскраска молекул по типу атомов (cpk). Получено с помощью Jmol
Проект генно-инженерного эксперимента с белком RadA из структуры 3NTU
Роль атома натрия в белке RadA не определена. Он может быть кофактором, то есть участвовать вместе с белком в катализе реакции, а может быть побочным атомом, добавленным для упрощения кристаллизации белка при рентгеноструктурном анализе. Для выяснения роли иона натрия можно провести в белке точечную мутацию. С ионом натрия связан 164-й остаток глутаминовой кислоты. Смотрим таблицу кодонов РНК. Кодоны глутаминовой кислоты (GAA, GAG) отличаются от кодонов аланина (GCA, GCG) лишь второй буквой. Аланин - неполярная аминокислота, ион натрия связывать не будет. Таким образом, для определения роли иона натрия в белке можно заменить всего 1 нуклеотид ДНК и проверить полученный мутантный белок на ферментативную активность.
Также можно проверить вклад аминокислоты, находящейся относительно далеко (1,104 нм) от области связывания - аминокислотного остатка аргинина 314. Я считаю, что если заменить его на любую неполярную аминокислоту, то белок сохранит способность связывать лиганд AMP-PNP. Возможно, это будет происходить чуть медленнее за счет потери части положительного заряда на поверхности белка в области сайта.