| Главная | Семестры | Обо мне | Ссылки |
|---|
Выравнивания последовательностей и их отображение с помощью JalView
JalView - редактор выравниваний, написан на Java. Официальный сайт программы: ссылка.
Для работы было использовано множественное выравнивание: загрузить исходное выравнивание, проект JalView. Все выравнивание покрашено раскраской ClustalX с параметром консервативности (Above identity threshold ) >70%.
Для начала в выравнивании были найдены 2 вертикальных блока. Блок - это фрагмент выравнивания, позволяющий ожидать гомологию между отдельными аминокислотными остатками каждой последовательности. В вертикальном блоке есть абсолютно консервативные позиции.
В данном множественном выравнивании встречаются всего 2 вертикальных блока, которые приведены на рис. 1 и 2.
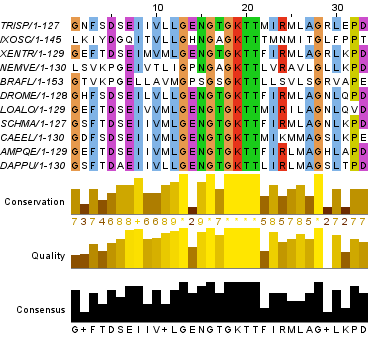
Рис. 1. Первый вертикальный блок выравнивания (позиции №№ 6-38). Получено с помощью JalView.
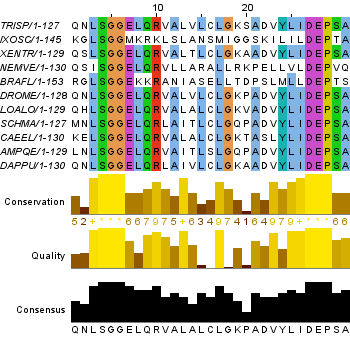
Рис. 2. Второй вертикальный блок выравнивания (позиции №№ 131-161). Получено с помощью JalView.
Далее я выделила участок, на котором выделяются 3 последовательности, которые имеют не гомологичные остальным участки - короткие вставки (рис. 3). Также можно заметить, что внутри блоков эти последовательности практически не имеют совпадений аминокислотных участков с остальными. Таким образом, судя по выделенному фрагменту, нельзя утверждать о гомологии последовательностей IXOSC/1-145, NEMVE/1-130, BRAFL/1-153 остальным.

Рис. 3. Участок выравнивания (позиции №№ 52-110). Получено с помощью JalView.
Рассмотрим более подробно блок на рис. 1. Можно ли считать позиции данного блока гомологичными между собой, т.е. имеющими общее происхождение?
Таблица 1. Консервативность позиций блока 6-38 множественного выравнивания
Количество № позиций Процент позиций Всего позиций 33 1-161 100% Абсолютно консервативных позиций 7 13, 16, 18-21, 28 21% Абсолютно функционально консервативных позиций 8 1, 6, 8, 11, 12, 23, 25, 26 24% Консервативных на 70% позиций 12 3, 5, 7, 10, 14, 15, 17, 24, 27, 30, 32, 33 36% Функционально консервативных на 70% позиций 2 4, 9 6%
Итого 87% позиций в блоке имеют большое сходство между собой и могут быть гомологичными.
Рассмотрим теперь подробнее оба вертикальных блока множественного выравнивания. Можно считать, что эти блоки расположены близко, т.к. между ними менее 100 позиций. Возьмем участок выравнивания, содержащий конец 1-го блока и начало 2-го (24-137 позиции), он показан на рис. 4.
Всего позиций: 114
Позиций с гепами: 35
Процент позиций с гепами: 31%
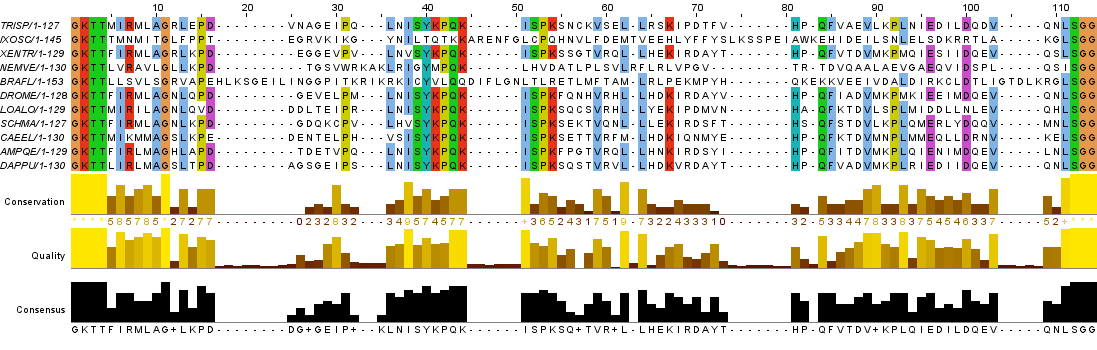
Рис. 4. Участок выравнивания между вертикальными блоками (позиции №№ 24-137). Получено с помощью JalView.
Добавим к выравниванию еще одну последовательность (File -> Add Sequence) и выровняем ее с блоком на рис. 2.
Чтобы вручную выровнять последовательность, я использовала поиск (Select -> Find), а также перемещение стрелками блока относительно последовательности.
Полученное выравнивание см. на рис. 5. Можно сделать вывод, что последовательность гомологична остальным.
Консенсусная последовательность данного выравнивания:
QNLSGGELQRVALALCLGKPADVYLIDEPSA
С помощью сервиса http://weblogo.berkeley.edu/logo.cgi я создала лого данного выравнивания, то есть графическое отображение консенсусной последовательности. Оно показано на рис. 6.

Рис. 5. Выравнивание вертикального блока 2 и последовательности. Получено с помощью JalView.
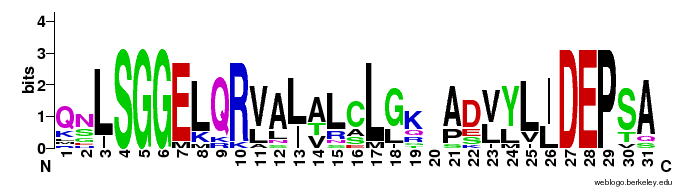
Рис. 6. Лого консенсусной последовательности.
Выравнивание заведомо негомологичных последовательностей
Я выбрала 8 белков, с которыми работают студенты 1 курса ФББ 2013. С помощью сервиса ID Mapping на сайте базы данных UniProt я перевела идентификаторы белков NCBI в UniProt's AC. Далее импортировала их в JalView (File -> Fetch sequences) и построила выравнивание (Web services -> Alignment -> Muscle with Defaults). Проект в JalView: загрузить .Сразу бросается в глаза огромное количество гепов в последовательностях, что отнюдь не свидетельствует об их гомологии. На рис. 7 и 8 показаны 2 найденных мной блока выравниваний, раскрашенные с разными параметрами консервативности для наглядности.
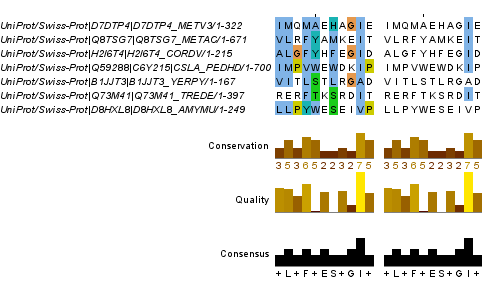
Рис. 7. Участок выравнивания (позиции №№ 411-421), раскрашенный с параметрами консервативности (Above identity threshold ) слева >0%, справа >50%. Получено с помощью JalView.
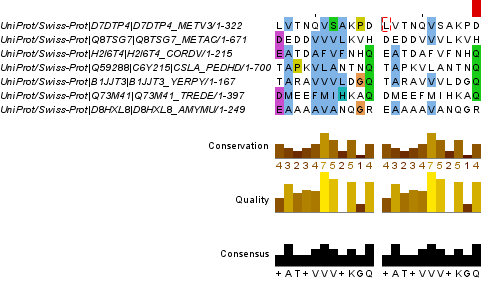
Рис. 8. Участок выравнивания (позиции №№ 623-633), раскрашенный с параметрами консервативности (Above identity threshold ) слева >0%, справа >70%. Получено с помощью JalView.
Аналогично первому выравниванию, я выделила участок, на котором выделяются 3 последовательности, Q8TSG7, Q59288, Q73M41. Они имеют короткие "вставки", правда, всего с одной совпадающей позицией. Участок - на рис. 9, покрашен с разными параметрами консервативности. Для себя я сделала вывод, что раскраска ClustalX по умолчанию показывает довольно оптимистичную картинку: даже в таком выравнивании видны отдельные блоки, внутри которых достаточно много позиций одного цвета. Поэтому нужно всегда задавать порог консервативности, отличный от нуля.
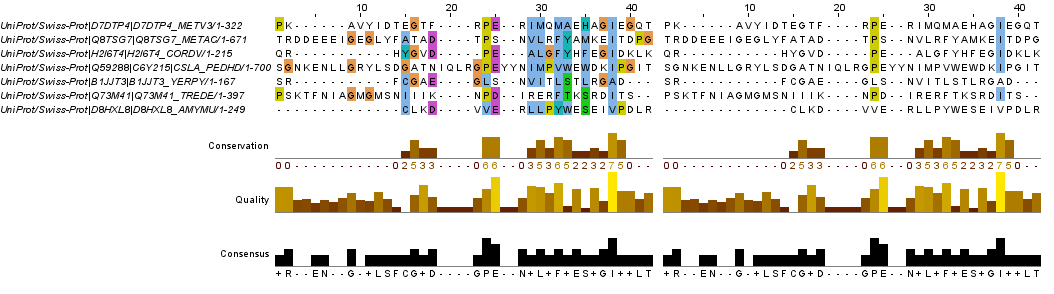
Рис. 9. Участок выравнивания (позиции №№ 383-424), раскрашенный с параметрами консервативности (Above identity threshold ) слева >0%, справа >70%. Получено с помощью JalView.
Для сравнения я посчитала процентное содержание позиций с гепами в первом и втором выравнивании.
1-е выравнивание: 38 с гепами, всего 161 - 24%
2-е выравнивание: 94 без гепов, 710 с гепами, всего 804 - 88%
Такое сравнение выравниваний друг с другом очень условно, т.к. в них разное количество последовательностей, длина также различается.
Но из этого можно сделать вывод, что сходство последовательностей выражается в наличии консервативных участков, то есть участков, где много совпадений аминокислотных остатков, а также небольшим содержанием гепов, которые соответствуют инделям.
Из степени сходства последовательностей можно сделать вывод о наличии гомологии последовательностей, а значит, и об общем происхождении организмов, откуда были получены исследуемые белки.