| Главная | Семестры | Обо мне | Ссылки |
|---|
Предсказание парных выравниваний
В данной работе я сравнивала парные выравнивания двух последовательностей белков, полученные разными способами:
- парное выравнивание, полученное из множественного;
- глобальное парное выравнивание двух последовательностей с помощью программы needle пакета программ EMBOSS.
Проект JalView с полученными выравниваниями: загрузить.
Для начала было получено парное выравнивание из множественного. Исходное множественное выравнивание: загрузить.
Далее я сохранила последовательности белков в отдельные файлы в формате fasta (под названиями pr10_1.fasta и pr10_2.fasta) и получила глобальное выравнивание программой needle.
Она работает по алгоритму Нидлмана — Вунша.
На рис.1 показано полученное выравнивание.
Команда bash выглядит так:
needle pr10_1.fasta pr10_2.fasta pr10_needle
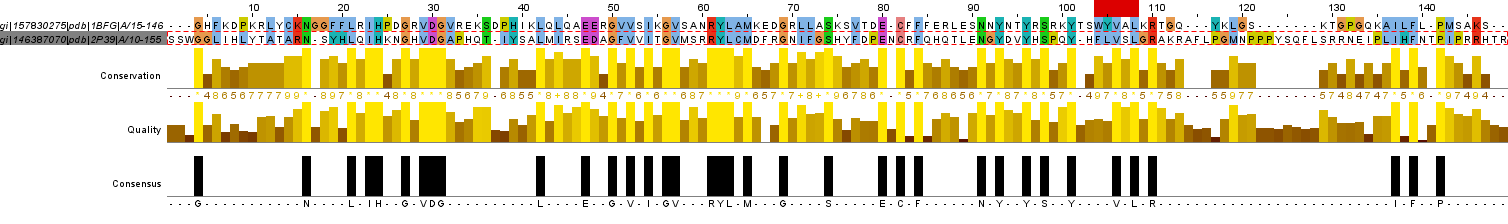
Рис. 1. Парное выравнивание, полученное программой needle
На выходе получился файл с выравниванием в формате srspair (загрузить). Чтобы не перепутать, я переименовала его в pr10_needle.srspair.
В нем помимо выравнивания содержится информация о сходстве последовательностей: количество и процент идентичных и сходных аминокислотных остатков, число и процент гэпов, вес выравнивания (Score).
Для получения выходного файла в формате fasta необходимо добавить опцию -aformat3 fasta. Команда в этом случае выглядит так:
needle pr10_1.fasta pr10_2.fasta pr10_needle -aformat3 fasta
С помощью программы water пакета EMBOSS было получено локальное выравнивание тех же последовательностей.
Механизм работы этой программы основан на алгоритме Смита-Ватермана.
Если в глобальном выравнивании идет поиск сходства целых последовательностей, то в локальном ищутся 2 наиболее похожих фрагмента в каждой из последовательностей и выравниваются только они.
При этом локальное выравнивание содержит только эти 2 фрагмента, а не последовательности целиком.
Команда bash для получения локального выравнивания:
water pr10_1.fasta pr10_2.fasta pr10_water
Как и в случае программы needle, я получила файл в формате srspair (загрузить).
Команда для получения выравнивания в формате fasta:
water pr10_1.fasta pr10_2.fasta pr10_water -aformat3 fasta
Для сравнения я построила глобальное и локальное выравнивания заведомо негомологичных белков.
Были использованы последовательности белка репарации и рекомбинации RadA археи Methanococcus voltae (RadA_METVO.fasta) и алкогольдегидрогеназы археи Pyrobaculum aerophilum (белок Буяновой Софьи, Q8ZUP0_PYRAE.fasta). Глобальное выравнивание представлено на рис.2.
Результат сравнения выравниваний приведен в таблице 1. Первые две последовательности - гомологичных белков, третья и четвертая - заведомо не гомологичных.
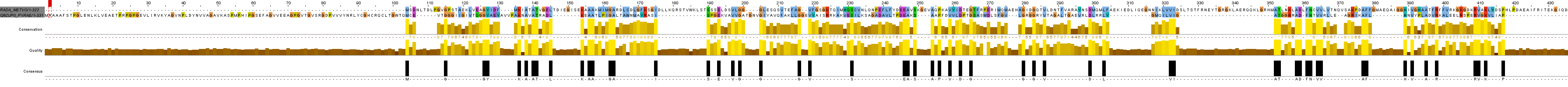
Рис. 2. Парное выравнивание негомологичных последовательностей, полученное программой needle
Таблица 1. Сравнение глобальных и локальных выравниваний гомологичных и негомологичных белков.
| Последовательности | pr10_1.fasta; pr10_2.fasta | RadA_METVO.fasta; Q8ZUP0_PYRAE.fasta | ||
|---|---|---|---|---|
| Программа | needle | water | needle | water |
| Длина выравнивания | 166 | 96 | 435 | 145 |
| Число совпадений | 39 | 33 | 56 | 29 |
| Процент совпадений | 23,50% | 34,40% | 12,90% | 20,00% |
| Число сходных остатков | 56 | 47 | 104 | 56 |
| Процент сходных остатков | 33,70% | 49% | 23,90% | 38,60% |
| Число гэпов | 54 | 11 | 217 | 36 |
| Процент гэпов | 32,50% | 11,50% | 49,90% | 24,80% |
| Число открытий гэпов | 6 | 2 | 15 | 6 |
Теперь сравним выравнивания гомологичных белков, полученные из множественного выравнивания (выравнивание 1) и с помощью программы needle (выравнивание 2). На рис. 3 показано расположение двух парных выравниваний относительно друг друга.
Я отметила "+" все те позиции, где выравнивание, с моей точки зрения, достоверное. Буквой S отмечены позиции, для которых есть совмещение C-α атомов в пространственной структуре.
Ошибка 1го рода - случай, когда C-α атомы пары остатков в колонке выравнивания, отмеченной "+", в структуре, очевидно, не совмещаются.
Ошибка 2го рода: C-α атомы двух остатков, очевидно, хорошо совмещаются, но соответствующие буквы либо не находятся в одной колонке, либо в одной колонке, но колонка не отмечена "+".
Выравнивание 1: 3 ошибки 1 рода, 10 ошибок 2 рода
Выравнивание 2: 3 ошибки 1 рода, 2 ошибки 2 рода
На рис.4 показан один из участков различия выравниваний, его координаты в общем выравнивании 39-60. В обоих выравниваниях процент совпадающих аминокислот небольшой, но в первом - одно открытие гэпов, тогда как во втором их 2. В этом случае верхнее выравнивание лучше.
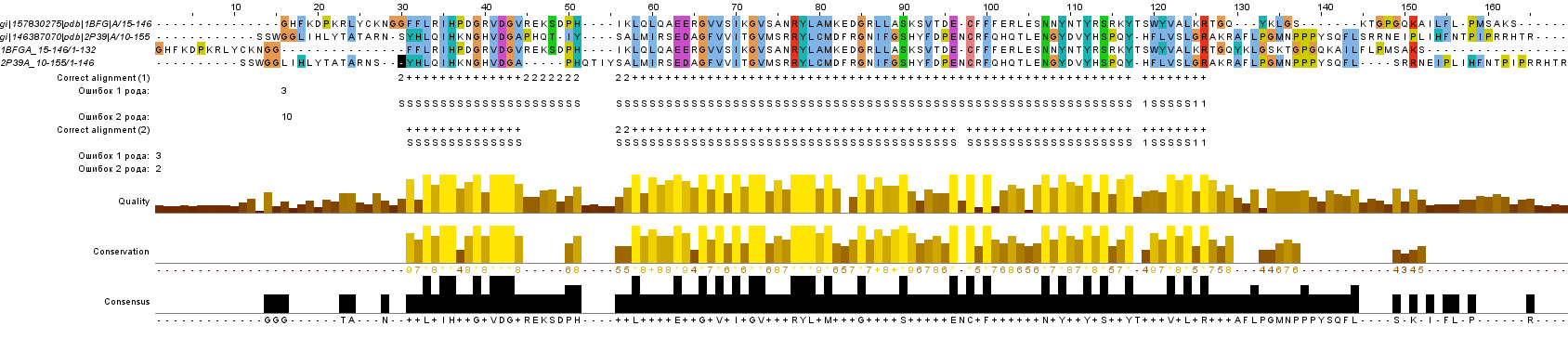
Рис. 3. Сравнение парных выравниваний
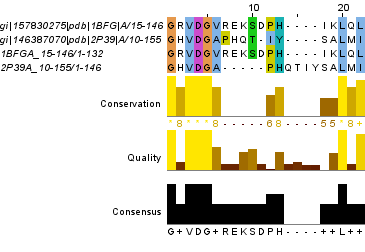
Рис. 4. Участок различия парных выравниваний, координаты 39-60
На самом деле, в выравнивании 1 гораздо больше позиций, где атомы отмечены и "+", и "S". К тому же, по моему мнению, в нем лучше выровнены края последовательностей. Мне кажется, выравнивание 1 можно считать "правильным".