| Главная | Семестры | Обо мне | Ссылки |
|---|
Построение множественного выравнивания. Работа в Pfam
Проект JalView со всеми выравниваниями: загрузить.
Для начала я провела 4 итерации PSI-BLAST для поиска последовательностей для выравнивания.
Входные данные: АС моего белка O73948, база данных Swiss-Prot, таксон для поиска Archaea.
Я решила не брать последовательности организмов из родов Methanococcus и Methanocaldococcus, так как их белки имеют очень большой процент сходства с моим.
На рис. 1 показаны лучшие находки PSI-BLAST и выбранные последовательности.
Файл в формате fasta с выбранными последовательностями: загрузить.
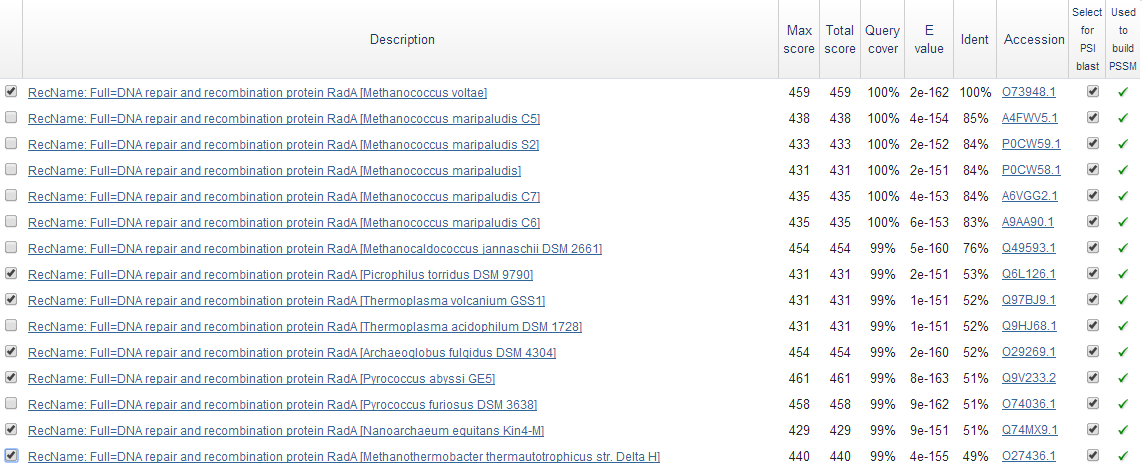
Рис. 1. Результат PSI-BLAST и выбранные последовательности
Получим выравнивание этих последовательностей программой muscle на сервере kodomo. Результат представлен на рис. 2. Команда bash для получения выравнивания:
muscle -in pr13_input.fasta -out pr13_muscle.fasta

Рис. 2. Выравнивание последовательностей с помощью muscle. Здесь и далее: раскраска BLOSUM62, порог консервативности 70%
Второе выравнивание сделано с помощью программы mafft на сервере kodomo. Его можно увидеть на рис. 3. Команда выглядит так:
mafft pr13_input.fasta > pr13_mafft.fasta

Рис. 3. Выравнивание последовательностей с помощью mafft
Программа muscle также строит выравнивание выравниваний для их сравнения. Для этого я ввела такую команду:
muscle -profile -in1 pr13_muscle.fasta -in2 pr13_mafft.fasta -out pr13_both.fasta
Результат работы muscle на рис. 4.
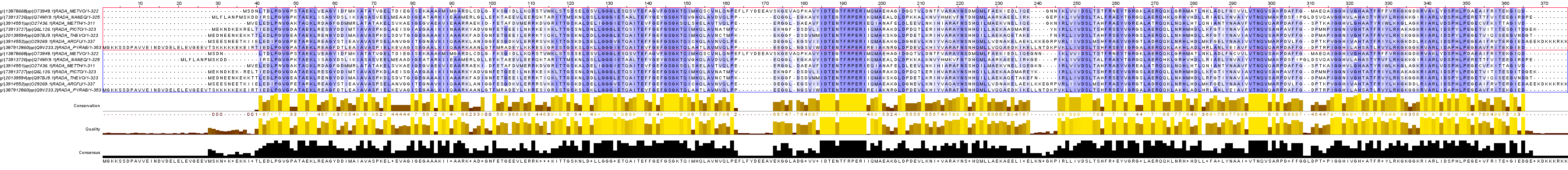
Рис. 4. Сравнение выравниваний, полученных с помощью muscle (в красной рамке) и mafft (в синей рамке)
Как видно из сравнения выравниваний, они практически идентичны. Обнаружилось только 2 небольших участка различия: начало (рис. 5) и 239-245 (рис. 6). Но сложно сказать, что на участках различия одно из выравниваний заметно лучше другого. Они оба дают понять, что в этих местах нет гомологии между последовательностями. Значит, оба выравнивания за вычетом позиций с координатами 1-41 и 239-245 можно назвать правильными: гомологичные участки находятся в одной колонке, а вне участков выравнивания гомологичных остатков нет.
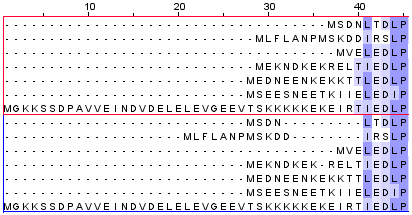
Рис. 5. Участок различия выравниваний №1 (координаты 1-41)

Рис. 6. Участок различия выравниваний №2 (координаты 239-245)
Поиск доменов в Pfam
Pfam - база данных белковых доменов.
Для поиска доменов в моей последовательности я ввела АС белка репарации и рекомбинации RadA O73948.
Результат поиска представлен на рис. 7. Всего обнаружилось 2 домена: HHH_5 и Rad51.
seed-выравнивание для семейства Rad51: загрузить.
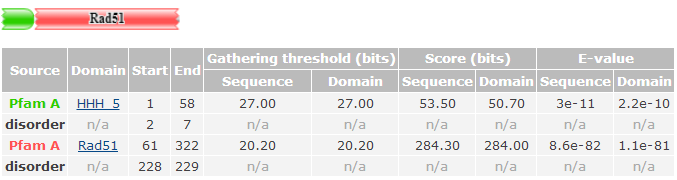
Рис. 7. Результат поиска доменов в структуре белка RadA на сайте Pfam
HHH_5 (Helix-Hairpin-Helix) - это домен, который отвечает за связывание белка с ДНК.
Rad51 - домен белка репарации и рекомбинации с тем же названием Rad51 у эукариот, он занимает 61-322 остаток в белке RadA (81%).
На схеме Sunburst (рис. 8) можно увидеть, что этот домен встречается у огромного количества групп среди эукариот, а также у архей. Исходя из этого, можно предположить, что эукариоты "позаимствовали" у архей белок репарации и рекомбинации.
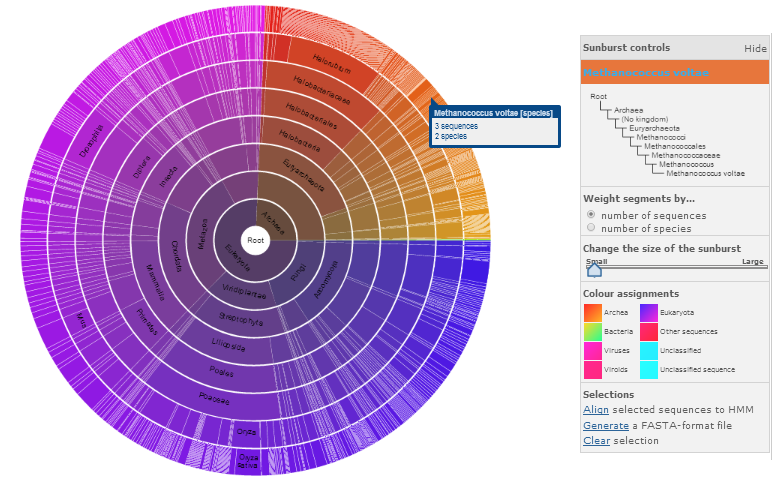
Рис. 8. Схема Sunburst для домена Rad51, отражающая многообразие таксонов с этим доменом. Получено с помощью Pfam